摘要
最近的工作强调了微生物组在人类健康中的重要性,并在很大程度上将表型的差异归因于个体之间存在的物种差异1,2,3.,4,5.然而,移动基因可以在同一物种的不同菌株上赋予截然不同的表型。人们对人类微生物组中移动基因的功能和分布知之甚少,特别是基因库是全球同质的还是受人类人口结构的限制。在这里,我们使用单细胞基因组学和宏基因组学相结合的方法,通过比较在81个北美大都市和172个斐济农业岛民微生物群中发现的移动基因来研究这个问题。我们发现斐济和北美微生物群之间的移动基因含量有很大差异,功能差异反映了已知的饮食差异,如斐济个体中发现的植物性淀粉降解基因过多。值得注意的是,我们还观察到邻近斐济村庄的移动基因库之间的差异,尽管村庄之间的微生物组组成是相似的。最后,我们观察到高比率的重组导致个体特定的移动元素,这表明一些基因的丰度可能反映了环境选择而不是扩散限制。总之,这些数据支持了这样一个假设:人类活动和行为提供了形成移动基因库的选择压力,而移动基因的获取对于殖民特定的人类群体很重要。
这是订阅内容的预览,通过你的机构获取
相关的文章
引用本文的开放存取文章。
来自半孤立的人类群体肠道微生物组的质粒网络揭示了独特和共享的代谢和毒力特征
科学报告开放获取2022年7月15日
人类肠道中的普雷沃氏菌属主要由copri普雷沃氏菌属、stercorea普雷沃氏菌属及相关谱系组成
科学报告开放获取2022年5月31日
抗生素耐药基因的全球健康风险评估
自然通讯开放获取2022年3月23日
访问选项
订阅期刊
获得1年的完整期刊访问权限
199.00美元
每期仅需$3.90
所有价格均为净价格。
增值税将在稍后的结帐中添加。
税金计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问权限。
32.00美元
所有价格均为净价格。
加入代码
参考文献
Turnbaugh, P. J.等。一种与肥胖相关的肠道微生物群,其能量收集能力增强。自然444, 1027-1031 (2006)
Giongo等人。定义1型糖尿病的自身免疫微生物群。ISME J。5, 82-91 (2011)
秦杰等。2型糖尿病患者肠道菌群的宏基因组相关性研究。自然490, 55-60 (2012)
谢尔,J. U.等。肠道扩张普氏菌copri与关节炎易感性增强相关。eLife2e01202 (2013)
Kang, D. W.等。发病率降低普氏菌以及自闭症儿童肠道菌群中的其他发酵剂。《公共科学图书馆•综合》8, e68322 (2013)
斯迈利,C. S.等。生态学驱动着连接人类微生物群的全球基因交换网络。自然480, 241-244 (2011)
Kumarasamy, k.k.等人。一种新的抗生素耐药机制在印度、巴基斯坦和英国的出现:一项分子、生物学和流行病学研究。柳叶刀感染。说。10, 597-602 (2010)
De Filippo, C.等。一项欧洲和农村儿童的比较研究揭示了饮食对肠道菌群形成的影响非洲。美国国家科学院学报。美国107, 14691-14696 (2010)
Yatsunenko, T.等。跨越年龄和地域的人类肠道微生物组。自然486, 222-227 (2012)
王晓燕,王晓燕,王晓燕。哺乳动物相关乳酸菌的分解代谢灵活性。活细胞。细胞的事实。12, 48 (2013)
夏皮罗,B. J.等。细菌生态分化早期事件的群体基因组学研究。科学336, 48-51 (2012)
黑埃曼,黄永发。et al。海洋细菌中碳水化合物活性酶向日本肠道菌群的转移。自然464, 908-912 (2010)
萨默斯,a.o.等人。从牙科“银”填充物中释放出的汞会刺激灵长类动物口腔和肠道菌群中汞和抗生素耐药性细菌的增加。Antimicrob。代理Chemother。37, 825-834 (1993)
福斯伯格,K. J.等。土壤细菌和人类病原体共有的抗生素耐药体。科学337, 1107-1111 (2012)
Forslund, K.等。国家特定的抗生素使用做法会影响人类肠道耐药体。基因组Res。23, 1163-1169 (2013)
人类微生物组项目联盟。健康人体微生物群的结构、功能和多样性。自然486, 207-214 (2012)
Coyne, m.j., Zitomersky, n.l., McGuire, a.m., Earl, a.m. & Comstock, l.e.人类肠道内拟杆菌类物种之间广泛DNA转移的证据。MBio5, e01305-e01314 (2014)
Jones b.v, Sun F. & Marchesi, J. R.人类肠道微生物组中质粒编码功能的比较宏基因组学分析。BMC基因组学11, 46 (2010)
Brown Kav, A.等。牛瘤胃质粒组的研究。美国国家科学院学报。美国109, 5452-5457 (2012)
Sentchilo, V.等人。全社区质粒基因的动员和选择。ISME J。7, 1173-1186 (2013)
布莱巴特等人。人类粪便中未培养病毒群落的宏基因组学分析。j . Bacteriol。185, 6220-6223 (2003)
雷耶斯等人。同卵双胞胎及其母亲粪便菌群中的病毒。自然466, 334-338 (2010)
人类微生物组Jumpstart参考菌株联盟人类微生物组参考基因组的目录。科学328, 994-999 (2010)
康塔雷尔,B. L.隆巴德,V.和亨里萨特,B.健康人类微生物群对复杂碳水化合物的利用。《公共科学图书馆•综合》7, e28742 (2012)
Clemente, J. C.等。未接触的美洲印第安人的微生物群。科学。睡觉。1, e1500183 (2015)
Faith, J. J.等。人体肠道菌群的长期稳定。科学341, 1237439 (2013)
大卫,洛杉矶等。宿主的生活方式在日常时间尺度上影响人类微生物群。基因组医学杂志。15, r89 (2014)
Dean, F. B, Nelson, J. R, Giesler, T. L. & Lasken, R. S.利用Phi 29 DNA聚合酶和多重引物滚动圈扩增快速扩增质粒和噬菌体DNA。基因组Res。11, 1095-1099 (2001)
科尔,J. R.等。核糖体数据库项目:用于高通量rRNA分析的数据和工具。核酸储备。42, d633-d642 (2014)
班克维奇等人。SPAdes:一种新的基因组组装算法及其在单细胞测序中的应用。j .第一版。医学杂志。19, 455-477 (2012)
吴明,吴晓燕。细菌和古菌序列的系统基因组学分析。生物信息学28, 1033-1034 (2012)
Parks, D. H., Imelfort, M., Skennerton, c.t, Hugenholtz, P. & Tyson, G. W. CheckM:评估从分离物、单细胞和宏基因组中恢复的微生物基因组的质量。基因组Res。251043-1055 (2015)
Lagesen, K.等。RNAmmer:核糖体RNA基因的一致和快速注释。核酸储备。35, 3100-3108 (2007)
Nawrocki, E. P. & Eddy, S. R. Infernal 1.1: RNA同源性搜索速度快100倍。生物信息学29, 2933-2935 (2013)
Price, M. N, Dehal, P. S. & Arkin, A. P. FastTree 2 -大型对齐的近似最大似然树。《公共科学图书馆•综合》5, e9490 (2010)
凯悦等人。浪子:原核基因识别与翻译起始位点的鉴定。BMC生物信息学11, 119 (2010)
搜索和聚类的数量级比BLAST快。生物信息学26, 2460-2461 (2010)
Kanehisa等人。数据、信息、知识和原理:回到KEGG的新陈代谢。核酸储备。42, d199-d205 (2014)
dbCAN:一个自动标注碳水化合物活性酶的网络资源。核酸储备。40, w445-w451 (2012)
Gibson, M. K, Forsberg, K. J.和Dantas, G.对抗生素耐药决定因素的改进注释揭示了微生物耐药体的生态学聚集。ISME J。9, 207-216 (2015)
基于Burrows-Wheeler变换的快速、准确的短读对齐。生物信息学25, 1754-1760 (2009)
核糖体靶向抗生素和细菌耐药机制。微生物学。12, 35-48 (2014)
Gilbart, J, Perry, C. R. & Slocombe, B.金黄色葡萄球菌对莫比罗星的高水平耐药:两种不同的异亮基trna合成酶的证据。Antimicrob。代理Chemother。37, 32-38 (1993)
陶,施梅尔,和希尔,J.氨基酰tRNA合成酶作为新的抗感染靶点。美国实验生物学学会联合会J。12, 1599-1609 (1998)
垒,J. G., O 'Neill, A. J., Mody, L., Chopra, I. & Bradley, S. F.高水平莫比罗星耐药的体内转移葡萄球菌epidermidis对耐甲氧西林金黄色葡萄球菌与莫匹罗星预防失败有关。j . Antimicrob。Chemother。56, 1166-1168 (2005)
Widmann, J., Harris, J. K, Lozupone, C., Wolfson, A. & Knight, R.仅使用76个核苷酸的稳定的基于trna的系统发育。核糖核酸16, 1469-1477 (2010)
Laslett, D. & Canback, B. ARAGORN,一个检测核苷酸序列中tRNA基因和tmRNA基因的程序。核酸储备。32, 11-16 (2004)
刘斌,Gibbons, T., Ghodsi, M., Treangen, T. & Pop, M.从宏基因组鸟枪序列中准确快速地估计分类剖面。BMC基因组学12(增补2),S4 (2011)
Quast, C.等。SILVA核糖体RNA基因数据库项目:改进的数据处理和基于网络的工具。核酸储备。41, d590-d596 (2013)
罗伯逊(编著)《斐济的食品和营养:与营养有关的疾病及其预防》(南太平洋大学,1991年)
E. Rush, Hedges, R., Alsbersberg, B., Qionibaravi, D. & Laulu, M.维拉塔一个农村的主食摄入量,斐济。Pac.健康对话框8, 44-46 (2001)
粮农组织。太平洋粮食安全工具包:建立应对气候变化、根茎作物和渔业生产的韧性(联合国粮农组织,2010)
戈艾滋,J。手工捕鱼对斐济一个岛屿礁鱼群的影响和深度避难的证据(论文,西澳大利亚大学,2009)
朱庇特(S. Jupiter),萨拉德劳(Saladrau), W. &瓦夫(Vave), R。通过斐济刘省的针对性调查对海参渔业进行评估(野生动物保护协会/南太平洋大学/斐济渔业部/哈立德·本·苏丹生命海洋基金会,2013)
库马尔(Kumar M.)、阿尔伯斯伯格(Aalbersberg B.)和莫尔(molle L.)斐济海产品中的汞含量及其对健康的潜在影响。(世界卫生组织,西太平洋区域,2004年)
莫提,A. &莫尔斯,Z.斐济成人牙科抗生素处方。Int。凹痕。J。57, 65-70 (2007)
国家药物和治疗小组委员会。抗生素的指导方针3.理查德·道金斯(卫生部,斐济政府,2011年)
汤普森,C. N.等。斐济的伤寒:一种可逆转的瘟疫?太。地中海,Int。健康19, 1284-1292 (2014)
Sommer, m.o.a, Dantas, G. & Church, g.m.人类微生物区系中抗生素耐药性库的功能表征。科学325, 1128-1131 (2009)
多重抗药性志贺杆菌的死亡:来自斐济的一个案例研究。Pac.健康对话框13, 111-114 (2006)
确认
我们感谢我们在斐济群岛的实地合作者:斐济野生动物保护协会、国际湿地大洋洲、K. Jenkins、S. Korovou、N.利蒂达木和K. Kishore。我们感谢T. Poon提供的样本、测序和数据协调,感谢A. Materna (QIAGEN)提供的技术支持。这项工作得到了国家人类基因组研究所(U54HG003067)、布罗德研究所、麻省理工学院环境卫生科学中心、麻省理工学院微生物信息学和治疗学中心以及斐济卫生部的资助。哥伦比亚大学地球研究所奖学金(i.l.b)提供了其他支助;布罗德研究所劳伦斯·萨默斯奖学金(l.x);科学界面巴勒斯威康基金职业奖(P.C.B.);由NIDCR和ENIGMA资助的R01 DE020891,以及由美国能源部、科学办公室、生物和环境研究办公室(S.Y.和A.K.S.)支持的劳伦斯伯克利国家实验室科学重点领域项目。桑迪亚是洛克希德·马丁公司旗下的桑迪亚公司为美国能源部运营的多项目实验室,合同为DE-AC04-94AL85000。
作者信息
作者和隶属关系
贡献
i.l.b和e.j.a设计了这项研究。I.L.B, S.D.J, a.p.j和W.N.负责监督和执行FijiCOMP数据和样本的现场收集。I.L.B, L.X, S.Y和M.T.完成了所有的实验工作。d.g., b.w.b., j.r.w., P.C.B, r.j.x和A.K.S.负责DNA测序的生产。I.L.B.和K.H.处理了霰弹枪的数据并进行了校准。I.L.B, k.h.和D.G.提供了新的分析工具。I.L.B, K.H.和C.S.S.进行了计算分析。i.l.b和e.j.a写了手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
额外的信息
审核人信息自然感谢P. Bork, K. Forslund, P. Hugenholtz和C. Rinke对本研究的同行评议做出的贡献。
扩展的数据图和表
扩展数据图1研究中使用的组件的系统发育跨越了细菌生命树。
利用本分析中使用的所有参考基因组和单细胞组合的16S rRNA全基因或16S rRNA基因V68区的多重序列比对构建的系统发育树。使用RDP构建16S对准线。然后使用FastTree组装树。树中所有深分支的支持度都很低,因此仅为了说明目的,原始分支作为外群。外部的颜色条显示了古类群和细菌门的分类联系。内部的颜色条显示该操作分类单元的来源:HMP参考单元(n= 387蓝色)和FijiCOMP单单元程序集(n= 110,红色)。70个FijiCOMP单细胞组合的16S rRNA基因序列无法获得,因此未包含在该树中。
图2鉴定水平转移基因和评估它们在宏基因组样本中的分布的方法。
水平转移区域首先通过HMP参考基因组和FijiCOMP单细胞组合之间的成对blast进行识别。开放阅读框在水平传输区域内进行注释。移除移动基因集中的遗传冗余,以确保使用UCLUST和BLAST组合进行准确的丰度估计。然后将宏基因组读数与独特的移动基因数据集进行比对。对对齐进行过滤,只保留那些在超过50%的读取长度中与99%标识对齐的读取。在宏基因组样本中,比对深度至少为4 ×比对深度大于基因长度80%的基因进行基因丰度测定。
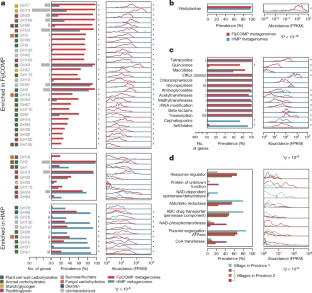
图3移动基因家族的丰度很大程度上取决于队列。
一个,热图显示了每个宏基因组样本(81个HMP样本和172个FijiCOMP样本)中由功能基因家族(COG分配,KEGG, TIGRFAM或PFAM家族)聚集的移动基因的丰度(FPKM)。利用完全连锁对个体间功能基因家族谱之间的欧氏距离和个体间移动基因组成之间的距离进行了层次聚类。数值以对数刻度标出。b,热图显示了在每个宏基因组样本中被认为有较高置信度的移动基因家族的丰度(FPKM)。这些包括来自移动基因的移动基因家族,它们被注释为水平转移机制或有额外的支持它们的系统发育位置。基因家族和个体的位置维持在一个为了比较。c热图显示了在每个宏基因组样本中被观察到在HMP参考基因组之间转移的移动基因的丰度(FPKM)。基因家族和个体的位置维持在一个为了比较。
图4高置信度移动基因内GH13基因和糖苷水解酶家族分布显示群体特异性富集。
一个, FijiCOMP中标注为GH13家族成员的移动基因的患病率和丰度(用FPKM衡量)(n= 172,红色)和HMP (n= 81,蓝色)宏基因组粪便样本。b在FijiCOMP(红色)和HMP(蓝色)宏基因组粪便样本中存在的高置信度移动基因亚群中的所有糖苷水解酶家族的患病率和丰度。这里只包括从移动基因中被注释为水平转移机制或对其系统发育位置有额外支持的独特基因家族。丰度通过FPKM测量,根据糖苷水解酶家族聚合,并作为样品密度的函数绘制。对于每个糖苷水解酶家族,观察到的独特水平转移基因的数量被记录下来,以及它们底物的来源。
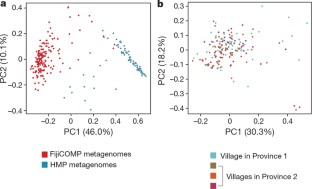
图5 HMP和FijiCOMP研究参与者的肠道微生物群组成。
一个,根据门绘制的个体宏基因组样本的细菌相对丰度(n=81,蓝色)和FijiCOMP (n=172,红色)队列。根据队列和优势门的丰度对样本进行分类。b,在HMP(蓝色)和FijiCOMP(红色)队列中个体的宏基因组样本中,拟杆菌科(bacterioales)目内的科的相对丰度。根据队列和拟杆菌门顶端成员的丰度对样本进行排序。
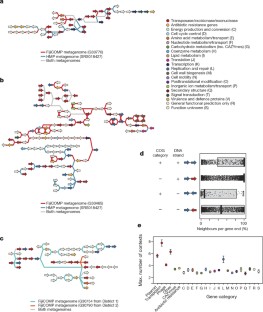
图6移动基因在两个队列中广泛的细菌宿主背景中被观察到。
一个,b热图显示了与一个tRNA基因和两个特定水平转移基因对齐的人均读对数。根据与特定tRNA基因相关的物种,热图中的颜色反映了read丰度。颜色条显示读出的数据来自哪个气象组群:FijiCOMP(红色)和HMP(蓝色)。
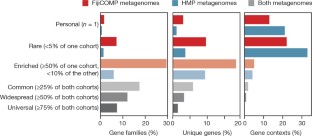
图7基因和环境的相对丰度在人群中对精确的定义并不敏感。
基因家族的百分比,由COG注释(左)、相同基因(中)和基因上下文(右)确定,用于广泛的参数。柱状图以5%的增量绘制。用黑色阴影的条形是绘制的参数图4.
图8在不同的系统发育距离上,细胞之间的水平转移是不同的。
一个比较全长16S rRNA和V68 16S rRNA区域的核苷酸识别切断,以避免密切相关细胞之间的比较。对于每一对HMP参考基因组,它们全长16S rRNA的核苷酸身份与它们的V68区域的核苷酸身份进行比对。97%的全长16S(对应大约7500万年的进化)身份被用作截止点,而当只有V68区域的序列可用时,95%被用作截止点。只有那些在全长和V68区域相似性均超过90%的基因组被显示出来。b,构成每一行的单元-单元比较的数量。c, HGT频率作为物种之间所有细胞-细胞比较(黑色)、仅HMP参考基因组(蓝色)和FijiCOMP单细胞组合(红色)之间的系统发育差异的函数绘制。这幅图只包含了可以识别全长16S rRNA基因的细胞。
图9为最终的移动基因数据集选择的具有代表性的基因与经过过滤以减少冗余的基因高度相似。
在参考基因组和单细胞组装之间的细胞-细胞BLASTn比较中观察到的每个重叠水平转移区域,基因聚类以识别唯一基因并减少基因集的冗余。这一步骤对读取比对后宏基因组数据集中这些基因的丰度进行精确测量至关重要。使用UCLUST对来自每个重叠水平传输区域的所有开放阅读帧进行分组。每个被过滤的基因的核苷酸特性和被选择用于读取对齐的基因(即质心)被绘制出来。
图10宏基因组在整个长度上以高保真度与移动基因对齐。
宏基因组的读取需要在至少50%的读取长度上与移动基因的99%身份一致。尽管截断率似乎很低,但在整个基因长度上,几乎所有的读取都与几乎完美的核苷酸身份一致。
权利与权限
关于本文
引用本文
布里托,伊尔马兹,S,黄,K。et al。人类微生物群中的移动基因从全球到个体尺度都是结构化的。自然535, 435-439(2016)。https://doi.org/10.1038/nature18927
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature18927
进一步的阅读
水平基因转移使合成菌群中可编程基因稳定
自然化学生物学(2022)
细菌的水平基因转移与适应性进化
自然评论微生物学(2022)
来自半孤立的人类群体肠道微生物组的质粒网络揭示了独特和共享的代谢和毒力特征
科学报告(2022)
人类肠道中的普雷沃氏菌属主要由copri普雷沃氏菌属、stercorea普雷沃氏菌属及相关谱系组成
科学报告(2022)
抗生素耐药基因的全球健康风险评估
自然通讯(2022)
