文摘
食管的癌症是全世界著名的;然而,很少有靶向治疗和为这些癌症存活率仍然低迷。164年我们进行一个全面的分子分析癌食管来自西部和东部的人口。除了已知的组织病理学和流行病学特征,分子功能分化从食管腺癌食管鳞状细胞癌。食管鳞状细胞癌和鳞状细胞癌的其他器官更比食管腺癌。我们分析确定三个分子食管鳞状细胞癌的子类,但没有证据显示人类乳头状瘤病毒的病原学的作用。鳞状细胞癌显示频繁基因组的扩增CCND1和SOX2和/或TP63,而ERBB2, VEGFA和GATA4和GATA6在腺癌通常放大。食管腺癌和胃腺癌的染色体不稳定的变异,表明这些癌症可以被视为一个单一的疾病实体。然而,一些分子功能,包括DNA甲基化,发生在食管腺癌的打击非常大。这些数据提供了一个框架来促进更合理分类这些肿瘤和新疗法的基础。
主要
食管肿瘤的5年存活率西方人群的12 - 20%1,2并导致每年全世界超过400000人的死亡3。食道癌被组织学分类为腺癌(EAC)或鳞状细胞癌(ESCC)4。EAC发病率增加了几倍近几十年来在西方国家5附近,主要发生在食管胃连接,并与肥胖有关,胃返流和前兆状态称为巴雷特食管。EAC利率上升平行增加近端胃癌的发病率6。ESCCs主要集中在上部和mid-oesophagus和与吸烟和酒精暴露在西方人群。在非西方国家,风险因素ESCCs不成立。
适当的划分之间的胃和食管腺癌和腺癌的分类横跨胃食管交界处(GEJ)仍未解决7,8,9,有争论关于组织学差别的效用4。提高食道癌分类,我们进行了全面的分子分析164食管肿瘤,359胃腺癌和36 GEJ额外的腺癌。我们评估方法分类食管肿瘤和分子识别特性和候选路径定义分子子组和提供潜在的治疗靶点。
样本收集和分子特性
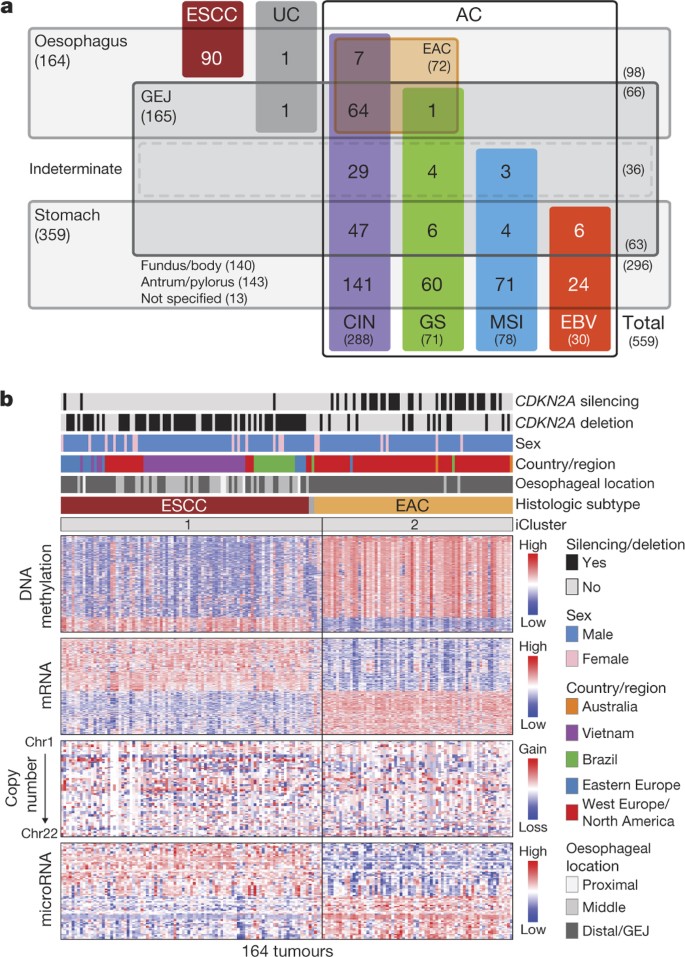
我们解决的挑战临床区分食管和胃通过审查原始GEJ附近的腺癌,腺癌使用解剖和病理标准数据,对肿瘤进行分类通过食管,胃或不确定的起源(图1一个,补充表1,补充图。1.1)。我们确认90 ESCCs 72 eac(61明确食管和11个可能的食管),36 GEJ癌起源于不定,63年胃GEJ癌(15定胃和48可能胃),140年胃底部的癌或身体,和143年胃或幽门窦的癌。我们无法本地化13胃胃内腺癌更狭隘,和2食管肿瘤未分化癌。
一个559食道癌和胃癌肿瘤分为样本集。CIN、染色体不稳定;EBV,巴尔病毒;GEJ胃食管结;GS基因稳定;MSI,微卫星不稳定。加州大学,未分化癌。b综合聚类四个分子平台显示食管癌分为两分子亚型(iCluster 1和iCluster 2)组织学类ESCC和EAC几乎相同。临床(上)和分子数据(底部)从164年肿瘤异形和所有四个平台进行描述。
用来进行肿瘤患者样本未接受化疗或放疗从多个国家获得知情同意和当地机构审查委员会批准。胚胎DNA收集血液或良性的食管粘膜。遗传物质受到whole-exome测序,单核苷酸多态性(SNP)阵列分析从而评估体细胞基因改变(大会),DNA甲基化分析和mRNA和微测序。51食管肿瘤的DNA受到低通(6 - 8×覆盖率)全基因组测序。反相蛋白数组上执行113年肿瘤蛋白质组学分析。
体细胞基因组改变食道癌
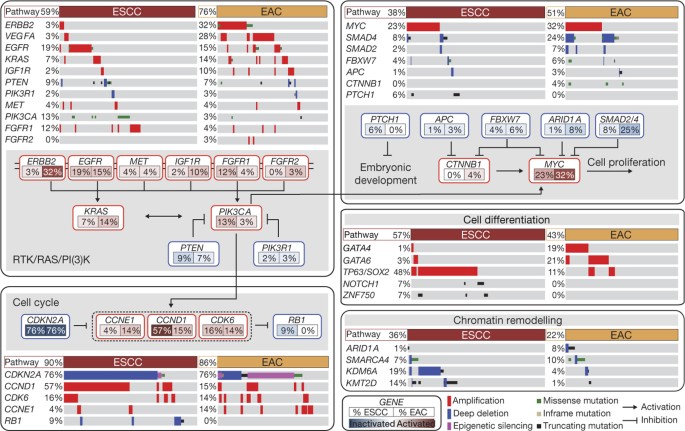
我们评估体细胞基因组变化分别使用MutSig ESCC和EAC13搜索与明显重复突变基因(扩展数据图3 a, b)。在ESCC,我们发现显著突变基因,TP53,NFE2L2,MLL2,ZNF750,NOTCH1和TGFBR2,与先前的研究一致14,15,16,17,18,19,20.。在EAC,我们识别重要的突变TP53,CDKN2A,ARID1A,SMAD4和ERBB2作为先前报道,21。这些研究结果是一致的重要性CDKN2A和TP53发育异常的突变巴雷特食管,EAC的前兆。同样的,我们分析与GISTIC大会做数据22定义循环地放大和删除区域(扩展数据图4;补充表2)。尽管EAC和ESCC共享一些反复出现的大会,我们证实疾病之间的实质性差异的变化模式19,23。大会在EAC复发(但没有ESCC)包括含放大VEGFA(6 p21.1),ERBB2(17 p12),GATA6(18 q11.2)CCNE1(19 q12),和删除SMAD4(18 q21.2)。反复出现的焦大会ESCC包括放大SOX2(3 q26.33)叔(5 p15.33),FGFR1(8 p11.23),MDM2(12 q14.3),NKX2-1(14 q13.2)和删除RB1(13 q14.2)。我们发现小说在3 p25.2 ESCC焦删除,包括河马的负调节通路VGLL4和自噬因素ATG7。
结合突变和大会做数据显示频繁改变细胞周期调控(图2)。失活的CDKN2A和放大CCND1在鳞状上皮肿瘤的76%和57%,分别;和额外的ESCCs放大的CDK6或损失的RB1。在eac细胞循环失调的模式不同,CCND1在只有15%的肿瘤是放大,但我们观察到更常见的放大CCNE1。CDKN2A在76%的eac灭活突变,删除或表观遗传沉默。这些数据显示的潜在作用的细胞周期激酶抑制剂治疗,尤其是ESCC。
为所选的基因和突变和大会CDKN2A表观遗传沉默eac和ESCCs所示。基因是分组的途径,用线条和箭头显示两两分子的相互作用。深删除显示超过一半的基因副本的损失。只有错义突变在宇宙库都包含。变更频率列出了每个基因内圆角矩形剩下ESCC利率和EAC,红色阴影表示基因激活,蓝色表示失活。
我们发现频繁变化的受体酪氨酸激酶和下游信号介质,特别是东盟。在ESCCs,我们确定放大或突变表皮生长因子受体19%的肿瘤和改变的PIK3CA,PTEN或PIK3R1,所有这一切被认为激活PI3K通路,在24%的肿瘤。eac有更广泛的潜在致癌放大,最常见的ERBB2改变在eac的32%,但只有3%的ESCCs。虽然临床试验导致的美国食品和药物管理局批准ERBB2-directed抗体曲妥珠单抗仅限于胃和GEJ腺癌24,ERBB2-positive eac通常与曲妥珠单抗治疗药物。值得注意的是,我们发现的突变ERBB2在四个肿瘤缺乏ERBB2放大,这意味着更多的病人可能受益于ERBB2-directed疗法。转录组数据发现6例ERBB2扩增外显子的融合转录本表达12ERBB2被融合到邻近基因3′端非翻译区JUP(补充图。3.1;补充表3)。因为这种融合转录省略了ERBB2跨膜和酪氨酸激酶域,其潜在的功能尚不清楚。其他eac显示放大的喀斯特,表皮生长因子受体,IGF1R或VEGFA。
额外的分析确定TGF-β通路的失调,数量较少CTNNB1(β-catenin)激活,在EAC比ESCC更常见。我们发现,6%的ESCCs(但没有eac)灭活改变的PTCH1如前所述,15刺猬信号,暗示激活。ESCC肿瘤,像其他鳞状癌症,有放大的染色体3 q,关注SOX2轨迹25。基因编码SOX2或鳞状转录因子p63 3 p号染色体上被放大在ESCCs的48%。此外,突变ZNF750和NOTCH1五月ESCCs同样调节鳞状细胞成熟15,16,17,18,19,20.。然而在eac,我们发现频繁放大的基因编码GATA4和GATA6发展因素,如胃腺癌26,27和(GATA6在EAC),实验验证28。
EAC和ESCCs显示改变chromatin-modifying酶(补充图。3.2)。改变影响瑞士/ SNF-encoding基因ARID1A,SMARCA4和叫做PBRM1在腺癌中较为常见,而ESCCs包含更频繁改变histone-modifying因素KDM6A(联合)KMT2D(MLL2)和KMT2C(MLL3)。因此,尽管许多相同的通路在eac和ESCCs不良改变,特定的基因影响是不同的,可能反映了不同的病理生理学和显示不同的治疗方法。这些数据告诫执行临床试验的种群混合eac和ESCCs。
食管鳞状细胞癌的分子亚型
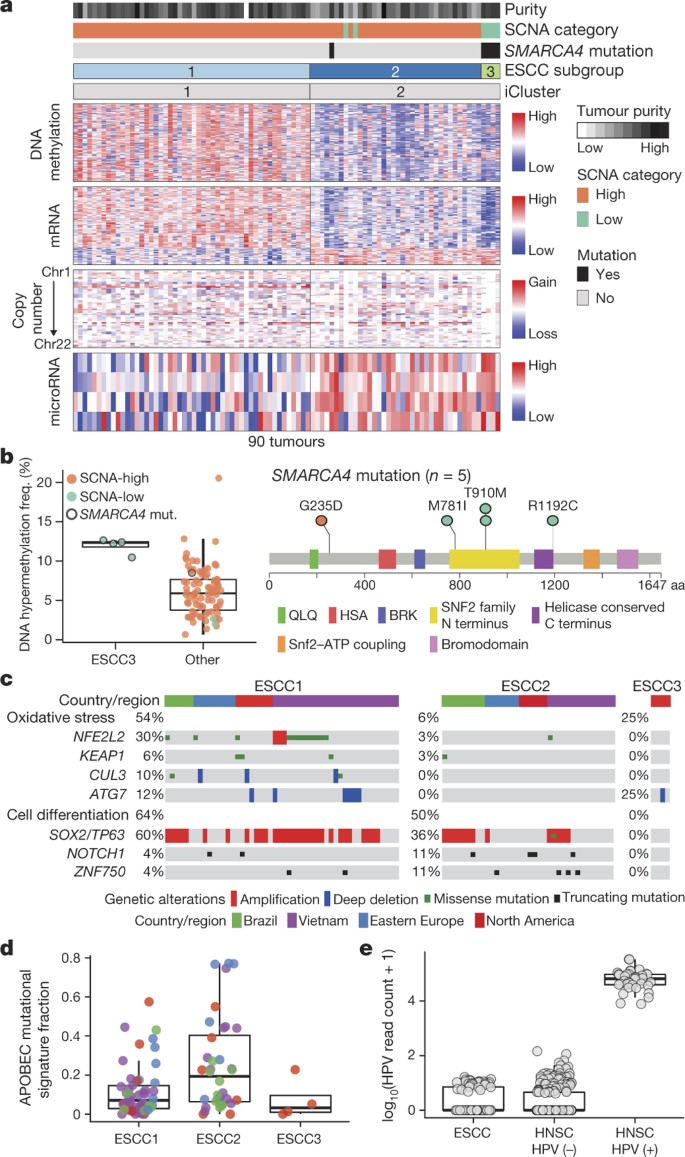
综合聚类使用iCluster ESCC数据显示两个类,表示iCluster 1和iCluster 2 (图3)。在iCluster 2,我们识别一群共享功能,包括突变的肿瘤SMARCA4(编码瑞士/ SNF因子缺失),增加DNA甲基化(图3,最右边的样品)和相对不变的大会资料(图3 b)。我们与这些特性指定不同的肿瘤亚型ESCC3,因此ESCCs划分成三个分子亚型:ESCC1 (n= 50),ESCC2 (n= 36)和ESCC3 (n= 4)。
一个,ESCCs分成亚型ESCC1 ESCC2 iCluster,与额外的识别集团ESCC3拥有SMARCA4突变和减少大会。临床和分子特性与分子数据列在上面。b(左)DNA甲基化ESCC3和其他ESCCs。对的,SMARCA4突变。c基因组的改变,影响氧化应激和细胞分化与样本ESCC亚型分离的地理起源。d,分数的突变APOBEC签字亚型和地理起源。e人类乳头状瘤病毒(HPV)转录水平在食管和头颈癌。
ESCC1被NRF2通路中的变化特点,调节适应氧化压力包括一些致癌物质和一些化疗药物。突变NFE2L2(NRF2)与预后不良和抵抗化疗相关联29日。改变被认为在NFE2L2在降低NRF2基因编码的蛋白质(KEAP1和CUL3),在ATG7编码一个NRF2通路自噬的因素30.,31日(图3 c)。ESCC1有更高的频率SOX2和/或TP63放大(图3 c,扩展数据图5)。ESCC1基因表达与描述的古典亚型癌症基因组图谱(TCGA)研究肺鳞状细胞癌32和头颈部鳞状细胞癌(HNSCC)33(扩展数据图6),它拥有类似的躯体变化。ESCC1显示更高的利率YAP1(11 q22.1)放大VGLL4 / ATG7删除,显示激活的河马。
ESCC2显示较高的突变NOTCH1或ZNF750(扩展数据图5),更频繁的灭活的改变KDM6A和KDM2D,CDK6放大,失活PTEN或PIK3R1。我们发现更多的白细胞浸润ESCC2肿瘤和更高水平的裂解Caspase-7蛋白(扩展数据图7所示),后者意味着潜力增强XIAP-directed代理促进细胞凋亡34。最低的基因P值之间的甲基化差异ESCC1 ESCC2是免疫调节分子BST2 (ref。35)(P= 3×10−4,确切概率法;补充表4少),这表明甲基化和高表达在ESCC2 (扩展数据图7所示),表明BST2抑制潜力。
ESCC3肿瘤显示,没有证据表明细胞周期的遗传放松管制和TP53只有一个突变的四个样品。然而,所有样品在ESCC3持续改变预测激活PI3K通路(扩展数据图5),三四个拥有躯体变化的KMT2D/MLL2除了SMARCA4。TCGA HNSCC数据集的分析与概要文件类似于ESCC3并没有发现肿瘤,建议这类可能仅限于ESCC鳞状上皮肿瘤。
ESCC亚型显示地理协会的趋势:从越南肿瘤患者,唯一的亚裔人口研究,往往是ESCC1(27的41 = 66%;P= 0.09,确切概率法),和更多的肿瘤来自东欧和南美患者ESCC2 (P= 0.118,确切概率法)。所有四个ESCC3肿瘤来自病人从美国和加拿大(P= 0.001,确切概率法)。从越南肿瘤患者丰富NFE2L2突变(图3 c);越南人群的24%(10的41)和6%在其他患者(3的49个;P= 0.017,确切概率法)。这个协会NFE2L2与越南患者显示了一个共同的氧化应激或突变遗传倾向。患者来自东亚的常见变异在酒精代谢的基因ALDH2和ADH1B36,这与ESCC风险相关联36,但是我们不可能调查他们的协会NFE2L2突变等所有越南患者变异(补充图。3.3)。
EAC相比,C >替换ESCCs显示浓缩和APOBEC(载脂蛋白B信使rna编辑酶,催化polypeptide-like)签名(P= 7×10−7和5×10−5分别由Wilcoxon rank-sum测试)。C >突变签名与吸烟和嚼烟37,但没有与ESCC子组或临床变量在我们的样例集。然而,当我们限制分析终身不吸烟者,C >签名明显高于在我们越南人口(PWilcoxon = 0.013),表明烟草咀嚼的角色。APOBEC签名在ESCC2过多(图3 d,P= 0.03,克鲁斯卡尔-沃利斯检验)和丰富的病人来自乌克兰和俄罗斯(P= 0.01,Wilcoxon rank-sum测试)。ESCC肿瘤缺乏偏爱> C在AA二核苷酸在EAC颠换(补充表5)。
我们评估是否人类乳头状瘤病毒(HPV),它有一个在宫颈鳞状细胞癌和HNSCC致病作用,也有助于ESCC,据报道38。比较ESCC mRNA测序数据,TCGA HNSCC数据发现ESCC HPV转录水平像HPV-negative HNSCC肿瘤(图3 e)。这些数据不支持ESCC aetiologic角色HPV。
EAC和胃癌的关系
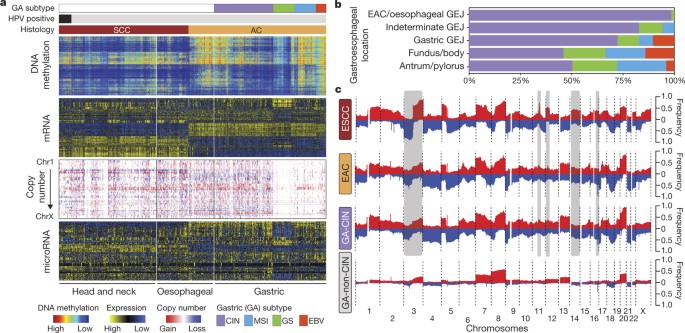
鉴于EAC的不确定性有关的适当划分相对于胃癌和ESCC,我们分析了EAC和ESCC相对于癌症发生的最近的食道,HNSCC和胃腺癌。分析mRNA的表达、DNA甲基化和大会做数据表明ESCC比EAC (HNSCC更强的相似之处图4)。同样,eac更比ESCC胃癌相似。在我们之前TCGA的研究27,我们胃肿瘤分为四个亚型的基础上有(1)eb病毒(EBV)感染,(2)微卫星不稳定性(MSI),(3)染色体不稳定(CIN)和(4)基因组稳定性(GS),一组主要由分散的组织学类型。当我们评估eac会同胃癌症,我们观察到eac和CIN胃癌症共同形成一组不同于EBV, MSI或GS肿瘤(扩展数据图8所示)。评估所有胃食管腺癌(gea),我们发现越来越流行的CIN向近端移动72年与71年eac分为CIN (图4 b)。没有eac MSI或EBV阳性。GEJ腺癌中,没有明显的食管的起源,我们确定了MSI-positive和EBV-positive肿瘤。
一个、分子的头部和颈部,食管和胃癌样本隔离和肿瘤类型的胃癌症细分分子亚型。b,胃分子亚型的解剖位置分布在胃食管腺癌。c、复合拷贝数ESCC概要,EAC, gastric-CIN和胃肿瘤non-CIN红色和蓝色和灰色的损失突出ESCC和EAC之间的差异。
CIN的浓缩EAC EAC的建议比较胃癌症将困惑non-CIN肿瘤几乎只在胃里。因此我们寻找功能,可以通过分析区分EAC CIN胃癌症的288 CIN赫亚(GEA-CIN;图1一个)。我们发现染色体畸变之间的明显的相似性在胃肿瘤CIN和EAC (图4摄氏度),相似性EAC和CIN胃癌症比EAC和ESCC之间。集群GEA-CIN数据从个人平台(扩展数据图9所示)和eac的综合聚类显示不一致的分离和CIN胃癌症,因此反对这些不同的疾病分类(扩展数据图10所示)。作为misannotation GEJ附近的肿瘤可以提高EAC的明显的相似性,CIN胃肿瘤,我们重复我们的分析排除模棱两可GEJ情况下,但没有看到明确的分离EAC和CIN胃腺癌(补充图。7.1)。
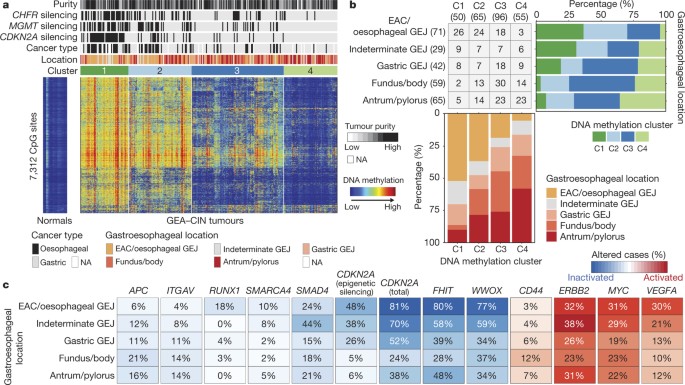
然而,集群的DNA甲基化数据显示DNA甲基化的功能从近端到远端GEA-CIN肿瘤(图5)。样品在集群1中,那些最常见的甲基化,丰富的食管或胃近端/ GEJ (图5 b)。癌症的比例显示更频繁的DNA甲基化(即集群1或2)在eac显著高于胃CIN癌症(分别为70%和30%;P= 1.0×10−8,确切概率法)。相比之下,集群4甲基化率最低的,包括更多的远端胃肿瘤(图5 b)。与hypermethylated胃CpG岛methylator表型的肿瘤,没有GEA-CIN肿瘤表观遗传沉默的展出一种,符合他们的MSI-negative地位,但是他们表现出较高的表观遗传沉默的倾向CDKN2A,(补充表6,图5度)。额外的基因沉默在集群1包括在内管理和CHFR,甲基化与烷基化反应有关代理和微管抑制剂,分别39,40。
一个,热点图表示DNA甲基化的共识集群GEA-CIN肿瘤分子和临床特征正常食管的上面和甲基化配置文件(n= 2)和胃(n左边= 13)。b属于每个甲基化组,部分肿瘤的解剖位置(右上角)和肿瘤解剖位置分布的甲基化集群(底部)。c,改变的频率选择基因与肿瘤抑制基因失活沿着解剖轴在红色蓝色和致癌基因的激活。
我们评估GEA-CIN肿瘤体细胞特征可以区分eac和胃CIN肿瘤(图5度)。eac突变的几率更高SMARCA4和删除的肿瘤抑制基因RUNX1低,但APC变异率相对于胃肿瘤,暗示在EAC Wnt /β-catenin不太突出的作用。人类基因组分析发现较高的假定的脆性位点基因的缺失FHIT基因或WWOX,提示潜在的基因组不稳定性的差异远端和近端之间GEA-CIN肿瘤。致癌基因的分析识别微妙的区别,VEGFA和MYC在eac放大更常见。尽管额外的样品需要改进的理解逐步渐变特性从远端胃食管,这些数据表明,胃和食管CIN肿瘤缺乏绝对的二歧的特性和似乎没有明显的肿瘤类型。
讨论
这些分析质疑预想的前提下食管癌作为一个单一的实体。这些分子数据显示,组织学亚型EAC和ESCC截然不同的分子特征在所有平台上测试。ESCC出现的疾病让人想起其他癌比EAC, CIN胃癌本身有着惊人相似之处。因此反对我们的分析方法,结合EAC和ESCC新辅助的临床试验,辅助或系统性疗法(补充图。3.4)。
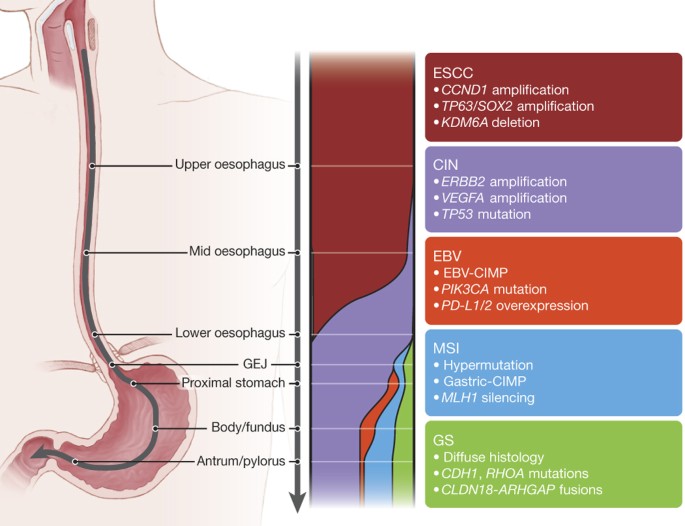
这些数据也通知长期争论关于适当的划分EAC的胃癌。我们发现赫亚显示累进亚型分类(图6),增加患病率的CIN表型向近端,以至于eac似乎代表一种染色体不稳定的疾病。这CIN梯度类似于大肠癌癌,CIN患病率增加远侧地向直肠41。EAC一直被视为独立于胃癌模型显示,EAC源于巴雷特食管,因此不是胃的起源。虽然起源的巴雷特食管仍有争议,最近的小鼠模型表明,巴雷特食管和EAC可能来源于近端胃细胞或胚胎残余GEJ细胞群42,43。的分子相似性EAC和CIN胃癌症提供间接支持胃巴雷特食管的起源和EAC和表明我们可能把赫亚视为一个单一的实体,类似地,结直肠腺癌。然而,这些相似性EAC和CIN胃癌症并不表明CIN赫亚都无法区分。事实上,差异更近端赫亚应该预期,鉴于其独特的流行病学、西方国家的快速增长和逆协会幽门螺杆菌。继续探索EAC可能不是绝对的分子特征区分他们从CIN胃癌症,但可能揭示丰富的附加功能赫亚的变体。
示意图表示将胃食管癌亚型的比例从近端向远端胃食管。彩色带的宽度代表的比例亚型解剖区域内。关键特性的子类型相关的文本表示。
方法
数据报告
没有统计方法被用来预先确定样本容量。实验并不随机,调查人员没有被分配在实验和结果评估。
标本收集和分期
组织来源(TSS)中列出的网站补充信息S1.1。食管肿瘤收集和运送到一个中央Biospecimen核心资源(BCR) 2011年12月1日至2013年12月23日。样本从患者获得之前没有收到任何化疗或放射治疗他们的疾病。每个冻结主要肿瘤标本有同伴正常组织标本(血液或血液成分,包括DNA提取的TSS)。相邻nontumourous食管组织也提交给病人的一个子集。
例了根据美国癌症联合委员会第七版分期系统44。病理质量控制进行每个肿瘤和邻近的正常组织标本(如果可用)冰冻切片幻灯片确认肿瘤标本组织学检查符合食道癌,邻近组织标本中不含肿瘤细胞。肿瘤细胞核肿瘤样本与≥60%,≤20%坏死提交核酸提取。
核酸处理和资格
DNA和RNA co-isolated,质量评估在中央BCR如前所述(补充S1.1在裁判。27)。自定义Sequenom SNP面板或AmpFISTR Identifiler(应用生物系统公司)是用于验证肿瘤DNA和DNA生殖系代表来自同一个病人。RNA进行了分析通过RNA6000纳米分析(安捷伦)来确定一个RNA完整性号码,只有分析物有一个完整的编号≥7.0是包括在内。只有情况下,收益率最低的6.9μg肿瘤DNA, 5.15μg RNA和4.9μg生殖系的DNA被包括在内。
BCR收到肿瘤与生殖系控制样品共322例食道癌病例,其中185合格,BCR病理学的基础上审查和分子特征。分布和质量控制情况下所示补充图。1.1。合格的185例病例中,171例用于基因组分析,独立的病理检查后14例被排除在外(中描述“病理学专家评审”,下文)或发现的临床分子disqualifiers。
的171名符合条件的情况下,匹配nontumourous 58例食管组织。后与残余肿瘤组织样本提取核酸被认为是进行蛋白质组学分析。当可用时,一块10 - 20毫克的snap-frozen肿瘤邻近块用于分子测序和描述提交反相蛋白组分析。我们比较这171食管腺癌388同样具有胃腺癌(补充图。1.1)。
微卫星不稳定性分析
微卫星不稳定性(MSI)合格的食管腺癌肿瘤提取DNA样本被BCR评估全国儿童医院,美国俄亥俄州哥伦布市。MSI-mono-dinucleotide试验进行测试的四个单核苷酸重复位点(polyadenine大片BAT25, BAT26, BAT40和转化生长因子受体II型)和三个二核苷酸重复位点(CA重复在D2S123 D5S346和D17S250)如前所述27。
病理学专家审查
所有癌症包括在本研究其次综述了由一个专家病理学家的委员会,由七个经验丰富的胃肠道病理学家、,S.McC。,Z.Z.,J.K.,L.T.,M.B.P. and J.W.). A centralized virtual pathology review system was constructed using an Aperio slide scanner housed at the BCR at Nationwide Children’s Hospital. Typically, two frozen sections flanking the tumour tissue from which all material was extracted for this study and one additional high-quality formalin-fixed paraffin-embedded tissue section were scanned and reviewed. Two committee members reviewed all cases before inclusion into the study. For cases with discrepant results, a tiebreaker reviewer was assigned.
食管肿瘤都归类为鳞状上皮或腺癌,根据世界卫生组织分类的消化系统肿瘤,第四版45。9例排除病理的基础上审查,包括四个质量控制确认材料分析不足的情况下,两种情况下只观察非侵入性肿瘤,2例,肿瘤不可归类的基础上可用的材料进行审查。作为评估的一部分,一个额外的77胃腺癌,没有经历了病理检查作为集团发表的分析的一部分也受到病理学re-review之前执行27。
临床分期评估44由两个评论家根据标准每个肿瘤类型(ESCC或EAC)。T、N和M状态和肿瘤级别(0、1、2或3)是基于TSS的病理报告。
解剖腺癌涉及GEJ的子分类
腺癌(食管或胃)从附近的一个潜在来源的TCGA集合GEJ提炼他们的解剖位置进行进一步审查。病理报告与原总从tss获得肿瘤切除或内镜活检的病理描述。两个独立的临床审查员审查每个TSS病理报告。肿瘤分为食管,可能食管,不定,可能胃或胃,根据标准中列出补充信息S1.2。对于下游分析,食管和可能的食管的组合在一起,可能是胃和胃。
体细胞人类基因组分析
大会进行分析的基础上,每个肿瘤或生殖系样本的DNA分析Affymetrix SNP 6.0的基因组分析平台Broad研究所如前所述46。作为这个过程的一部分人类的评估和分类,相应区域从而生殖系基因变化被通过应用过滤器从TCGA的生殖系样本生成我们的卵巢癌分析或从样品在这个集合。分析复发性广泛和焦大会GISTIC 2.0执行算法22使用集群在R,欧几里得距离的基础上使用阈值在重复拷贝数改变山峰从GISTIC分析使用沃德的方法,如前所报道27。等位基因的拷贝数和使用绝对纯洁和倍性估计计算算法47。肿瘤被划分高染色体不稳定,SCNA-high,如果他们拥有至少一个arm-level损失(除了18 p, q 18或21日在肿瘤复发性低和高的人类事件),否则SCNA-low。染色体臂被认为改变如果失去了至少80%的手臂或获得相关日志2复制比率的变化至少0.15(施等未发表的观察)。这种分类方法拷贝数不稳定有93%的一致性与之前描述人类集群27。
DNA甲基化
基因组DNA(每样1μg) bisulfite-modified,进行质量控制,并使用Illumina公司英飞纳姆DNA甲基化分析平台,HumanMethylation450,详细S2的补充信息。生成数据文件中列出补充信息S2.3。
CDKN2A表观遗传沉默的电话
CDKN2A(也称为p16INK4)表观遗传沉默调用是使用DNA甲基化和RNA-seq数据。CDKN2A在每个样本DNA甲基化状态评估基于探针(cg13601799)位于p16INK4发起人CpG岛。p16INK4表达式是由日志2(RPKM + 1)水平的第一外显子(chr9: 21974403 - 21975132)。每个样品的表观遗传沉默的电话是由评估一个散点图显示一个逆中描述DNA甲基化和表达之间的联系S2的补充信息。
DNA序列分析
外显子组和足额的全基因组测序是分成两个测序中心。样本,提交TCGA胃腺癌(STAD,标签的TSS)被用来测序Broad研究所。测定样品贴上食管肿瘤(ESCA)是华盛顿大学测序。每个中心负责生成BAM文件从肿瘤和正常的DNA样本测序过程的附加过滤除去可能的文物。从这些BAM文件,TCGA四个不同的分析网站执行不同的突变和插入/删除检测程序。集成这些不同mutation-calling努力的结果是生成一个常见变异注释文件进行后续分析。看到补充部分S3.1。
Broad研究所测序
Whole-exome测序DNA的0.5到3μg肿瘤和正常的血液样本进行如前所述32使用安捷伦SureSelect人类所有外显子V5装备,其次是2×76 - bp在Illumina公司HiSeq paired-end测序平台。全基因组测序,2×101 - bp读取测序在同一平台。读一致性和处理进行使用burrows - wheeler对准器(BWA)和皮卡德Broad研究所(http://broadinstitute.github.io/picard/)以前公布的27。联盟第一次使用比赛进行质量控制48为了避免misannotation肿瘤和生殖系DNA样本,或肿瘤样品之间的交叉污染。只有样品估计不到5%交叉污染进一步进行分析。
华盛顿大学测序
Whole-exome测序和全基因组Illumina公司库建立了如前所述49使用罗氏SeqCap EZ人类外显子组库v3.0加上额外120 - mer IDT定制调查,针对癌症相关的DNA病毒(例如,HPV, EBV)和测序在多个车道的Illumina公司HiSeq 2000流细胞达到最小覆盖的20×80%的编码外显子目标。每个车道或sub-lane数据对齐使用BWA v0.5.9。GRCh37-lite +增加目标病毒(ftp://genome.wustl.edu/pub/reference/GRCh37-lite_WUGSC_variant_2/)。
体细胞突变的识别和插入/删除
BAM文件(外显子组测序)是用于调用在四个不同的突变分析中心:Broad研究所,华盛顿大学,加州大学圣克鲁斯和不列颠哥伦比亚癌症研究中心(如详细补充方法S3.1)。
如上所述过滤电话每个分析中心合并,和生殖系SNP网站报道的1000人基因工程是过滤和删除。此外,对于正常的生殖系BAM,假定的变体少于8×的报道引用等位基因或大于一个体细胞variant-supporting读或1%体细胞变异等位基因分数被移除。肿瘤的BAM,两个支持读取和等位基因变体作为最低需要分数的5%。过滤的推定地假突变称由于8-oxoguanine文物进行去除候选基因突变归因于这些测序文物。进一步过滤去除候选基因突变,通过测序确定群non-neoplastic DNA样本删除替代文物或未经过滤的生殖系电话。阅读数量生成的所有剩余小说假定的变异,这些变异被纳入最终的变异注释文件如果他们遇到相同的最小覆盖,最大覆盖率,等位基因变体分数上面描述的要求。
变异注释和意义分析
执行功能注释的突变与Oncotator (http://www.broadinstitute.org/cancer/cga/oncotator)使用Gencode V18。显著地突变基因被确定使用MutSigCV2.0算法13。
突变特征分析
突变签名发现是使用贝叶斯非负矩阵分解算法执行变异特征分析中描述补充信息S3.2。
低通全基因组测序的重排识别
基因组DNA (500 - 700 ng /样本)被剪成250 - bp片段使用Covaris E220超声发生器,然后使用KAPA生物转化为paired-end Illumina公司库套件与卡尺(PerkinElmer)机器人挥动套件(Partek基因组学)根据制造商的协议。所有图书馆都使用一个样本测序在HiSeq2000每车道与paired-end 2×51-bp读取长度。肿瘤DNA及其匹配正常DNA通常是装载在同一流动单元。原始数据被转换为FASTQ格式,BWA对齐(hg19)被用来生成文件如前所述的BAM (补充S3.6在裁判。27)。检测使用两种算法进行结构重组,跳霹雳舞50和猫鼬51。组结构变体调用从每个肿瘤样本被调用的匹配过滤正常DNA切除生殖系变异。数据然后使用猫鼬重新检验算法,它需要至少两对不和谐的阅读的识别,和一个读覆盖实际断点连接。改变中找到简单或卫星重复也被排除在外。(候选人融合基因从这个分析所示补充表3结构改变的更详细的清单补充表7。)
信使核糖核酸测序和分析方法
信使rna序列数据生成的如前所述(补充S5.1在裁判。27)。结合聚类分析的食管、胃、头部和颈部肿瘤,北卡罗莱纳大学基因组特征中心再加工胃腺癌和食道癌数据MapSplice / RSEM管道32。我们生成的候选人从信使rna序列数据融合事件如前所述(补充S5.4在裁判。27),除了我们使用TransABySS v1.4.8 (http://www.bcgsc.ca/platform/bioinfo/software/trans-abyss/releases/1.4.8)。
识别子类型在我们的各种人群,我们使用层次聚类与pheatmap v1.0.2 r .输入在每种情况下是每千碱基读取每百万读取映射到外显子的转录组(RPKM)数据矩阵前25%最变量意味着大于10 RPKM基因。我们通过日志转换矩阵的每一行10(RPKM + 1),然后使用pheatmap规模行。我们使用了病房。D2相关性和欧氏距离的聚类方法和措施为集群列和行,分别。我们确定差异表达的基因,使用未配对的两种意义分析微阵列(samr v2.0),与一个RPKM输入矩阵和错误发现率阈值为0.05。
比较食道癌亚型HNSCC建立亚型52和肺鳞状细胞(LUSC)肿瘤53,重心基因表达谱用来分类90食管鳞状上皮肿瘤为典型,基底,HNSCC古典和间叶细胞的分类;和基底,古典、原始和分泌LUSC分类。的839个基因用于HNSCC重心,809年与基因重叠ESCC数据集。此外,209年的基因用于LUSC预测重心,202重叠ESCC的基因数据集。然后我们生成一个RPKM矩阵90份ESCC肿瘤样本的每一个基因集。这些矩阵是日志2转换和median-centred。最后,我们计算每列之间的皮尔逊相关性矩阵和HNSCC LUSC重心。
评价食管的mRNA表达相对于其他类型的肿瘤,我们通过期望最大化结合RNA序列从STAD RSEM-normalized表达数据,光电子能谱和HNSC同志们。样品被命令首先器官,然后通过组织学(腺癌或鳞状),然后通过胃癌的分类类别(EBV, MSI, GS或CIN)最后由HPV状态。我们选择前25%最变量(通过变异系数)基因在食管内癌样本集与意思表达大于1000 RSEM-normalized计数。我们通过日志转换矩阵的每一行10(RSEM + 1),然后使用pheatmap规模和集群的行。
微测序和分析
我们生成的微rna序列数据如前所述补充S6.1在裁判。27)。识别子类型在我们的各种人群,我们使用层次聚类与pheatmap v1.0.2 r .每种情况的输入是reads-per-million (RPM)数据矩阵为303年miRBase v16 5 p或者3 p成熟股最大的差异在每个队列。我们通过日志转换矩阵的每一行10(RPM + 1),然后使用pheatmap规模行。我们使用了病房。D2相关性和欧氏距离的聚类方法和措施为集群列和行,分别。分析比较食管和胃和头部和颈部癌症,我们使用了前25%(300年~)大多数变量5 p或者3 p成熟链小分子核糖核酸54食管内癌样本集。我们通过日志转换矩阵的每一行10(RPM + 1),然后使用pheatmap规模行。为集群的行中,我们使用病房。D2和欧氏距离度量。
反相蛋白数组
蛋白质分离肿瘤被用来准备反相蛋白数组与187年验证主要抗体通过前面描述的方法(补充S7在裁判。27)。数据规范化,聚类分析进行详细补充部分S4。
病原体的分析
我们使用两个工具来检查whole-exome微生物的存在和RNA序列数据序列:《(BioBloomTools v1.2.4.b1)和PathSeq。这些提供了分析的细节补充部分S5。微数据分析使用一个内部管道(如前所述)(补充S9.2在裁判。27)。
信使rna的途径分析
我们执行pathway-level基因表达分析比较EAC和ESCC样本。组基因通路,从国家癌症研究所获得的途径相互作用数据库(NCI-PID)55。一个P价值,比较EAC和ESCC利用克鲁斯卡尔-沃利斯单向方差分析,得到每个基因。为每个224通路,能够p值对数转换,总结通过使用一种方法根据费雪的统计结合产生pathway-level综合得分。这个分数的统计显著性是通过类似的经验估计为每个NCI-PID途径得分10000随机生成的路径,在匹配路径的大小。
综合聚类
发现肿瘤样本跨平台共享分子特征,以下四个综合聚类方法被使用:iCluster,多个内核的学习k——(MKLk),则超星系团,集群的集群作业(可口)。在iCluster方法10,56,57子组被发现通过他们的代表,潜变量联合多元回归。MKLk——结合了k聚类算法则使用内核编码样本之间的相似性,为分类定义特征的肿瘤。超星系团和可口从个体分子中都使用集群平台形成一个整体分类的描述每一个样本,但他们不同的细节,如指标用于比较的样本。超星系团进行差异调整,这样每个分子平台收到平等的重量,而可口的实现使用这里之前(补充S10.2在裁判。27)使用加权法,考虑部门的粒度在每个特定于平台的类别。对这些方法给出更多细节补充部分S7。
数据可用性
主要用于生成本文提供的分析和处理数据可以从TCGA手稿出版页面,下载(https://tcga-data.nci.nih.gov/docs/publications/esca_2016),从基因组数据共享(https://gdc-portal.nci.nih.gov/legacy-archive)。
引用
旧金山,r . et al .癌症生存在欧洲1999 - 2007年国家和年龄:EUROCARE-5-a人群为基础的研究的结果。柳叶刀杂志。1523-34 (2014)
西格尔,r . L。,Miller, K. D. & Jemal, A. Cancer statistics, 2016.CA癌症j .中国。66年7-30 (2016)
老爹,洛杉矶等。全球癌症统计数据,2012年。CA癌症j .中国。65年87 - 108 (2015)
西j . r . &奥特k .食管鳞状上皮和腺癌是相同的疾病?Semin。Radiat。肿瘤防治杂志。1738-44 (2007)
布朗,l . M。,Devesa, S. S. & Chow, W. H. Incidence of adenocarcinoma of the esophagus among white Americans by sex, stage, and age.j .国家的。癌症本月。One hundred.1184 - 1187 (2008)
Devesa s s & Fraumeni j·F。,Jr. The rising incidence of gastric cardia cancer.j .国家的。癌症本月。91年747 - 749 (1999)
大米、t·W。,Blackstone, E. H. & Rusch, V. W. 7th edition of the AJCC Cancer Staging Manual: esophagus and esophagogastric junction.河南安171721 - 1724 (2010)
Suh y s . et al . esophagogastric结的腺癌应归类为食道癌吗?比较分析根据第七与TNM分类。安。杂志。255年908 - 915 (2012)
抛媚眼,j . m . et al .临床特点、生物学行为和生存食管切除术后胃食管腺癌的类似结和远端食管。j . Thorac。Cardiovasc。杂志。138年讨论,594 - 602,601 - 602 (2009)
沈,R。,Olshen, A. B. & Ladanyi, M. Integrative clustering of multiple genomic data types using a joint latent variable model with application to breast and lung cancer subtype analysis.生物信息学252906 - 2912 (2009)
Carneiro, p . et al .上皮功能障碍在胃cancer-cellular后果,临床应用和开放的问题。2月。586年2981 - 2989 (2012)
巴比里,c, E。唐,l . J。,Brown, K. A. & Pietenpol, J. A. Loss of p63 leads to increased cell migration and up-regulation of genes involved in invasion and metastasis.癌症Res。66年7589 - 7597 (2006)
Lawrence m . s . et al .异质性突变在癌症和寻找新的癌症相关的基因。自然499年214 - 218 (2013)
程,c . et al。全基因组测序揭示了不同模型的结构变化在食管鳞状细胞癌。点。j .的嗡嗡声。麝猫。98年256 - 274 (2016)
高,y . b . et al,食管鳞状细胞癌的遗传景观。Nat,麝猫。461097 - 1102 (2014)
林特区et al .基因组和分子食管鳞状细胞癌的特征。Nat,麝猫。46467 - 473 (2014)
秦,h . d . et al .食管鳞状细胞癌的基因特征揭示了关键基因潜在的肿瘤发生和预后不良。点。j .的嗡嗡声。麝猫。98年709 - 727 (2016)
泽田师傅,g . et al .基因组景观食管鳞状细胞癌的日本人口。胃肠病学150年1171 - 1182 (2016)
歌,y等。在食管鳞状细胞癌基因改变的识别。自然509年91 - 95 (2014)
张,l . et al .基因组分析揭示突变签名和经常改变基因在食管鳞状细胞癌。点。j .的嗡嗡声。麝猫。96年597 - 611 (2015)
Dulak, a . m . et al .外显子组和全基因组测序的食管腺癌识别复发司机事件和突变的复杂性。Nat,麝猫。45478 - 486 (2013)
Mermel, c . h . et al . GISTIC2.0促进敏感和自信的本地化的目标焦点体细胞人类基因组改变在人类癌症。基因组医学杂志。12R41 (2011)
Bandla, s . et al .比较基因组学食管腺癌和鳞状细胞癌。安。Thorac。杂志。93年1101 - 1106 (2012)
爆炸,y . j . et al .曲妥珠单抗联合化疗与单独化疗治疗her2阳性癌症晚期胃或gastro-oesophageal结(宽外袍):第三阶段,非盲、随机对照试验。《柳叶刀》376年687 - 697 (2010)
鲈鱼,a . j . et al。SOX2是一个放大lineage-survival致癌基因在肺和食管鳞状细胞癌。Nat,麝猫。411238 - 1242 (2009)
Dulak, a . m . et al .胃肠道腺癌的食道、胃和结肠表现出截然不同的基因组不稳定和肿瘤形成的模式。癌症Res。72年4383 - 4393 (2012)
癌症基因组图谱的研究网络。全面的胃腺癌的分子特征。自然513年202 - 209 (2014)
林,l . et al .激活GATA结合蛋白6 (GATA6)维持致癌lineage-survival食管腺癌。Proc。《科学。美国109年4251 - 4256 (2012)
柴田则t . et al . NRF2突变带来恶性潜能和抵抗放化疗治疗晚期食管鳞状上皮癌。瘤形成13864 - 873 (2011)
小松,m . et al。压力反应的选择性自噬衬底p62激活转录因子Nrf2通过Keap1的失活。Nat,细胞生物。12213 - 223 (2010)
田口,k . et al . Keap1自噬降解氧化还原体内平衡的维护。Proc。《科学。美国109年13561 - 13566 (2012)
癌症基因组图谱的研究网络。全面的鳞状细胞肺癌基因组特征。自然489年519 - 525 (2012)
癌症基因组图谱网络。全面的基因组头颈部鳞状细胞癌的特征。自然517年576 - 582 (2015)
Twiddy D。,Cohen, G. M., Macfarlane, M. & Cain, K. Caspase-7 is directly activated by the approximately 700-kDa apoptosome complex and is released as a stable XIAP-caspase-7 approximately 200-kDa complex.生物。化学。281年3876 - 3888 (2006)
李,美国x et al . Tetherin / BST-2促进树突状细胞激活在急性逆转录病毒感染和功能。科学。代表。620425 (2016)
崔,r . et al .功能变体在ADH1B和ALDH2加上酒精和吸烟协同提高食道癌的风险。胃肠病学137年1768 - 1775 (2009)
Alexandrov l . b . et al .签名人类癌症的突变过程。自然500年415 - 421 (2013)
Petrick, j·l . et al。人类乳头状瘤病毒患病率在食管鳞状细胞癌病例:系统回顾和荟萃分析。Br。j .癌症110年2369 - 2377 (2014)
哈西娜,r . et al . O-6-methylguanine-deoxyribonucleic酸甲基转移酶甲基化增强应对temozolomide治疗食道癌。j . Carcinog。1220 (2013)
Yun, t . et al .甲基化CHFR糖分会让食管鳞状细胞癌多烯紫杉醇和紫杉醇。癌症的基因638 - 48 (2015),
癌症基因组图谱网络。全面的分子表征人类结肠癌和直肠癌。自然487年330 - 337 (2012)
Wang x et al .剩余胚胎细胞的前体巴雷特它们上皮化生。细胞145年1023 - 1035 (2011)
Quante, m . et al .胆汁酸和炎症激活胃贲门干细胞在小鼠模型Barrett-like化生。癌症细胞2136-51 (2012)
边缘,s . et al。(eds)与癌症分期手册(Springer,纽约,2010)
那一刻,f . T。,Carneiro, F., Hruban, R. H. & Theise, N. D. (eds)消化系统肿瘤的分类(国际癌症研究机构,2010年)
McCarroll, s . a . et al。综合检测和基因的单核苷酸多态性分析和拷贝数变异。Nat,麝猫。401166 - 1174 (2008)
卡特,s . l . et al .绝对量化体细胞的DNA改变在人类癌症。生物科技Nat。》。30.413 - 421 (2012)
Cibulskis k . et al .竞赛:估计人类样本在新一代测序数据的交叉污染。生物信息学272601 - 2602 (2011)
Kandoth, c . et al。集成化的子宫内膜癌基因特征。自然497年67 - 73 (2013)
陈,k . et al .霹雳舞:基因组结构变异的高分辨率的映射算法。Nat方法。6677 - 681 (2009)
杨,l . et al。不同机制的体细胞在人类癌症基因组结构变化。细胞153年919 - 929 (2013)
沃尔特,诉等。分子亚型在头部和颈部癌症表现出不同的模式规范的癌症基因的染色体得失。《公共科学图书馆•综合》8e56823 (2013)
威尔克森,m . d . et al .肺鳞状细胞癌mRNA表达亚型是可再生的,临床上重要的,对应于正常细胞类型。中国。癌症Res。164864 - 4875 (2010)
Kozomara, a & Griffiths-Jones s miRBase:注释高信心microrna使用深度测序数据。核酸Res。42D68-D73 (2014)
Schaefer, c . f . et al . PID:通路相互作用数据库。核酸Res。37D674-D679 (2009)
沈,r . et al。综合使用iCluster亚型发现胶质母细胞瘤。《公共科学图书馆•综合》7e35236 (2012)
莫问:et al .模式发现和癌症的基因识别综合癌症基因组数据。Proc。《科学。美国110年4245 - 4250 (2013)
确认
我们感谢所有患者导致这项研究中,k . Hoadley和r . Kucherlapati科学编辑,j . Zhang和i Felau行政支持。这项工作得到了校内的研究项目和下面的资助来自美国国立卫生研究院:5 u24ca143799 5 u24ca143835 5 u24ca143840 5 u24ca143843 5 u24ca143845 5 u24ca143848 5 u24ca143858 5 u24ca143866 5 u24ca143867 5 u24ca143882 5 u24ca143883 5 u24ca144025 U54HG003067, U54HG003079, U54HG003273 P30CA16672。
作者信息
作者和联系
财团
贡献
癌症基因组图谱研究网络贡献集体研究。Biospecimens组织源提供的网站和处理Biospecimen核心资源。数据生成和分析基因测序中心,癌症Genome-characterization中心和基因组数据分析中心。所有数据通过数据协调中心被释放。美国国家癌症研究所和国家人类基因组研究所项目团队协调项目活动。TCGA以下调查食管的分析工作小组作出了显著贡献的分析和写作手稿。项目负责人:亚当·j·巴斯,彼得·w·Laird Ilya Shmulevich;数据协调员:Vesteinn Thorsson;分析协调员:旧金山Sanchez-Vega Vesteinn Thorsson;手稿协调员:芭芭拉·g·施耐德;图形协调员:Toshinori Hinoue;DNA序列分析:安德鲁•Dunford Jaegil金,Michael d . McLellan Angeliki Pantazi,嘉莉Cibulskis,梅兰妮Kucherlapati, Peter j .公园,力行杨;塞缪尔·r·迈耶;信使rna分析:Reanne鲍比,安德鲁j . Mungall;microrna的分析:Reanne鲍比;DNA甲基化分析:彼得·w·Laird Toshinori Hinoue;人类基因组分析:安德鲁·d·Cherniack Juliann施;蛋白质分析:Ju-Seog李,之一Apurva对冲基金,Rehan Akbani;路径/综合分析:Francisco Sanchez-Vega Varsha Dhankani,克里斯蒂娜·柯蒂斯,何塞·安东尼奥·Seoane Ronglai沈,本杰明·j·拉斐尔Hsin-Ta Wu亚历山德拉m . Wong Vesteinn Thorsson,尼古拉斯Schultz Arshi Arora;病理的专业知识和临床数据:亚历克斯Boussioutas,芭芭拉·g·施耐德,大卫Kelsen,罗伯特·d·Odze香农j·考尔,肯尼思•王Arjun Pennathur,约瑟夫·e·威利斯,玛格丽特·l·加里,凯瑟琳·s . Garman m·布兰卡Piazuelo莎拉Derks,克里斯汀·m·Leraas塔拉·m··利希滕贝格John a . Demchok David g .啤酒,布莱恩·j·里德众人周劳拉·唐Jihun Kim于jaf a Ajani;微生物分析:Charles s . Rabkin玛格丽特·l·加里Reanne鲍比,钱德拉Sekhar Pedamallu, Sara Sadeghi Akinyemi Ojesina,苏珊Bullman,凯伦Mungall。
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
额外的信息
审核人信息
自然由于美国麦格雷戈和其他匿名审稿人(s)为他们的贡献的同行评审工作。
扩展数据数据和表
图1扩展数据的特定于平台的无监督聚类分析食管的癌症。
一个- - - - - -e无监督聚类的食管肿瘤基于DNA甲基化(一个),大会(b)、基因表达谱(c),microRNA概要文件(d)和反相蛋白数组数据(e)显示强大的分离EAC和ESCC多个分子平台。样品显示为列。EAC,食管腺癌;ESCC,食管鳞状细胞癌;加州大学,未分化癌。
图2扩展数据通路显著表达差异EAC和ESCC。
一个,NCI PID路径表达式EAC和ESCC之间显著不同(P年代< 10−3,在那里P年代统计学意义的途径得分(见方法))列出。色标显示中值(日志2)表达式值的显著差异表达基因(P< 10−3)在相应的路径,归一化单元范围。bEAC TP63ΔN转录水平测量,固体组织正常,ESCC样本。c、中值NCI-PID通路中的基因表达的基因值的验证转录目标ΔN p63亚型的EAC和ESCC。每个点代表一个样本,该值的中值表达式值46通路中的基因。
扩展数据图3 MutSig显著突变基因在EAC和ESCC的分析。
一个、情节显著突变基因的MutSigCV2从EAC whole-exome测序数据样本的计算分析。基因是由水平的意义(问在正确的值作为绘制)。左边是每个突变的患病率在样例集。彩色框显示样本与特定的突变,突变的类型标签的盒子颜色,传说在右上角。上面显示了每个样本的变异数图(Syn)同义和非同义(非Syn)突变分别绘制。底部图显示,突变的等位基因的分布分数样品测序。b,显示了ESCC MutSig情节一样EAC样品上面。
扩展数据图4 GISTIC疫源地分析复发性放大和删除。
这些数据显著的展示焦点复发焦点放大和删除确定从GISTIC 2.0从SNP分析体细胞拷贝数数据数组。独立的情节CIN-gastric癌症(左)所示,EAC(中间)和ESCC(右)。每个情节数组X染色体1(上)(下)和显示病灶的显著放大(底部的左边,红与规模)或删除(对,蓝色与规模上)。候选目标的焦点放大或删除标签所示的各自的峰值。山峰没有明确目标是由染色体带标签。在括号中表示数量的基因数量在每个峰值GISTIC计算。标有星号的基因很可能司机位于相邻峰值区域由GISTIC定义的。
扩展数据图5的比较体细胞改变ESCC和HNSC亚型。
选择基因突变和人类基因组变化选择信号通路所示三ESCC亚型识别在我们的研究和HPV-negative (n= 243)和患有乳腺癌(n= 36)亚型HNSC以前被TCGA的研究。放大和深度缺失表明超过一半的基线的改变基因副本。错义突变包括如果他们被发现在宇宙库。变更频率表示为比例的改变情况下在每个分子亚型。底部面板显示比例的改变情况下,每个信号通路分子亚型和比例的改变情况下每每个信号通路的分子亚型。
图6不同的集群ESCC扩展数据。
列表明皮尔逊相关性的mRNA概要90 ESCC肿瘤的质心mRNA表达分析发达的亚型肺鳞状细胞癌(LUSC,顶部)和头颈部鳞状细胞癌(HNSC,底部)基因表达分析。样品在ESCC集群顺序图3。
扩展数据图7描述ESCC亚型。
一个展示,我们发现基因表观遗传沉默相比,个别样品和样本的数量,每个基因沉默在ESCC1和ESCC2。基因显示统计样本数量的沉默和ESCC亚型之间的关联是显示在表(P< 0.01,确切概率法)。两个基因Bonferroni调整后仍然显著。右边的面板显示了DNA甲基化与基因表达BST2和SH3TC1。b,一个详细的分析BST2DNA甲基化在ESCC样本和非癌症控制。c,d块(c)估计白细胞分数和(d)水平的裂解caspase-7蛋白质显示值,第25和第75百分位值(单杠,底部和顶部的盒子),和最高和最低值1.5倍四分位范围(顶部和底部的胡须,分别)。
扩展数据图8 eac比其他更类似于CIN-type胃腺癌胃亚型。
一个,b,综合集群的特定于平台的集群胃食管腺癌(GEA)都使用了超星系团的方法(一个)和集群的集群作业(可口)(b)。
扩展数据图9的特定于平台的无监督聚类分析GEA-CIN肿瘤。
一个- - - - - -d,所示的热图表示基因表达(一个)、微(b),大会(c数组)和反相蛋白GEA-CIN肿瘤(列)的配置文件(d)。
权利和权限
这项工作是基于知识共享署名4.0国际4.0 (CC)许可证。本文中的图片或其他第三方材料都包含在本文的创作共用许可证,除非另有说明在信贷额度;如果材料不包括在Creative Commons许可下,用户需要获得许可执照持有人繁殖材料。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/
关于这篇文章
引用这篇文章
癌症基因组图谱的研究网络。集成化的食管的癌基因特征。自然541年,169 - 175 (2017)。https://doi.org/10.1038/nature20805
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature20805
进一步的阅读
临床验证的原产地90 -基因表达检测肿瘤组织诊断:大规模的1417名患者的多中心研究
转化医学杂志》(2022)
DEPDC1B与GABRD调节ESCC进展
癌细胞国际(2022)
HyperChIP:高变的信号跨ChIP-seq或ATAC-seq样本的识别
基因组生物学(2022)
有针对性的遗传和表观遗传分析食管腺癌和non-dysplastic巴雷特食管
临床实验胚胎学(2022)
FOXO转录活动与放化疗在EAC反应有关
转化医学杂志》(2022)