摘要
全基因组关联研究(GWAS)的一个主要挑战是确定可能的因果变异。GWAS的结果通常不会直接转化为因果变量,因为大多数命中都是非编码区域或基因间区域,而且连锁不平衡的存在导致影响在统计上分散到多个变量。后gwas注释有助于选择最可能的因果变量。有多种资源可用于gwas后注释,但这些资源可能很耗时,而且不能为数据解释提供集成的可视化辅助。因此,我们开发了fua:一个综合的基于网络的平台,使用来自多种生物资源的信息,以促进GWAS结果的功能注释,基因优先排序和交互式可视化。fua提供位置、表达量性状位点(eQTL)和染色质相互作用映射,并提供基于基因的、路径和组织富集结果。fua的结果直接有助于产生在旨在证明因果关系的功能实验中可测试的假设。
简介
在过去的十年里,超过2500个全基因组关联研究(GWAS)已经确定了数百个性状的数千个遗传位点1.在过去的3年里,GWAS的样本量出现了爆炸式的增长2,3.,4预计在未来一年甚至更长时间里,这一数字还将进一步增加到50万至50万辆5.这些功能强大的GWAS不仅将导致更可靠的结果,而且还将增加检测到的与疾病相关的遗传位点的数量。为了从这些结果中受益,将遗传位点转化为可操作的变异,从而指导功能基因组学实验和药物靶点测试是至关重要的6.然而,由于大多数GWAS命中位点位于非编码区或基因间区7从显著相关的单核苷酸多态性(SNPs)直接推断很少产生功能变异。更常见的是,GWAS击中跨越一个基因组区域(“GWAS风险位点”),该区域以多个相关snp为特征,可能覆盖多个紧密定位的基因。其中一些基因可能与疾病相关,而另一些则没有,但由于紧密定位的遗传变异的相关性质,根据关联往往无法区分相关和非相关基因P单独的值。确定最可能的相关、因果基因和变异需要整合有关区域连锁不平衡(LD)模式和相关snp的功能后果(如变异的毒性)的现有信息,还需要整合它们对基因表达的影响以及它们在染色质相互作用位点上的作用。理想情况下,从不同的知识库中获得的功能推断是集成的,注释SNP效应在基因和分子途径的更广泛的背景下被解释。例如,考虑一个基因组风险位点,一个前导SNP与一种疾病的风险增加相关,以及LD中其他几十个SNP与前导SNP也显示出低相关性P-value,跨越多个基因。如果在LD中这些检测的SNP和其他(未检测但已知的)具有先导SNP的SNP都不具有功能后果(即,改变基因的表达、影响结合位点或破坏蛋白质结构),则不能指出因果基因。然而,如果已知一个或几个snp会影响该区域某一基因的功能,而不影响其他基因,那么该单一基因在功能上与疾病相关的可能性就更高。确定哪些基因以及如何受到与某一性状相关的snp的影响,对于增加我们对该性状背后的生物机制的洞察至关重要。解释snp -性状的关联需要从多个资源和存储库中添加功能信息,例如基因型-组织表达(GTEx)。8, DNA元素百科全书(编码)9,路线图表观基因组学项目10,或染色质相互作用信息11.
在实践中,从可用的知识库中提取和解释相关的生物信息并不总是直接的,而且可能很耗时而且容易出错。因此,我们开发了fua,它在功能上注释GWAS的发现,并使用来自18个生物数据库和工具的信息,对最可能的因果snp和基因进行优先排序。基因优先排序是基于位置作图、表达定量性状位点(eQTL)作图和染色质相互作用作图的组合。结果是可视化的,以促进快速洞察相关的分子功能。fua是一个在线工具http://fuma.ctglab.nl,其中用户可以自定义设置,例如只使用外显子snp进行注释,或只使用特定组织中的eqtl snp进行基于表达数据的注释。作为输入,FUMA要求GWAS汇总统计数据和输出包括多个表格和图表,其中包含大量信息,例如基因组风险位点中snp的功能,包括蛋白质改变的后果、基因表达的影响、开放染色质状态以及三维染色质相互作用。该在线工具包括交互式图形,可用于更深入地探索关联并提供帮助,例如,识别指向同一优先基因的多条证据线,或通过生物途径连接多个基因命中。
结果
fua web应用概述
fua整合了18个生物数据库和工具来处理GWAS汇总统计数据,并提供各种注释(补充表1).为了完成这一任务,fua由两个独立的过程组成,详细描述如下。
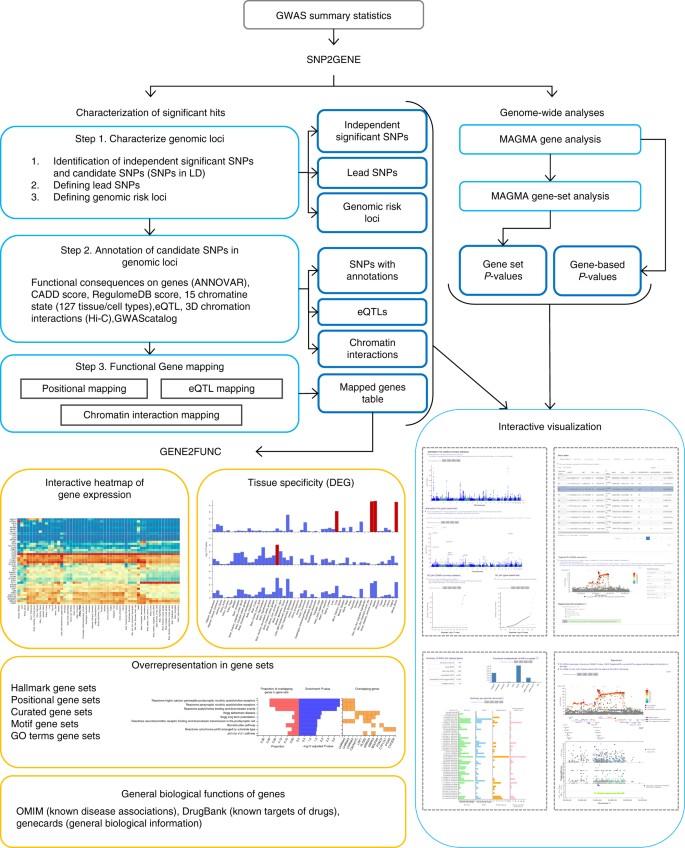
FUMA的核心功能是SNP2GENE过程。1),根据snp的位置、eQTL和染色质相互作用信息,对snp的生物功能进行标注,并将其映射到基因上。首先,根据所提供的汇总统计数据(输入格式可在补充说明中获取)1),利用FUMA根据LD结构识别独立的显著SNPs及其周围基因组位点,并确定先导SNPs和基因组风险位点(方法)。然后使用ANNOVAR注释独立显著性snp和具有独立显著性snp的LD中的snp对基因功能的功能后果(基于集成基因(build 85))12)、毒性评分(CADD评分)13),潜在的调节功能(RegulomeDB评分14和ChromHMM预测的15核染色质状态15127种组织/细胞类型9,10),利用不同组织类型的eqtl和染色质的3D结构与Hi-C数据相互作用对基因表达的影响(方法)。此外,独立的显著snp和相关snp也与GWAS目录相关联1以深入了解先前报道的风险位点中的snp与各种表型的关联。
随后,基于基因上的功能结果,通过(i)基因组上的物理位置(位置映射)、(ii) eQTL关联(eQTL映射)和(iii) 3D染色质相互作用(染色质相互作用映射)将功能注释的snp映射到基因上。基因定位可以通过设置几个参数来控制(补充表)2),允许加入或排除snp的特定功能类别(补充图。1).位置映射用于基于位于基因内的物理位置映射snp,默认使用10 kb窗口,但用户可以设置基因周围的自定义窗口。用户可以选择只使用具有特定功能后果的snp,如编码或拼接snp,以限制位置映射到功能相关的snp。因此,通过在位置定位功能中选择排除内含子snp,独立显著snp的LD中只包含内含子snp的基因将不会被fua优先考虑。eQTL定位用于将SNP映射到与之具有显著eQTL相关性的基因(即,该基因的表达与SNP的等位基因变异相关)。eQTL映射使用来自4个数据存储库(GTEx8,血液eQTL浏览器16, BIOS QTL浏览器17和BRAINEAC18),目前是基于独联体- eqtl可以将snp映射到相距1mb的基因上。用户可以选择与感兴趣的表型相关的组织/细胞类型,eqtl可以通过名义筛选P-value或由原始数据源提供的FDR(方法和补充说明2).当疾病相关区域与附近或远处基因之间存在显著的染色质相互作用时,染色质相互作用映射用于将snp映射到基因。染色质相互作用映射可能涉及远程相互作用,因为它不像eQTL映射那样有距离边界。fua目前包含Schmitt等人研究的14种组织类型和7个细胞系的Hi-C数据。11,但新的染色质相互作用数据将添加时,fua还允许用户上传自己的染色质相互作用矩阵,不限于Hi-C,但也适用于cia - pet, 5C或捕获Hi-C数据(方法和补充说明3.).由于染色质相互作用通常定义在一定的分辨率(作为基因组区域),如40kb,一个相互作用区域可能跨越多个基因。为了从染色质相互作用图谱中进一步确定候选基因的优先级,从表观基因组学项目路线图中获取组织/细胞类型特异性增强子和启动子区域的信息10可选择与相互作用的区域集成,以过滤snp和目标基因(详见方法)。
对于这三种映射策略中的每一种,基于功能注释(即CADD、RegulomeDB和15核染色质状态)的snp附加过滤是可选的(方法和补充表2).例如,设置CADD评分阈值将导致FUMA只使用高度有害的snp,或通过RegulomeDB评分或开放染色质状态过滤snp,优先考虑可能影响每个映射策略中的调控元素的snp。
三种映射策略(位置、eQTL和染色质相互作用映射)基于GWAS输入和特定的用户定义过滤器设置,产生一组优先排序的基因。eQTL和染色质相互作用定位都可能导致优先排序的基因不一定位于基因组风险位点内,尽管它们与基因组风险位点内的snp相关。有害编码SNPs的位置定位、eQTL定位和(相关)组织类型的染色质相互作用定位的组合可能揭示指向同一基因的多条证据线,并使人们能够优先考虑那些极有可能与感兴趣的性状有关的基因。
为了深入了解优先排序基因的假定生物学机制,GENE2FUNC过程在生物学背景下注释了这些基因(图。1;详情请参见方法)。具体来说,每个输入基因的生物学信息被提供,以通过绘制OMIM来洞察先前相关的疾病和药物靶点19ID和DrugBank20.ID。基于GTEx v6 RNA-seq数据的组织特异性表达模式8每个基因都被可视化为交互式热图。除单基因水平分析外,差异表达基因组(DEG;基于GTEx v6 RNA-seq数据,在53种组织类型中,每一种组织类型中表达更多(或更少)的基因集8也可用于识别优先排序基因的组织特异性(方法;补充表3.).利用超几何测试对从MsigDB中获得的基因集进行生物途径和功能类别中优先排序基因的富集测试21和WikiPathways22.重叠基因的比例,富集P-value和哪些输入基因与被测基因集重叠在图和表中可视化,这提供了优先排序基因共享的生物学功能的快速概述。
SNP2GENE和GENE2FUNC过程的结果在web应用程序上以交互表或图的形式显示。此外,表可以作为纯文本文件下载(补充说明1)和绘图可下载为多种格式(PNG、JPEG、PDF和SVG)的高质量图像。
fua涵盖了现有工具的各种功能
随着各种生物信息学工具被开发出来,以获得GWAS结果的见解23,24,25,我们比较了fua中可用的功能列表与其他工具中可用的功能列表,并在下面进一步描述这些功能(表1).
LD计算是通过计算群体特异性LD结构来表征GWAS风险位点的第一步,也就是所谓的簇化,它可以识别出独立的显著snp,并定义基因组风险位点。叮铃声26是这项任务中使用最广泛的软件,它采用GWAS汇总统计数据(需要参考面板)或基因型数据作为输入。在fua中,通过使用成对的LD (r2)的SNPs(1000个基因组计划第3阶段27),根据GWAS输入文件,得到独立的显著snp、前导snp和基因组风险位点列表。FUMA还将snp添加到已识别的风险位点中P-value(即,它们在GWAS输入文件中不可用),但它们是已识别的主要snp的LD代理,因为这些snp可能是因果相关的。或者,用户可以预先计算先导snp或风险位点,并将其上传到fua。
需要对风险位点上snp的生物学后果进行变异注释。有一些工具,如ANNOVAR12和VEP28注释了基因的功能后果,以及诸如毒性和系统发育保守等变异评分(在Hou和Zhang29).特别是对于非编码snp, SCAN30., RegulomeDB14和HaploReg31注释调控信息,如eqtl,增强子/启动子区域和转录因子结合位点(见Tak和Farnham)32广泛的概述)。虽然SCAN和HaploReg对LD是正确的,但上面提到的工具的输入是一个感兴趣的snp列表,它没有考虑到遗传关联,因此需要用户对GWAS结果进行预处理。fua对单个流中独立的重要snp的LD中的snp进行注释,不需要额外的数据预格式化。
基于基因的检测/基因集分析是一种能够在基因水平上总结SNP关联并将基因集与生物通路关联起来的方法。例如,VEGAS执行基于排列的模拟33,34, MAGMA采用多元线性回归35Pascal计算卡方统计量的和和最大值36获得基于基因的P值。此外,还有一些工具不仅可以进行基于基因的检测,还可以使用遗传关联的完整分布进行基因集分析(例如MAGMA)35,洋红色37,富裕38,描述39).FUMA对完整的GWAS输入数据进行基于MAGMA的基因分析和基因集分析。此外,在GENE2FUNC过程中,还测试了SNP2GENE或用户优先选择的基因在各种基因集中的过代表性。
可视化是允许(快速)洞察GWAS结果的基本特性之一,例如,总结snp和基因的注释信息。LocusZoom是GWAS结果中应用最广泛的可视化工具之一,它可以绘制风险位点的LD结构、基因位置以及SNP关联值40.locstrack是LocusZoom的扩展,它还可以将Chip-seq和染色质状态等附加信息绘制在一起41.3D基因组浏览器是最近开发的一个web应用程序,它包含了完整的3D染色质相互作用数据集,如Hi-C和cia - pet42,尽管它没有与GWAS汇总统计数据集成。这些工具主要关注功能相关数据源子集的可视化。fua集成了来自多线证据的结果,并提供结果的交互式可视化,促进了快速解释。
目前缺乏一个整合所有后GWAS注释资源的单一平台,这妨碍了我们对GWAS结果的理解,因为不同的GWAS研究可能使用不同的查询资源选择,导致其后GWAS解释不完整,难以进行比较。fua为各种各样的后gwas注释策略提供了一个中心位置,据我们所知,fua是这方面最通用的工具。
在体质指数GWAS中的应用
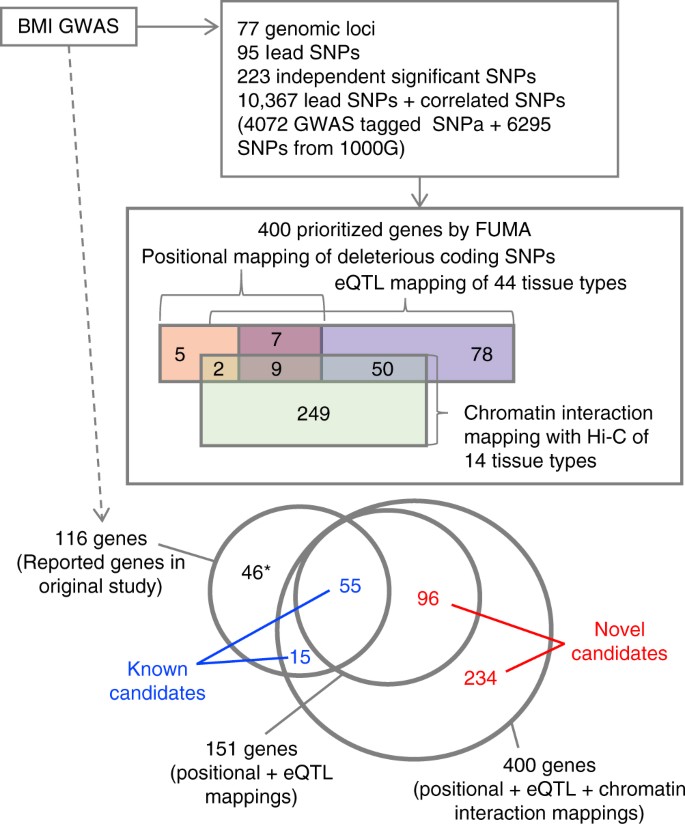
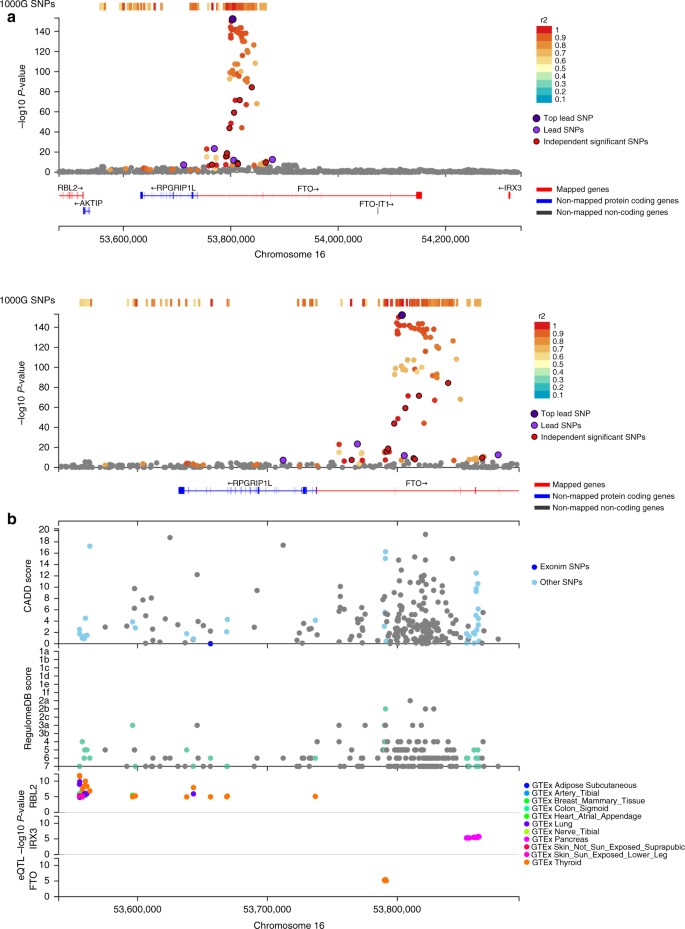
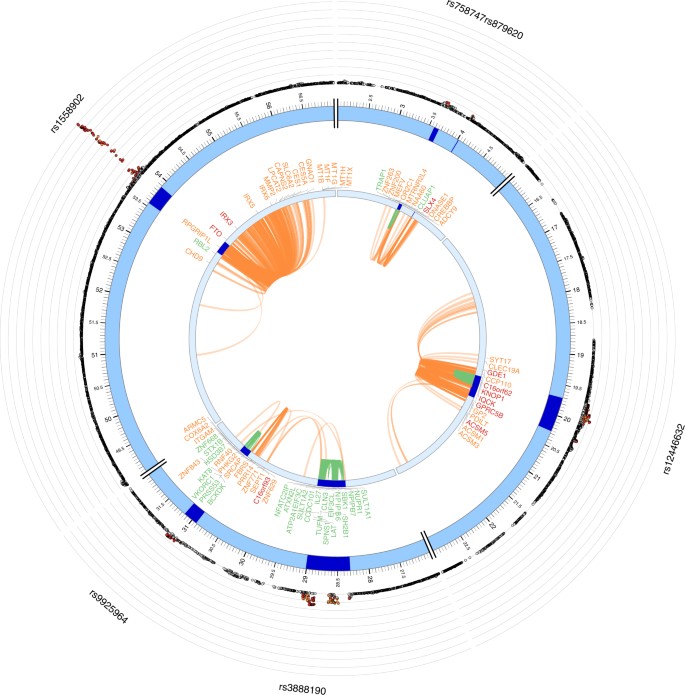
为了验证fua的实用性,我们将其应用于最新的体质指数(BMI;236231人)43.fua在77个基因组风险位点中鉴定出95个先导snp(来自223个独立的显著snp)(图。2和补充数据1- - - - - -3.),根据最初的研究。我们首先进行了有害编码SNPs的定位定位和eQTL定位(方法),对151个独特基因进行了优先排序;23个含有有害编码SNPs的基因(位置图谱),144个含有可能改变这些基因表达的eQTL基因(eQTL图谱),其中16个基因同时含有有害编码SNPs和eQTL(补充资料)4).这151个基因由55个基因组成,这些基因在最初的研究中也被报道过43FUMA涉及的96个新基因,其中45个基因位于风险位点外。这些新的候选基因与55个已知的候选基因具有相同的生物学功能,如“碳水化合物代谢”、“脂质和脂蛋白代谢”、“免疫系统”和“钙信号”(补充数据)5).此外,fua结果显示,尽管BMI的几个基因组位点包含多个优先级基因,但在包含至少一个优先级基因的43个基因座中,有22个基因是单基因优先级(补充图。2),这表明这22个基因很有可能是该区域的因果基因。22个“极有可能导致体重指数的基因”包括几个众所周知的基因,例如NEGR1,TOMM40,TMEM18.GWAS与BMI的相关性最强的信号出现在16q.12.2,其中三个基因被优先考虑;FTO,RBL2,IRX3(无花果。3.).这三个基因仅通过eQTL定位进行了优先排序,因为定位定位显示这些基因中没有定位有害编码snp。最初的研究43只提到了FTO,因为相关的SNPs位于该基因中,但没有相关的SNPs有潜在的直接影响,如编码SNPsFTO.其中两个基因被fua (RBL2而且IRX3)在物理上位于基因组位点外,当使用基于顶部SNP周围LD的传统方法优先考虑位于感兴趣位点的基因时,这些基因会被遗漏。虽然IRX3基因在最初的研究中未被报道43最近的功能研究确实证实了该基因是其表达受16q.12.2位点snp影响的因果基因44.
然后我们使用14种组织类型的Hi-C数据进行染色质相互作用映射(方法)。FUMA优先筛选了310个基因(补充数据4),其中61个基因与位置和/或eQTL映射优先排序的基因重叠,232个基因位于基因组风险位点外(图。2).通过结合三种定位策略,包括330个原始研究中没有报道的新候选基因,总共产生了400个优先排序的基因(表2)2和补充数据4).这些新的候选基因进一步支持与先前报道的已知基因共享的生物功能,如脂质和脂蛋白代谢、内稳态过程和各种代谢途径,与没有Hi-C数据的映射相比,具有更多的基因数量(补充数据5).在400个优先排序基因中,59个基因被eqtl和染色质相互作用定位,包括IRX3在16q.12.2位点上(Fig。4),这进一步支持了这些基因与BMI风险有关的假设。在48个至少包含一个定位和eQTL映射的优先级基因的位点中,染色质相互作用映射在另外18个位点中识别出候选基因(补充图。2),包括已知与BMI相关基因的位点,如中的,FOXO3,ADCY9.400个优先排序的基因在9个氧化石墨烯术语中富集,如“对锌离子的反应”和“寡肽结合”,与多个与肥胖风险相关的金属硫蛋白和谷胱甘肽s -转移酶基因重叠45,46(补充数据6).
因此,利用BMI汇总统计数据,fua确认了已知基因,但也优先考虑了新基因,包括位于BMI GWAS风险位点之外的潜在因果基因,这些基因在原始研究中被遗漏了。
GWAS在克罗恩病中的应用
为了进一步说明其实用性,我们将fua应用于克罗恩病的汇总统计47(CD;6333例,15,056对照)。利用fua,从71个基因组位点的184个独立的显著snp中筛选出95个前导snp(补充图)。3.和补充数据7- - - - - -10).首先,为了描述有害编码SNPs的位置定位和eQTL定位结果,fua对来自32个位点的95个独特基因进行了优先排序(补充图。4),其中39个基因与有害编码SNPs有关,69个基因与影响这些基因表达的eqtl有关(12个基因同时含有有害编码SNPs和eqtl;表格2和补充数据11).优先排序的95个基因包括37个已知的候选基因,这些基因也在最初的研究中报道过47包括众所周知的cd相关基因,比如NOD2,IL23R,SLC22A5, 58个新基因(补充图。3.;见补充说明4和补充无花果。5- - - - - -7对细节的结果)。这些新的候选基因包括18个物理上位于GWAS风险位点外的基因,这些新候选基因主要与37个已知基因共享免疫系统相关的生物学功能(补充资料12).
利用小肠和肝脏的Hi-C数据进行染色质相互作用定位优先的199个基因,其中18个基因与位置和/或eQTL定位优先的基因重叠,149个基因位于基因组风险位点外(补充数据11).结果共有276个优先基因,其中包括215个原始研究中没有报道的新候选基因2和补充图。3.).在通过位置和eQTL映射映射到至少一个基因的23个位点中,另外23个位点通过染色质相互作用映射映射到候选基因,其中从这些位点排序的几个基因参与了免疫系统和细胞因子信号通路(补充图。4和补充数据12).在这23个风险位点中,17q12位点被肝脏中的Hi-C定位到6个趋化因子配体:CCL1,CCL2,CCL7,CCL8,CCL11,CCL13.此外,优先基因包括11种细胞因子(IL4,IL5,IL10,IL19,IL23R,IL24,IL27,IL33,IL1RL1,IL18R1,IL18RAP)在IL18R1而且IL18RAP在全血和IL23R而且IL27这些趋化因子和细胞因子在炎性疾病中的作用已经得到了广泛的研究48然而,染色质相互作用映射从风险位点中识别出额外的相关候选基因。优先排序的276个基因在123个典型通路中富集,如免疫系统和细胞因子相关通路,这些通路已知与乳糜泻高度相关49(补充数据13).
GWAS在精神分裂症中的应用
我们还将FUMA应用于最近的精神分裂症(SCZ;36,989例,113,075例对照)GWAS汇总统计3.从109个基因组位点的269个独立显著snp中鉴定出128个前导snp(补充说明5补充图。8和补充数据14- - - - - -17).有害编码SNPs定位定位和eQTL定位确定了84个独特基因,其中36个基因与有害编码SNPs相关,65个基因与影响这些基因表达的eQTL相关(6个基因同时含有有害编码SNPs和eQTL;补充数据18).优先排序的84个基因包括65个基因,它们在最初的研究中被报道为候选基因3.19个基因是新基因2)包括11个物理上位于GWAS风险位点之外的基因。这19个新的候选基因与65个已知基因具有一些共同的生物学功能,如“母体体”和“神经元系统”(补充资料)19).在84个优先基因中,有60个也被最近的TWAS鉴定50和高c51研究包括10个位于风险位点外的基因。优先排序的基因覆盖了109个基因组位点中的34个,其中20个位点被定位到单个优先排序基因(补充图。9;见补充说明5和补充图。10详细的结果)。这20个基因极有可能驱动基因组位点中的关联信号。这些基因包括CACNA1C,LRP1恰巧,PLCB2,GRIN2A,NMUR2它们与阿尔茨海默病、长期增强、钙信号和化学突触间传递等通路有关。
利用海马和前额叶皮层的Hi-C数据进行染色质相互作用定位,确定其中33个基因的优先级DPYD而且WBPIL也被一种有害的编码SNP映射,而VPS45而且PITPNM2也被大脑中的eQTL映射(补充数据18).在这33个基因中,有15个位于基因组风险位点之外。加上定位和eQTL定位,总共产生了113个候选基因,其中包括35个原始研究中没有报道的新候选基因(表5)2和补充图。8).只有染色质相互作用优先的29个基因与其他基因具有相同的功能,如“调节应激反应”(RWDD3)、“细胞内信号传递”(SGSM3),以及调控转录的几个功能(OTUD7B而且ZBTB18;补充数据19).
在一些脑系统相关的通路中发现了富集,如尼古丁乙酰胆碱受体(nAChR),长期增强和神经递质受体结合(补充资料)20.).nAChR是一种重要的神经元受体,其亚基之一α -7 (CHRNA7)最近被研究为一种新的精神分裂症药物靶点52,53.在最近的人类大脑皮层Hi-C研究中,nAChR也被确认为富集通路51提示nAChR通路可能参与SCZ风险。
讨论
我们介绍了一个名为fua的web应用程序,它允许处理GWAS汇总统计数据,并注释、优先级snp和基因,并通过提供交互式可视化方便解释。fua提供了一个建立在最流行的gwas后注释工具之上的单一平台,包括丰富的数据存储库集合,以深入了解感兴趣的表型,在fua中注释通常只需±30分钟。对于每个优先级基因,fua提供了精确定位该基因的基本原理,例如当优先级基因的表达被与感兴趣的疾病相关的SNP改变时。相互作用的区域图。3.和补充无花果。5- - - - - -7,10)显示了基因组风险位点上哪些基因具有优先级,哪些基因没有,并且在优先级基因中注释的snp有助于功能验证实验的假设生成。例如,如果一个基因由于相关的功能缺失SNP而被优先考虑,关注该基因敲除的后续验证实验可能会提供疾病相关的功能信息。另一方面,如果一个基因被优先考虑是因为一个SNP的风险相关等位基因增加了该基因在大脑中的表达,那么在神经元细胞培养中对该基因进行过表达实验将是一个更相关的实验。
可用于解释GWAS结果的生物资源(如Hi-C和ChIA-PET)的可用性最近显著增加,一些研究通过整合其结果(例如染色质相互作用)从GWAS风险位点中确定了新的候选基因51,54,55,56,57.这些技术有潜力识别启动子和增强子的远端相互作用。特别是对于由于基因甜点的存在而难以识别靶基因的风险位点,远端相互作用可能指向因果基因。事实上,我们通过对三个GWAS研究(BMI、CD和SCZ)的结果进行染色质相互作用映射,确定了其他假定的因果基因,基于染色质相互作用信息额外确定的基因大多位于风险位点之外,并显示与已知的候选基因具有相同的功能。尽管染色质相互作用是高度组织/细胞类型特异性的,并且依赖于时间,而且目前可用的数据在这些方面仍然有限,fua提供了上传自定义相互作用矩阵的选项。此外,FUMA的构建方式使新发布的数据,包括3D染色质相互作用,eqtl和其他变体注释可以很容易地包含在SNP2GENE过程中。这使得fua成为一个灵活的网络工具,不仅可以用于新的GWAS结果,还可以用于先前发表的GWAS,用最新的生物数据源重新注释风险位点。
总之,fua提供了一个易于使用的工具,功能注释、可视化和解释遗传关联研究的结果,并快速洞察重要遗传关联的方向性生物学含义。fua将最先进的生物数据源信息合并在一个平台中,以促进功能后续分析的假设生成,旨在证明遗传变异和疾病之间的因果关系。
方法
数据预处理
本研究中使用的所有遗传数据集都基于hg19人类组合,如有必要,rsid被映射到dbSNP build 146。为了计算次要等位基因频率和LD结构,我们使用了1000基因组计划的数据27第三阶段(1000克)。次要等位基因频率和r2成对snp的最小值r2= 0.05,一对snp之间的最大距离为1 Mb)使用PLINK预先计算26对于每个可用人群(AFR、AMR、EAS、EUR和SAS)。snp的功能注释来自以下三个知识库;CADD13, RegulomeDB14和染色质核心15态模型9,10,15.Cis-eQTL信息从以下四个不同的数据库中获得;GTEx portal v68,血液eQTL浏览器16, BIOS QTL浏览器17, BRAINEAC18,如有必要,将基因映射到集成基因ID(补充说明2).从GSE87112中获得14种组织类型和7种细胞系的预处理Hi-C数据11(补充注3.).111个表观基因组的预测增强子和启动子区域从表观基因组学项目路线图中获得10.GWAS目录的基因组坐标1使用liftOver软件从hg38降至hg19。来自GTEx门户v6的归一化基因表达数据(RPKM, Read Per Kilobase Per Million)853种不同的组织被用于不同的目的。详细信息请参见“GTEx基因表达数据集”章节。来自MsigDB v5.2的策展途径和基因集21和WikiPathways22它们被分配了entrez ID。
基于GWAS的基因组风险位点的表征
为了根据提供的GWAS汇总统计数据确定该性状感兴趣的基因组位点,使用了基于相关参考群体1000G (BMI、CD和SCZ为EUR)预计算的LD结构。首先,具有全基因组显著性的独立显著性snpP-value (< 5e-8)和彼此独立于r2< 0.6被识别。对于每个独立的显著SNP,所有已知的(即,无论是否在GWAS输入中可用)SNP具有r2≥0.6且其中一个独立的显著snp被纳入进一步注释(候选snp)。因此,这些SNP可能包括GWAS输入中没有的SNP,但在1000G参考面板中有,并且在LD中具有独立的显著SNP。可根据用户自定义的次要等位基因频率(MAF,默认≥0.01)筛选候选snp。
根据已确定的独立显著性snp,如果它们在点相互独立,则定义独立的前导snpr2< 0.1。此外,如果独立的显著snp的LD块彼此距离很近(根据每个LD块的最左右snp值小于250 kb),它们将合并成一个基因组位点。因此,每个基因组位点可以包含多个独立的显著snp和前导snp。
除了使用FUMA根据GWAS汇总统计数据确定领先snp外,用户还可以提供预先定义的领先snp列表。此外,用户可以提供预定义的基因组区域列表,以限制由fua执行的所有注释到这些区域。
基因组风险位点中候选snp的注释
snp在基因上的功能结果通过ANNOVAR得到12(“基于基因的注释”)使用ensemble基因(构建85)。注意,snp可以被注释到多个基因,如果基因间snp被注释到两个最近的上游和下游基因。CADD评分、RegulomeDB评分和15核染色质状态通过匹配染色体、位置、参考和替代等位基因注释到1000G第3期的所有snp。还可以通过匹配所有独立显著性snp的染色体、位置和等位基因来提取eqtl,以及对用户选择的每种组织类型具有一个独立显著性snp的LD中的snp,其中对于不同的基因和组织类型,snp可以有多个eqtl(补充说明2).通过匹配染色体和位置,还可以检索GWAS目录中报告的先前已知的snp -性状关联的信息。
基因图谱
基因注释基于ensemble基因(build 85)。为了匹配外部基因ID, ENSG ID被映射到entrez ID,产生35808个基因,其中包括19,436个蛋白质编码基因,9249个非编码RNA,以及其他7123个基因(如假基因、加工转录物、免疫球蛋白基因和t细胞受体基因)。
位置映射是基于ANNOVAR获得的注释执行的12.提供了两个可选的过滤器来控制snp到基因的最大距离,并选择snp在基因上的特定功能结果。当定义前一种选项时,FUMA根据ANNOVAR注释将snp映射到基因,并对基因间snp应用用户定义的最大距离。当提供后一种选项时,fua只映射有ANNOVAR注释的选择注释的snp(例如,编码或拼接snp)。
eQTL映射是将所有独立的显著snp及其LD中的snp都映射到用户定义的组织类型中的eQTL。默认情况下,只使用显著的snp基因对(错误发现率(FDR)≤0.05)。此外,还可以根据用户定义的参数筛选eqtlP价值。eQTL映射将snp映射到相距1mb的基因上(独联体-eQTLs)。
染色质相互作用映射是通过将独立的显著性snp和LD中的snp与用户选择的组织/细胞类型中的显著性相互作用区域的一端重叠来完成的。然后,这些snp被定位到启动子区域(默认情况下转录起始位点上游250 bp和下游500 bp)与重要相互作用的另一端重叠的基因上。选择性地,可以筛选那些与用户选择的表观基因组的预测增强子区域重叠的snp。类似地,也可以过滤映射基因的启动子区域与用户选择的表观基因组的预测启动子区域重叠。
根据SNP2GENE第2步得到的功能注释(即CADD评分、RegulomeDB评分、15核染色质状态)可选筛选snp,分别对位置、eQTL和染色质相互作用映射进行筛选。当这些过滤器中的任何一个被激活时,候选snp就会被筛选到基因定位的初级阶段。注意,这种基于特定映射的功能注释的snp过滤不影响其他映射,例如,当snp在位置映射中按CADD评分过滤而在eQTL映射中不过滤时,snp在位置映射之前被过滤,但eQTL映射使用原始的候选snp集。
对于已映射的基因,标注了对功能突变不耐受的两个分数;功能丧失不耐受概率(pLI)58和非编码残差不耐受评分(ncRVIS)59.
用于基因分析和基因集分析
fua使用输入的GWAS汇总统计数据进行基于基因的计算P-值(基因分析)和基因集P-value(基因集分析)使用MAGMA35工具。对于基因分析,以基因为基础P的值,如果snp位于基因内,则通过将snp映射到基因计算蛋白质编码基因。对于基因集分析,基因集P-value使用基于基因的P-value为4728个策划基因集(包括规范途径)和6166个GO术语从MsigDB v5.2中获得。对于这两种分析,都使用默认的MAGMA设置(用于基因分析的SNP-wise模型和用于基因集分析的竞争模型),并使用Bonferroni校正(基因)或FDR(基因集)来校正多重测试。1000克三期27作为一个参考面板来计算snp和基因之间的LD。
GTEx基因表达数据集
从GTEx中获得53种组织类型的归一化基因表达(每千碱基每百万的读数,RPKM)(补充表)3.).GTEx中共有56,320个可用基因,我们在至少一种组织类型中按每个组织的平均RPKM大于或等于1进行过滤。结果得到28520个基因的转录本,其中22146个基因被映射到entrez ID上(详见“基因图谱”部分)。在GENE2FUNC中,优先排序基因的热图显示两个表达值;(i)每个基因的每个组织的平均log2(RPKM+1),其中RPKM winsorized为50,以便比较跨基因和组织类型的表达水平;(ii)每个基因的每个组织的归一化表达的平均值(log2(RPKM+1)的零平均值),以便比较基因内跨组织类型的表达水平。
获得差异表达的基因集(DEG;对于53种组织类型中的每一种,使用归一化表达(log2的零均值(RPKM+1))。双面的学生的t-测试是针对每个基因每个组织对所有其他组织进行的。经过Bonferroni校正后,基因配合校正P-值< 0.05和绝对对数倍变化≥0.58定义为给定组织中的DEG集,即这些基因在给定组织中的表达与所有其他组织中的表达差异最大。此外,我们通过标记来区分特定组织中基因的上调和下调t分考虑。在GENE2FUNC中,通过超几何测试对基因与这些DEG集进行测试,以评估优先排序的基因(或感兴趣的基因列表)在特定组织类型的DEG集中是否过度代表。
基因集富集试验
为了测试生物功能的过度表现,使用超几何测试,将优先排序的基因(或感兴趣的基因列表)与从MsigDB(即,标记基因集、位置基因集、策划基因集、motif基因集、计算基因集、GO基因集、致癌基因特征和免疫特征)和WikiPathways获得的基因集进行测试。背景基因集(即,优先排序基因集所针对的基因)是19,283个蛋白质编码基因。背景基因也可以从基因类型中选择,如“基因定位”部分所述。用户还可以提供自定义的背景基因集。对被测基因集的每个数据源(例如,规范途径、GO生物过程、标记基因)执行多重测试校正(即默认的Benjamini-Hochberg)。FUMA报告调整后的基因集P-value≤0.05,默认与基因集> 1重叠的基因数。
应用于GWAS汇总统计的fua参数
在上述应用中,三种映射策略应用于GWAS汇总统计,设置如下:位置映射通过选择CADD得分≥12.37的外显子和拼接snp来执行(由Kircher等人定义)。13)以限制有害编码snp的映射。eQTL定位采用GTEx eQTL, FDR<0.05。使用Schmitt等人的Hi-C数据进行染色质相互作用映射。11相互作用被FDR<1e-6过滤。用于eqtl和染色质相互作用映射的组织类型将在下面的章节中描述三种表型中的每一种。其他未提及的参数保留为默认值(补充表)2).
在BMI GWAS中的应用
参数设置如上节所述,我们在来自GTEx的44种组织中使用eqtl。染色质相互作用作图使用GSE87112中14种组织类型(肾上腺、主动脉、膀胱、背外侧前额皮质、海马、左心室、肝、肺、卵巢、胰腺、腰大肌、右心室、小肠和脾脏)的hic数据。Indels被排除在外。rsID映射到dbSNP build 146,根据人类基因组hg19参考提取染色体和位置。只使用蛋白质编码基因对53种组织类型的DEG进行基因定位和富集,检测了Canonical Pathways和GO术语。
在CD GWAS中的应用
我们按照上述参数设置,在GTEx中与CD相关的五种组织类型中使用eqtl,即小肠、结肠乙状结肠、结肠横断面、胃和全血。利用两种组织类型的Hi-C数据进行染色质相互作用作图;肝脏和小肠,来自GSE87112。MHC区域和指数被排除在分析之外。由于输入的GWAS汇总统计数据只包含发现阶段的结果,除了如上所述识别的独立先导snp外,我们手动向富马提交了71个报告的先导snp(补充数据7).在53种组织类型中,只使用蛋白编码基因对DEG进行映射和富集,对Canonical Pathways和GO项进行了测试。
在SCZ GWAS中的应用
参数设置如上所述,eqtl在10个GTEx脑组织中进行检测。利用两个脑区的Hi-C数据进行染色质相互作用映射;海马体和前额皮质。扩展的MHC区域(25-34 Mb)、X染色体和索引被排除在本分析之外。输入的GWAS汇总统计数据基于发现阶段,并不是所有来自发现和复制阶段的联合结果的先导snp都达到全基因组显著性。为了包括所有报告的铅单核苷酸多态性,向FUMA提供了111个非不确定的铅单核苷酸多态性,并在以下位置确定了其他独立的铅单核苷酸多态性P< 5e-8(补充数据14).在53种组织类型中,只使用蛋白编码基因对DEG进行映射和富集,对Canonical Pathways和GO项进行了测试。
代码的可用性
fua web应用程序的源代码可以通过git库在https://github.com/Kyoko-wtnb/FUMA-webapp/.
数据可用性
fuuma所使用的数据和工具均可从以下链接获得(详情载于补充表)1).dbSNP编译146 rsID档案:ftp.ncbi.nlm.nih.gov / snp /生物/ human_9606_b146_grch137p13 /数据库/ organism_data / RsMergeArch.bcp.gz, 1000个基因组phase 3参考面板:ftp.1000genomes.ebi.ac.uk / vol1 / ftp /释放/ 20130502 /CADD:http://cadd.gs.washington.edu/downloadRegulomeDB:http://www.regulomedb.org/downloads, 15核染色质状态:http://egg2.wustl.edu/roadmap/data/byFileType/chromhmmSegmentations/ChmmModels/coreMarks/jointModel/final/GWAS目录:https://www.ebi.ac.uk/gwas/GTEx v6:http://www.gtexportal.org/home/, Blood eQTL浏览器:http://genenetwork.nl/bloodeqtlbrowser/、BIOS QTL浏览器:http://genenetwork.nl/biosqtlbrowser/BRAINEAC:http://www.braineac.org/嗝(GSE87112):https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE87112启动子/增强子区域:http://egg2.wustl.edu/roadmap/data/byDataType/dnase/、pLI分数:ftp.broadinstitute.org/pub/ExAC_release/release0.3.1/functional_gene_constraintncRVIS得分:http://journals.plos.org/plosgenetics/article/file?type=supplementary&id=info:doi/10.1371/journal.pgen.1005492.s011MsigDB:http://software.broadinstitute.org/gsea/msigdb/WikiPathways:http://wikipathways.org/index.php/WikiPathwaysANNOVAR:http://annovar.openbioinformatics.org/en/latest/,岩浆。https://ctg.cncr.nl/software/magma.本研究中使用的GWAS汇总统计数据来自以下几点:体重指数:http://portals.broadinstitute.org/collaboration/giant/index.php/GIANT_consortium_data_filesCD:ftp.sanger.ac.uk / pub /财团/ ibdgenetics /SCZ:http://www.med.unc.edu/pgc/results-and-downloads.
参考文献
威尔特,D.等人。NHGRI GWAS目录,snp -性状关联的策划资源。核酸Res。42D1001-D1006(2014)。
伍德,A. R.等。定义成人身高基因组和生物结构中常见变异的作用。Nat,麝猫。46, 1173 - 1186(2014)。
Ripke, S.等。108个精神分裂症相关基因位点的生物学见解。自然511, 421 - 427(2014)。
Okbay, A.等。全基因组关联研究确定了74个与教育成就相关的基因座。自然533, 539 - 542(2016)。
Sudlow, C.等。英国生物库:一种可公开获取的资源,用于查明各种各样的中老年人复杂疾病的原因。科学硕士。12, 1 - 10(2015)。
布林,G.等。将全基因组关联的发现转化为精神病学的新疗法。Nat。>。19, 1392 - 1396(2016)。
Maurano, m.t.等。调控DNA中常见疾病相关变异的系统定位。科学337, 1190 - 1195(2012)。
GTEx财团。基因型-组织表达(GTEx)先导分析:人类多组织基因调控。科学348, 648 - 660(2015)。
ENCODE项目联盟。人类基因组中DNA元素的综合百科全书。自然48957 - 74(2012)。
表观基因学财团。111个参考人类表观基因组的综合分析。自然518, 317 - 330(2015)。
施密特,a.d.等人。染色质接触图谱的纲要揭示了人类基因组的空间活跃区域。细胞的代表。17, 2042 - 2059(2016)。
王凯,李敏,李敏。高通量测序数据中遗传变异的功能注释。核酸Res。38e164(2010)。
Kircher等人。估计人类遗传变异的相对致病性的一般框架。Nat,麝猫。46, 310 - 315(2014)。
波义耳,A. P.等。利用RegulomeDB注释个人基因组的功能变异。基因组Res。22, 1790 - 1797(2012)。
厄恩斯特,J. & Kellis, M. ChromHMM:色谱状态的自动发现和表征。Nat方法。9,215 - 216 (2012)。
Westra周宏儒。et al。系统识别反式eqtl作为已知疾病关联的假定驱动因素。Nat,麝猫。45, 1238 - 1243(2013)。
热尔纳科娃,D. V.等。全血中上下文依赖性表达数量性状位点的鉴定。Nat,麝猫。49, 139 - 145(2016)。
Ramasamy, A.等。人类大脑十个区域基因表达调节的遗传变异性。Nat。>。17, 1418 - 1428(2014)。
安伯杰,J. S.,博奇尼,C. A., Schiettecatte, F.,斯科特,A. F.和哈莫什,A. OMIM.org:人类在线孟德尔遗传(OMIM),人类基因和遗传疾病的在线目录。核酸Res。43D789-D798(2015)。
Wishart, d.s.等。药物库:硅类药物发现和开发的综合资源。核酸Res。34D668-D672(2006)。
Liberzon等人。分子特征数据库(MSigDB) 3.0。生物信息学27, 1739 - 1740(2011)。
库特蒙等人。维基路径:获取路径知识的完整多样性。核酸Res。44D488-D494(2016)。
爱德华,S. L.,比斯利,J.,弗伦奇,J. D.和邓宁,M.超越GWASs:照亮从关联到功能的黑暗之路。点。j .的嗡嗡声。麝猫。93, 779 - 797(2013)。
马乔拉姆,祖拜尔,A. &努日丁,S. v Post-GWAS:下一个是哪里?更多样本,更多snp还是更多生物学?遗传(Edinb)。112, 79 - 88(2014)。
Visscher, p.m., Brown, m.a, McCarthy, m.i. & Yang, J. GWAS发现的五年。点。j .的嗡嗡声。麝猫。907-24(2012)。
Purcell, S.等。PLINK:用于全基因组关联和基于群体的连锁分析的工具集。点。j .的嗡嗡声。麝猫。81, 559 - 575(2007)。
Auton, A.等。人类基因变异的全球参考文献。自然526, 68 - 74(2015)。
McLaren, W.等人。整体变异效应预测器。基因组医学杂志。17122(2016)。
侯磊,赵浩。gwas后优先排序方法综述。前面。麝猫。4, 2009 - 2014(2013)。
Gamazon, E. R.等。SCAN: SNP和拷贝号注释。生物信息学26, 259 - 262(2010)。
沃德,L. D. &凯利斯,M. HaploReg v4:系统挖掘人类复杂性状和疾病的假定因果变异、细胞类型、调控因子和靶基因。核酸Res。44D877-D881(2016)。
Tak, Y. G. & Farnham, P. J.理解GWAS:使用表观基因组学和基因组工程来理解人类基因组非编码区域snp的功能相关性。Epigenet。染色质857(2015)。
刘建中,刘建中,等。一种用于全基因组关联研究的多功能基于基因的测试。点。j .的嗡嗡声。麝猫。87, 139 - 145(2010)。
米什拉,A.和麦格雷戈,S.维加斯2:更灵活的基因检测软件。双胞胎研究的嗡嗡声。麝猫。181 - 6(2014)。
de Leeuw, c.a, Mooij, j.m, Heskes, T. & Posthuma, D. MAGMA: GWAS数据的广义基因集分析。公共科学图书馆第一版。医学杂志。11-(2015)。
Lamparter, D., Marbach, D., Rueedi, R., Kutalik, Z. & Bergmann, S.从基于snp的汇总统计数据中快速和严格地计算基因和途径评分。公共科学图书馆第一版。医学杂志。12e1004714(2016)。
Ayellet, V. S., Groop, L., Mootha, V. K., Daly, M. J. & Altshuler, D.线粒体基因中常见的遗传变异与2型糖尿病或相关血糖特征的关联并不丰富。公共科学图书馆麝猫.6e1001058(2010)。
Lee, P. H., O 'dushlaine, C. Thomas, B.和Purcell, S. M. INRICH:全基因组关联研究的基于间隔的富集分析。生物信息学28, 1797 - 1799(2012)。
Pers, t.h.等。利用预测基因功能对全基因组关联研究的生物学解释。Commun Nat。65890(2015)。
Pruim, R. J.等。LocusZoom:全基因组关联扫描结果区域可视化。生物信息学27, 2336 - 2337(2011)。
Cuellar-Partida, G., Renteria, m.e.和MacGregor, S. locstrack: GWAS结果的综合可视化和基因组注释。源代码杂志。地中海。101(2015)。
王宇,王玉华等。3D基因组浏览器:基于web的浏览器,用于可视化3D基因组组织和远程染色质相互作用。预印在http://biorxiv.org/content/early/2017/02/27/112268(2017)。
洛克,a.e.等人。对体重指数的遗传研究为肥胖生物学提供了新的见解。自然518, 197 - 206(2015)。
克劳斯尼策等人。人体FTO肥胖变异电路与脂肪细胞褐变。心血管病。j .地中海。373, 895 - 907(2015)。
佐藤美,川上美,加多田美,森美,铃木美,肥胖与金属硫蛋白咕咕叫。制药。Biotechnol。14, 432 - 440(2013)。
Raza, S.等人。谷胱甘肽- s -转移酶(GSTM1和GSTT1)和FTO基因多态性与印度北部2型糖尿病患者的关系。Balkan J. Med. Genet。1747-54(2014)。
弗兰克,A.等。全基因组meta分析增加到71个确定的克罗恩病易感位点。Nat,麝猫。42, 1118 - 1125(2010)。
Turner, m.d., Nedjai, B, Hurst, T. & Pennington, D.细胞因子和趋化因子:在细胞信号转导的十字路口和炎症性疾病。Biochim。Biophys。学报1843, 2563 - 2582(2014)。
布拉特,H., Peppelenbosch, M. P. & Hommes, D. W.克罗恩病免疫学。安。纽约理工学院。1072, 135 - 154(2006)。
卡,. .et al。精神分裂症和染色质活性的转录组全范围关联研究产生了机制疾病的见解。预印在http://biorxiv.org/content/early/2016/08/02/067355(2016)。
Won, H.等人。染色体构象揭示了人脑发育过程中的调节关系。自然538, 523 - 527(2016)。
Martin, l.f. & Freedman, R.精神分裂症和- 7尼古丁乙酰胆碱受体。Int。启一般。78, 225 - 246(2007)。
α 7-尼古丁乙酰胆碱受体激动剂对精神分裂症的认知增强作用。为基础。启地中海。65, 245 - 261(2014)。
Mifsud, B.等。用高分辨率捕获Hi-C绘制人类细胞中的远程启动子接触。Nat,麝猫。47, 598 - 606(2015)。
马丁,P.等。捕获Hi-C揭示了新的候选基因和与相关自身免疫风险位点复杂的远程相互作用。Commun Nat。610069(2015)。
麦戈文等人。捕获Hi-C在泛自身免疫遗传易感性区域6q23中发现了一种新的致病基因IL20RA。基因组医学杂志。17212(2016)。
发起人,t.g.等人。谱系特异性基因组结构将增强子和非编码疾病变体连接到目标基因启动子。细胞167, 1369 - 1384(2016)。
Lek, M.等人。60706人蛋白质编码遗传变异分析。自然536, 285 - 291(2016)。
Petrovski, S.等。调控序列对遗传变异的不耐受预示着基因的剂量敏感性。公共科学图书馆麝猫。111 - 25(2015)。
确认
这项工作由荷兰科学研究组织(NWO VICI 453-14-005)和Ingrosyl资助。我们感谢GIANT联盟、WTCCC和PGC提供的GWAS汇总统计数据和RNA-seq和eQTL数据的GTEx门户。我们也感谢Patrick Sullivan教授对3D染色质相互作用数据的讨论。
作者信息
作者和联系
贡献
D.P.构思了这项研究。K.W.和A.v.B.开发了这个网络应用程序。K.W.进行了分析并起草了手稿。k.w.、e.t.和D.P.参与了讨论、解释结果和编辑手稿。所有作者在项目的不同阶段提供了相关的投入,并批准了最终稿件。
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
额外的信息
出版商的注意:施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的使用范围,您将需要直接从版权所有者那里获得许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于这篇文章
引用这篇文章
K. Watanabe, E. Taskesen, A. van Bochoven。et al。与FUMA基因关联的功能定位和注释。Nat Commun81826(2017)。https://doi.org/10.1038/s41467-017-01261-5
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41467-017-01261-5
这篇文章被引用
功能基因组分析描述了gwas确定的双相障碍风险变异的调节机制
基因组医学(2022)
精神分裂症与心脏代谢性状共享的先导位点探讨
BMC基因组学(2022)
额颞叶痴呆和精神疾病之间共有的基因联系
BMC医学(2022)
中东后裔的GWAS揭示了肾脏功能的位点保护——一项横断面研究
BMC医学(2022)
解释种系和体细胞非编码突变在癌症中的作用:表达和染色质构象信息分析
临床实验胚胎学(2022)