文摘
微生物群落的功能配置文件通常使用综合生成宏基因组或metatranscriptomic序列搜索,阅读耗时,容易伪造的映射,通常限于基层量化。我们开发了HUMAnN2,分层搜索策略,使快速、准确、species-resolved host-associated和环境社区的功能分析。HUMAnN2标识一个社区的已知物种,将读取pangenomes,执行翻译搜索非机密的读取,最后量化基因家族和途径。相对于纯翻译搜索,HUMAnN2更快,产生更精确的基因家族的概要文件。我们应用HUMAnN2渐变群的海洋新陈代谢的变化,研究生态贡献模式在人类微生物组途径,不同物种的基因组与转录的贡献,和应变分析。进一步介绍contributional多样性的解释模式的生态组装不同微生物群落类型。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
肥胖女性Multi-omics肠道微生物组签名:饮食和不受控制的饮食行为的作用
BMC医学开放获取2022年12月27日
番茄的遗传rhizobacteria耐Ralstonia solanacearum
微生物组开放获取2022年12月15日
抗生素暴露阻止收购有益的早产婴儿肠道微生物的代谢功能
微生物组开放获取2022年7月07
访问选项
订阅自然+
得到直接在线访问自然和其他55个自然杂志
29.99美元
每月
订阅杂志
得到完整的期刊访问1年
99.00美元
只有8.25美元的问题
所有价格是净价格。
增值税将被添加后在付款。
税计算期间将完成付款。
买条
时间有限或全文访问ReadCube。
32.00美元
所有价格是净价格。


数据可用性
人类微生物组计划(HMP)基因组分析这项工作可以通过http://hmpdacc.org。IBDMDB基因组和metatranscriptomes分析这项工作可以通过http://ibdmdb.org。红海基因组分析这项工作以前NCBI BioProject沉积PRJNA289734。中使用的合成基因组和metatranscriptomes HUMAnN2和其他方法的评价可从作者和http://huttenhower.sph.harvard.edu/humann2。
引用
Shafquat,。乔伊斯,R。,Simmons, S. L. & Huttenhower, C. Functional and phylogenetic assembly of microbial communities in the human microbiome.Microbiol趋势。22,261 - 266 (2014)。
Fuhrman, j . a .微生物群落结构及其功能的影响。自然459年,193 - 199 (2009)。
罗伊德•普莱斯,J。,Abu-Ali, G. & Huttenhower, C. The healthy human microbiome.基因组医学。8,51 (2016)。
Franzosa, e·a . et al .测序和超越:整合分子微生物群落分析的“组学”。启Microbiol Nat。13,360 - 372 (2015)。
Segata: et al .宏基因组微生物群落分析使用独特clade-specific标记基因。Nat方法。9,811 - 814 (2012)。
Sunagawa, s . et al .宏基因组物种分析使用通用系统发育标记基因。Nat方法。10,1196 - 1199 (2013)。
席尔瓦·G·G。,Green, K. T., Dutilh, B. E. & Edwards, R. A. SUPER-FOCUS: a tool for agile functional analysis of shotgun metagenomic data.生物信息学32,354 - 361 (2016)。
沙玛,a K。古普塔,。库马尔,S。,Dhakan, D. B. & Sharma, V. K. Woods: a fast and accurate functionalannotator and classifier of genomic and metagenomic sequences.基因组学106年1 - 6 (2015)。
Petrenko, P。洛布,B。,Kurtz, D. A., Neufeld, J. D. & Doxey, A. C. MetAnnotate: function-specific taxonomic profiling and comparison of metagenomes.BMC医学杂志。1392 (2015)。
玻色,T。,Haque, M. M., Reddy, C. & Mande, S. S. COGNIZER: a framework for functional annotation of metagenomic datasets.《公共科学图书馆•综合》10e0142102 (2015)。
金,J。,Kim, M. S., Koh, A. Y., Xie, Y. & Zhan, X. FMAP: functional mapping and analysis pipeline for metagenomics and metatranscriptomics studies.BMC生物信息学17420 (2016)。
Huson d·h . et al。梅根社区Edition-interactive勘探和大规模的微生物基因组测序数据的分析。公共科学图书馆第一版。医学杂志。12e1004957 (2016)。
Nayfach et al。自动、准确估计猎枪基因组的基因家族丰富。公共科学图书馆第一版。医学杂志。11e1004573 (2015)。
Abubucker, s . et al .代谢重建人类微生物宏基因组数据及其应用。公共科学图书馆第一版。医学杂志。8e1002358 (2012)。
人类微生物组项目财团。结构、功能和人类健康的微生物多样性。自然486年,207 - 214 (2012)。
Buchfink B。,Xie, C. & Huson, D. H. Fast and sensitive protein alignment using Diamond.Nat方法。1259-60 (2015)。
赵,Y。,Tang, H. & Ye, Y. RAPSearch2: a fast and memory-efficient protein similarity search tool for next-generation sequencing data.生物信息学28,125 - 126 (2012)。
Hauswedell, H。,Singer, J. & Reinert, K. Lambda: the local aligner for massive biological data.生物信息学30.i349-i355 (2014)。
Truong, d . T。邰蒂,。,Pasolli E。,Huttenhower, C. & Segata, N. Microbial strain-level population structure and genetic diversity from metagenomes.基因组Res。27,626 - 638 (2017)。
朔尔茨,m . et al . Strain-level微生物流行病学和人口基因组学从猎枪宏基因组。Nat方法。13,435 - 438 (2016)。
罗,c . et al .约束识别微生物菌株在宏基因组数据集。生物科技Nat。》。33,1045 - 1052 (2015)。
Truong, d . t . et al . MetaPhlAn2增强metagenomic分类分析。Nat方法。12,902 - 903 (2015)。
Medini D。,Donati, C., Tettelin, H., Masignani, V. & Rappuoli, R. The microbial pan-genome.咕咕叫。当今。麝猫。Dev。15,589 - 594 (2005)。
Suzek b E。王,Y。,Huang, H., McGarvey, P. B. & Wu, C. H. UniRef clusters: a comprehensive and scalable alternative for improving sequence similarity searches.生物信息学31日,926 - 932 (2015)。
加尔佩林,m . Y。,Makarova, K. S., Wolf, Y. I. & Koonin, E. V. Expanded microbial genome coverage and improved protein family annotation in the COG database.核酸Res。43D261-D269 (2015)。
Kanehisa, M。佐藤,Y。,Kawashima, M., Furumichi, M. & Tanabe, M. KEGG as a reference resource for gene and protein annotation.核酸Res。44D457-D462 (2016)。
芬恩,r . d . et al .蛋白质包含了家庭数据库:向一个更可持续的未来。核酸Res。44D279-D285 (2016)。
基因本体论财团。基因本体论财团:前进核酸Res。43D1049-D1056 (2015) . .
卡斯皮,r . et al . MetaCyc数据库和酶的代谢通路和BioCyc通路/基因组数据库的集合。核酸Res。44D471-D480 (2016)。
罗伊德•普莱斯,j . et al .菌株、函数和动态扩展人类微生物组项目。自然550年,61 - 66 (2017)。
Sczyrba, et al。关键的评估metagenome interpretation-a宏基因组软件的基准。Nat方法。14,1063 - 1071 (2017)。
Hamady, m &骑士,r .微生物群落分析人类微生物组项目:工具,技术,和挑战。基因组Res。19,1141 - 1152 (2009)。
拉威尔,j . et al .生育年龄女性的阴道微生物。Proc。国家的。学会科学。美国108年,4680 - 4687 (2011)。
汤普森,l . r . et al .宏基因组共变密集采样环境梯度在红海。ISME J。11,138 - 151,https://doi.org/10.1038/ismej.2016.99(2017)。
Sunagawa,美国等全球海洋微生物的结构和功能。科学348年,1261359 (2015)。
天鹅,b . k . et al .基因组和代谢多样性的海洋集团我Thaumarchaeota海洋中层的两个副热带环流。《公共科学图书馆•综合》9e95380 (2014)。
汤普森,l . r . et al .噬菌体辅助代谢基因和蓝藻主机重定向碳代谢。Proc。国家的。学会科学。美国108年E757-E764 (2011)。
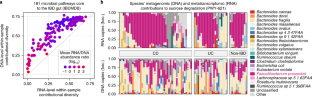
综合HMP (iHMP)研究网络联盟。综合人类微生物组项目:动态分析microbiome-host组学概要人类健康和疾病的时期。细胞宿主细菌16276 - 289 (2014). .
Franzosa, e·a·等。有关metatranscriptome和metagenome人类肠道。Proc。国家的。Acad.Sci。美国111年E2329-E2338 (2014)。
恩伯,p . j . et al .核心肠道微生物在肥胖和精益双胞胎。自然457年,480 - 484 (2009)。
伯克,C。,Steinberg, P., Rusch, D., Kjelleberg, S. & Thomas, T. Bacterial community assembly based on functional genes rather than species.Proc。国家的。学会科学。美国108年,14288 - 14293 (2011)。
Duran-Pinedo, a . e . et al .全社区的转录组受试者口腔微生物的,没有牙周炎。ISME J。8,1659 - 1672 (2014)。
梅森,o . et al, Metagenome metatranscriptome和单细胞测序揭示微生物对深水地平线石油泄漏。ISME J。6,1715 - 1727 (2012)。
Pasolli E。,Truong, d . T。马利克,F。,Waldron, L. & Segata, N. Machine learning meta-analysis of large metagenomic datasets: tools and biological insights.公共科学图书馆第一版。医学杂志。12e1004977 (2016)。
UniProt财团。UniProt:蛋白质信息的中心。核酸Res。43D204-D212 (2015)。
黄,k . et al . MetaRef: pan-genomic数据库比较微生物基因组学和社区。核酸Res。42D617-D624 (2014)。
Segata, N。,Börnigen, D., Morgan, X. C. & Huttenhower, C. PhyloPhlAn is a new method for improved phylogenetic and taxonomic placement of microbes.Commun Nat。42304 (2013)。
埃德加,r . c .搜索和集群数量级的速度比爆炸。生物信息学26,2460 - 2461 (2010)。
Langmead, b &扎尔茨贝格,s . l .快gapped-read符合领结2。Nat方法。9,357 - 359 (2012)。
你们y & Doak t . g .吝啬方法生物途径重建/推论基因组和基因组。公共科学图书馆第一版。医学杂志。5e1000465 (2009)。
黄,W。李,L。,Myers, J. R. & Marth, G. T. ART: a next-generation sequencing read simulator.生物信息学28,593 - 594 (2012)。
确认
作者感谢m . Wong t·夏普顿和HUMAnN用户组的成员的反馈HUMAnN2的开发和评估。这项工作是由NSF提供资金1565100 (J.G.C.);人计划(玛丽·居里行动)的欧盟第七框架计划(fp7/2007 - 2013)根据意图赠款协议由MIUR pcig13 - ga - 2013 - 618833和“Ricerca无缝化”RBFR13EWWI_001 (n);NIH的趋势U54DE023798, NSF mcb - 1453942, NIH NIDDK P30DK043351;和NSF dbi - 1053486 (h)。
作者信息
作者和联系
贡献
E.A.F.,L.J.M., and C.H. designed the methods. L.J.M. developed the software implementation. G.R., G.W., and N.S. produced datasets to support the software. E.A.F., L.J.M., G.R., L.R.T., M.S., and K.S.L. designed and carried out the evaluations and applications; R.K., J.G.C., and all other authors participated in interpretation of the resulting data. E.A.F., L.J.M., L.R.T., M.S., K.S.L., and C.H. wrote the paper with feedback from the other authors.
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
出版商的注意:施普林格自然保持中立在发表关于司法主权地图和所属机构。
综合补充信息
补充图1扩大HUMAnN2方法的概述。
(一个)HUMAnN2实现了一个分层元'omic搜索,旨在解释微生物群落DNA或RNA的起源之前读取基于检测到的微生物的pangenomes回落更多计算昂贵的翻译搜索。(b)读取编码的分层搜索产生比对序列已知的或模棱两可的分类法。特有的方式处理这些比对计算基因家族丰富和重建社区代谢途径。(c)HUMAnN2从而提供为每个社区元'ome:每个基因丰度,下游通路的存在/没有电话和丰度和可视化和统计测试
补充图2参考合作分析一个复杂的合成metagenome。
我们构建和分析HUMAnN2 100 -成员mock-even合成metagenome只包含非人类物种相关(~ 2×覆盖/物种)。(一个每个基因)读取采样的数量变化(与基因组的平均fold-coverage)作出了不平凡的贡献在每个物种基因丰度估计误差HUMAnN2(大约0.1 Bray-Curtis不同单位)。(b)基层基因家族丰度估计的准确性与社区的物种数量减少线性错过了HUMAnN2′年代分类预选(模拟物种在这里不包括集从底层pangenome引用集合)。(c)HUMAnN2′年代总体运行时线性增加,随着越来越多的物种被排除在分类预选(这将导致更多的工作做在翻译搜索)。运行时反映执行使用8个CPU核
补充图3 HUMAnN2分层搜索性能对人类基因组。
我们应用HUMAnN2′年代分层搜索概要397第一次访问HMP基因组在哈佛大学的奥德赛研究计算集群(8 CPU核/工作)。样本数量/身体网站如下:54前鼻孔,颊粘膜,65 68 supragingival斑块,舌背73,76凳子,34后穹窿。(一个)在大多数机构网站,~ 60%的读取被发现pangenomes解释,(b)~ 20%额外解释为下游翻译搜索(~ 80%)。Pangenome搜索性能(c)持续超过翻译搜索性能(d)1 - 2个数量级。从最小到最大,箱线图元素板模拟代表下内心的栅栏,第一四分位数、中位数,第三四分位数,上层内部栅栏。水平红线表示对所有样本中位数。(e)总运行时在很大程度上是由读取通过翻译搜索的数量,与< 1亿读)和(HMP样本的数量大约是线性输入读取(~ 1 h / 500万输入读取)。(f)峰值内存使用数量的次线性输入读取和可预测的。异常值的集群在f大样本的结果重新排队期间:这些样品晚些时候恢复HUMAnN2工作流,从而显示内存使用峰值较小
补充图4 HUMAnN2与其他方法相比(细节)。
我们异形1000万-读合成肠道metagenome使用HUMAnN2(分层和纯翻译搜索模式),HUMAnN1,兜兜转转,梅根,ShotMAP产生丰富的齿轮。在这里,预期(金标准)和观察齿轮丰度比较每百万单位的副本(cpm;这是。,raw abundance normalized by gene length and number of mapped reads). HUMAnN2′s tiered search was considerably more accurate than the other methods based on pure translated search. HUMAnN2′s pure translated search showed better agreement than other translated search methods, with its largest source of error being underreporting of low-abundance COGs (false negatives). This behavior is expected from the translated search coverage filters used in HUMAnN2, which we use to limit false positive detection events (that is., COGs with zero expected abundance and non-zero observed abundance). Ticks in the x- and y-axis margins represent zero values; x-axis ticks are false negatives and y-axis ticks are false positives
图5补充蛋白质翻译搜索报道阈值。
如果两个很大程度上不相关的蛋白质共享本地序列同源性,读取来自同源区域将映射到蛋白质,可能导致假阳性检测事件。限制这类事件,我们需要一个阈值在招募读取蛋白质分数的网站翻译搜索之前考虑蛋白质“发现”。我们评估潜在的阈值通过分析纯翻译搜索的结果的合成基因组与UniRef90数据库。权衡灵敏度和精度显示100名成员,甚至non-human-associated metagenome,和20名,交错,human-gut-associated metagenome b。当所有社区基因组覆盖,50%的覆盖率阈值(HUMAnN2′年代默认)收益率显著增加精度只有轻微损失的敏感性(一个)。损失灵敏度更高的阈值,当罕见(信号低)包括基因组、基因在基因组信号低往往无法满足覆盖阈值由于读取采样(不足b)。这些评估并不反映任何额外的翻译(例如搜索结果的后处理。权重通过对齐质量)提供额外的精度的改进
补充图6 HUMAnN2相比其他方法:合成metatranscriptome评估。
我们异形1000万-读合成肠道metatranscriptome使用HUMAnN2(分层和纯翻译搜索模式),HUMAnN1,兜兜转转,梅根,ShotMAP社区范围内齿轮的生产资料转录丰度。20个物种的基因丰度值几何交错(如肠道metagenome评价),而基因(转录)采样物种内部遵循对数正态分布分布[ln N (0, 1)]。(一个)测量方法的精度和性能评估。所有的方法都可以使用8个CPU核和30 GB的内存。这个面板是类似于图。1 e(重点是宏基因组齿轮丰度相同的合成社区)。(b)观察与预期齿轮转录丰度在六个方法。这个面板类似于补充图。4。CPM指“每百万副本。“蜱虫在x轴和y轴利润率代表零值;轴蜱虫假阴性和轴蜱虫是假阳性
补充图7 HUMAnN2相比其他方法:小说隔离已知的物种,UniRef90-based齿轮金本位制。
我们使用的异形1000万-读合成metagenome HUMAnN2(分层和纯翻译搜索模式),HUMAnN1,兜兜转转,梅根,ShotMAP丰富社区范围内齿轮的生产资料。最近二十,新的隔离(即已知的物种。,species present in HUMAnN2′s pangenome database) were sampled at staggered relative abundance. (一个)测量方法的精度和性能评估。所有的方法都可以使用8个CPU核和30 GB的内存。这个面板和分析类似于那些在无花果。1 e。(b)观察与预期齿轮转录丰度在六个方法。这个面板类似于补充图。4。CPM指“每百万副本。“蜱虫在x轴和y轴利润率代表零值;轴蜱虫假阴性和轴蜱虫是假阳性
补充图8 HUMAnN2相比其他方法:小说隔离已知的物种,UniRef50-based齿轮金本位制。
这个数字反映补充图。6,除了齿轮注释定义基于co-clustering UniRef50家庭(而不是UniRef90)。同样,HUMAnN2在UniRef50模式下运行。这些变化倾向于敏感性在特异性在隔离基因组注释和分析。(一个)准确性和六个功能的性能分析方法。(b)观察齿轮丰度和预期
补充图9 HUMAnN2相比其他方法:隔离的新物种,UniRef90-based齿轮金本位制。
我们使用的异形1000万-读合成metagenome HUMAnN2(分层和纯翻译搜索模式),HUMAnN1,兜兜转转,梅根,ShotMAP丰富社区范围内齿轮的生产资料。20最近,新的小说种类(即隔离。,species not present in HUMAnN2′s pangenome database) were sampled at staggered relative abundance. Note that, in this context, HUMAnN2′s tiered search relies entirely on the translated search phase to explain sample reads. (一个)测量方法的精度和性能评估。所有的方法都可以使用8个CPU核和30 GB的内存。这个面板和分析类似于那些在无花果。1 e。(b)观察与预期齿轮转录丰度在六个方法。这个面板类似于补充图。4。CPM指“每百万副本。“蜱虫在x轴和y轴利润率代表零值;轴蜱虫假阴性和轴蜱虫是假阳性。“预期齿轮丰富”的纵条纹结果单副本齿轮,只分配给一个基因组(因此都有相同的预期范围)
补充图10 HUMAnN2相比其他方法:隔离的新物种,UniRef50-based齿轮金本位制。
这个数字反映补充图。8,除了齿轮注释定义基于co-clustering UniRef50家庭(而不是UniRef90)。同样,HUMAnN2在UniRef50模式下运行。这些变化倾向于敏感性在特异性在隔离基因组注释和分析。(一个)准确性和六个功能的性能分析方法。(b)观察齿轮丰度和预期
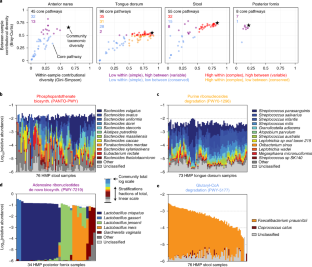
补充图11 Contributional多样性在额外的口语网站。
这个图是图的格式。2主要文本,包括数据的两个额外的口腔机构网站:颊粘膜和supragingival斑块。恒星显示背景了解社区的多样性
额外补充图12的例子试较低的核心人类微生物组通路和低主客体之间contributional多样性。
栏的高度代表总相对丰度的途径和对数。个别物种的贡献/其他/非保密是线性缩放在总杆高度
补充图13非阴道人类微生物组的例子与简单的途径,但不同contributional多样性。
栏的高度代表总相对丰度的途径和对数。个别物种的贡献/其他/非保密是线性缩放在总杆高度
补充图14的例子subspecies-level功能变异(基因水平)。
(一个)的乳酸菌jensenii充分体现在21 HMP后穹窿样本。似乎至少两个subspecies-level演化支,定义的基因块a1和a2(强调)。(b)的真细菌eligens充分体现在51 HMP粪便样本。似乎至少有三个subspecies-level演化支,定义的存在/缺乏基因块b1、b2和b3(强调)
补充图15的潜在niche-adapted亚种的例子嗜血杆菌haemolyticus。
宏基因组的菌株(UniRef90基因家族的存在/缺失资料)三个口语网站这个物种不同的发现。右侧情节说明coreness、可变性和单个基因位点专一的浓缩。变化的峰值为1.0 50%的基因检测的样本。特定场地浓缩基因时峰值为1.0焦网站普遍100%和0%普遍在所有其他网站(1对应完全相反的场景)
权利和权限
关于这篇文章
引用这篇文章
Franzosa,电子艺界McIver,剩下,Rahnavard, G.et al。了解基因组的功能分析和metatranscriptomes。Nat方法15,962 - 968 (2018)。https://doi.org/10.1038/s41592 - 018 - 0176 - y
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41592 - 018 - 0176 - y
本文引用的
大量重叠症状和无症状的泌尿生殖器的微生物群
微生物组(2022)
SeqScreen:准确、敏感功能筛选致病序列通过整体学习
基因组生物学(2022)
饮食接触杀虫剂对粪便微生物代谢的影响在成年双胞胎
环境卫生(2022)
抗生素暴露阻止收购有益的早产婴儿肠道微生物的代谢功能
微生物组(2022)
肥胖女性Multi-omics肠道微生物组签名:饮食和不受控制的饮食行为的作用
BMC医学(2022)
