摘要
背景
大多数肠道微生物组研究都是使用粪便样本进行的。然而,小肠对消化、营养吸收和免疫功能至关重要,对其微生物种群进行表征对于阐明其在人类健康和疾病中的作用至关重要。
目标
描述不同小肠节段的微生物群,并将其与粪便微生物群进行对比。
方法
前瞻性地招募接受食管胃十二指肠镜检查而无结肠准备的男性和女性受试者。从十二指肠、空肠和最远的距离获得腔内抽吸。一个子集也提供了粪便样本。使用CLC Genomics Workbench进行16S rRNA测序和分析。
结果
16S rRNA测序确定了小肠和粪便微生物群之间2000多个操作分类单元的差异。小肠中厚壁菌门和变形菌门最为丰富,拟杆菌门相对较少。在小肠中,厚壁菌门主要以乳酸菌为代表,包括链球菌科、乳酸菌科和肉杆菌科,变形菌门以奈瑟菌科、巴氏杆菌科和肠杆菌科为代表。十二指肠和FD的微生物特征明显不同,但十二指肠和空肠、空肠和FD的微生物特征存在重叠。粪便中厚壁菌门以Ruminococcaceae、Lachnospiraceae、Christensenellaceae为代表,变形菌门以delta变形菌门纲为代表。
结论
小肠的微生物群明显不同于粪便中的微生物群,在不同的节段之间也不同。这些发现可能对确定小肠菌群的组成变化如何影响人类疾病状态具有重要意义。
简介
人体微生物组计划[1是对了解人体微生物群的突破性努力。对人体几个重要部位的微生物种群进行了分析,包括口腔、鼻子、皮肤、阴道和肠道[2].由于容易获得粪便样本,肠道微生物组以粪便作为替代品进行了专门的表征。然而,尽管粪便在分析远端结肠的微生物种群方面是有用的,但它并不能充分代表整个胃肠道[3.].酸度、转运时间和微生物数量等条件沿肠道长度变化巨大,这些差异对存在的微生物种群有显著影响[3.].
这项利用粪便进行的初步研究为人类肠道微生物群的评价提供了一些参数。首先,拟杆菌门和厚壁菌门是粪便微生物群中的两个主要门[4,5],这两个门的比例和组成可能对肥胖和其他疾病很重要[6,7].第二个重要参数是微生物多样性,这已被发现是人类健康的一个重要决定因素。许多肠道疾病,如炎症性肠病,都与粪便样本中微生物多样性减少有关[8,9].
与结肠相反,小肠被分为十二指肠、空肠和回肠,对消化和营养吸收以及免疫功能至关重要[10].其中,十二指肠可能是最重要的,因为它是来自胃的食糜、来自胰腺的酶和来自肝胆系统的胆盐的主要汇集点。认识到描述小肠微生物群及其在人类健康和疾病中的作用的重要性,我们设计了REIMAGINE(揭示整个肠道微生物群及其与遗传、免疫和神经内分泌生态系统的关联)研究[11].小肠微生物组研究的一个关键部分是开发获取和分析小肠样本的技术。小肠是一个微生物量较低的环境,而粪便的微生物量较高[3.,12],传统上用于抽吸液体的开放式抽吸导管与口腔污染的高风险相关,这对微生物组分析非常有问题[13,14].小肠内的液体量也是有限的,特别是当患者处于禁食状态时,它通常是粘性的,使基质中的微生物难以接近。因此,作为REIMAGINE研究的一部分,我们优化了减少来自口腔和胃等其他部位的交叉污染的技术,开发了从粘性粘液中释放细菌的方法,并改进了低生物量液体的技术[15].在这项研究中,我们使用这些经过验证的技术来表征小肠不同部位的微生物群,并将其与粪便中的微生物群进行对比。
方法
研究对象
REIMAGINE研究是一项大型研究,旨在检查人类健康与疾病中小肠微生物群之间的关系[15].年龄在18-85岁、接受上消化道内镜[食管胃十二指肠镜(EGD)]或无结肠镜的顺行双气囊肠镜(DBE)的连续男性和女性患者都有资格参与。在完成全面的健康信息问卷后,收集小肠抽吸物[15].可选择的粪便样本也可从愿意提供的受试者那里获得。该研究方案得到了Cedars-Sinai医疗中心机构审查委员会的批准,所有受试者在参与研究前都提供了知情的书面同意。
研究过程
调查问卷
在EGD/DBE之前,所有受试者都完成了一份研究问卷,其中记录了他们的人口统计、医疗和家族史,包括药物使用、酒精和消毒史、旅行史、饮食习惯和变化。通过对所有受试者的医疗记录审计,验证了参与者提供的医疗信息。所有患者数据在分析前都被去识别。虽然使用抗生素的受试者被包括在REIMAGINE研究中,但在内镜检查前6个月内使用抗生素的受试者样本被排除在本研究之外。
小肠样本收集
在EGD过程中,如前所述,使用定制的无菌吸入导管(Hobbs Medical, Inc.)从十二指肠的第二部分获得腔内液体样本(最多2ml) [15].在DBE过程中,从十二指肠、空肠和过程中达到的最远距离获得腔内液体样本(最多2ml)。再次,使用定制的无菌导管从每个节段收集液体,以减少口腔和胃酸交叉污染的风险。
粪便样本集合
同意提供粪便样本用于小肠和粪便微生物群对比分析的受试者,在EGD或DBE程序后给予收集包。根据制造商的说明,使用OMNIgene-GUT微生物组试剂盒(DNA Genotek,渥太华,ON,加拿大)收集粪便样本。受试者在术后1-2天在家中收集粪便样本并邮寄至实验室。样品保存在25°C,然后冷冻并转移到−80°C,直到分析。
送气音的处理
抽吸后,立即使用一种经过验证的新方案对样本进行处理,该方案优化了小肠抽吸物中的微生物分离[15].简单地说,样品用无菌1 ×二硫苏糖醇(EMD Millipore Corp., Darmstadt, Germany)按1:1的比例处理,漩涡30 s。然后在高速(~ 14000 RPM)下离心10分钟,小心地取出上清,保存在−80°C下进行非靶向代谢组学分析。在每个颗粒中加入500微升无菌Allprotect试剂(Qiagen, Hilden, Germany)以稳定DNA、RNA和蛋白质,并在- 80°C下保存颗粒,然后进行DNA分离和小肠微生物组分析。
DNA提取与定量
对于小肠吸出样本,保存在Allprotect试剂下的微生物球在冰上解冻,无菌1 × DTT按1:1的比例加入,如前所述使Allprotect液化。[15然后进行DNA提取,如前所述,[15使用magattraction PowerSoil DNA KF试剂盒(Qiagen, cat。27000 - 4 - kf)。对于粪便样本,OMNIgene-GUT管被大力旋转10 s。250 μ L的样本被转移到无菌的2-mL管中,在那里进行裂解步骤。然后使用magattraction PowerSoil DNA KF Kit进行DNA提取。使用Qubit dsDNA BR检测试剂盒或quantum - it™PicoGreen对dna进行量化®在量子比特4荧光计(Invitrogen)上进行dsDNA检测(Thermo Fisher Scientific, Waltham, MA, USA)。
文库制备和16S rRNA测序
根据Illumina (Illumina, San Diego, CA, USA)协议,对小肠吸出物和粪便中的dna进行16S文库准备https://support.illumina.com/documents/documentation/chemistry_documentation/16s/16s-metagenomic-library-prep-guide-15044223-b.pdf如前所述[15],使用由Klindworth等人发表并验证的基因特异性引物s - d - act-0341-b- s -17和s - d - act-0785-a- a- 21。[16放大V3和V4区域。最终的文库在Qubit 4荧光计上使用Qubit 1X dsDNA HS检测试剂盒进行量化,并在Agilent 2100生物分析仪系统上使用Agilent DNA 1000芯片(Agilent Technologies, Santa Clara, CA)进行分析。
16S宏基因组测序与分析
如前所述,在MiSeq系统(Illumina, San Diego, California)上对15 - 20 pM的池库进行配对测序[15].操作分类单元(OTU)聚类和分类分析使用CLC Genomics Workbench v. 10.1.1和CLC Microbial Genomics Module v. 2.5 (Qiagen)进行。利用基于amplicon的OTU聚类工具对序列进行修剪合并,在97%序列相似性水平上聚类为OTU。选择最丰富的序列作为每个簇的代表,然后使用CLC微生物基因组学默认值和Greengenes数据库2013年版本分配到一个分类级别。低深度样本(每个样本少于9000个序列)从分析中删除。利用丰度分析工具计算Alpha多样性指数。加权Unifrac度量用于计算样本间多样性(beta多样性)。
统计分析
根据McMurdie和Holmes的建议,对每个小肠节段和粪便中显著差异的OTUs进行了预测[17,并从Weiss等人。[18],当每组的平均库大小近似相等和/或组间的折叠差异不高(平均为> 2-3x)时使用。
如前所述,使用CLC基因组学工作台v. 10.1.1和CLC微生物基因组学模块v. 2.5 (Qiagen)进行多重比较和统计分析[15].使用负二项GLM模型来获得两个条件之间OTU对数倍变化的最大似然估计,并使用Wald检验来确定显著性。错误发现率(FDR)进行纠正P值。分别使用GraphPad Prism 7.02 (GraphPad Software, La Jolla, CA, USA)和MetaboAnalyst(夏实验室@ McGill大学)进行图构建和PLS-DA分析。
结果
主题和样品
在本分析进行时,REIMAGINE研究前瞻性地收集了232例连续接受任何形式上镜检查(食管胃十二指肠镜(EGD)或口服双球囊内镜(DBE))的受试者的十二指肠分泌物,这些受试者在内镜检查前6个月内未服用任何抗生素。其中,53名接受EGD的受试者提供了十二指肠吸出物和匹配的粪便样本1).该组作为比较小肠近端和粪便微生物组的基础。第二组23名受试者进行了DBE,以遍历整个小肠,并提供了十二指肠和空肠的腔内液体样本和在该过程中达到的最远距离(FD)。在这23名DBE受试者中,有8人提供了匹配的粪便样本,形成了第三组进行分析(见表)1).
十二指肠和粪便微生物图谱(第1组)
对于在EGD期间同时提供十二指肠样本和粪便样本的53名受试者,相应微生物群的beta多样性分析在PCoA图上显示了两个完全不同的集群(图1)。1a),并在十二指肠和粪便微生物群之间确定了超过2000个OTUs的差异(图。1b)。
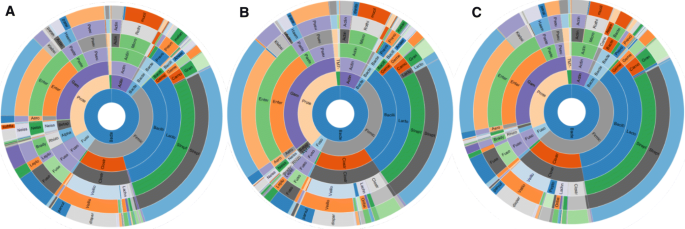
十二指肠微生物组的特征是厚壁菌门门(~ 55%)和变形菌门(~ 21%)的相对丰度较高,而粪便微生物组的拟杆菌门(~ 50%)和厚壁菌门(~ 40%)的相对丰度较高(补充表1)。与粪便中的微生物剖面相比,十二指肠的变形菌门、梭菌门、放线菌门和TM7门的相对丰度显著增加。最后一种仅存在于十二指肠菌群中(补充图1)。与粪便相比,十二指肠菌群中厚壁菌门的相对丰度也有所增加(Fold Change, FC = 1.40, FDRPvalue = 0.004)(表2).相比之下,与粪便相比,十二指肠菌群中的拟杆菌门显著减少,而Verrucomicrobia门在十二指肠中几乎检测不到(补充图1)2).在属水平上,十二指肠菌群中共有156个属的相对丰度与粪便菌群的相对丰度存在统计学差异,欧几里德沃德聚类分析揭示了两种不同的微生物特征(补充图2)。
不同小肠节段的微生物图谱(组2)
共有23名受试者在小肠的三个不同节段(十二指肠、空肠和DBE过程中达到的最远距离[FD])进行了微生物组的定位(表1).通过辛普森指数、香农熵和总OTU数(ANOVA)确定,所有三个区段的微生物α多样性相似P值> 0.05)。根据UniFrac的非加权距离,所有三个小肠段的beta多样性也相似,在PCoA图上没有观察到明显的聚类(补充图3)。在这些小肠段中观察到的主要门是厚壁菌门、变形菌门和放线菌门,它们加起来几乎占总相对丰度的90%(图3)。2其余的门,包括梭杆菌门、拟杆菌门和TM7,在小肠中的相对丰度均< 10%(补充表2)。虽然该模式在所有分析的节段中都是一致的,但其中存在一些变化。具体来说,拟杆菌门、变形菌门和梭菌门在不同小肠节段的相对丰度不同(似然比检验(LRT) FDRP值< 0.05),空肠变形菌门相对丰度高于十二指肠(FC = 9.99, FDRPvalue = 5.39E−4)和FD (FC = 8.16, FDR . value = 5.39E−4)和FD (FC = 8.16, FDR . value = 5.39E−4)P值= 2.64 e−3)。在这23个科目中,不论小肠节段,最丰富的前5个纲分别是杆菌和梭菌门(厚壁菌门)、γ变形菌门(变形菌门)、放线菌门(放线菌门)和梭菌门(梭菌门)。然而,Gammaproteobacteria和fusobacteria两个纲的相对丰度在不同段之间存在差异(LRT FDRP值< 0.001,FDRP值= 2.72E−7),空肠的γ变形菌门含量高于十二指肠和FD (FDRPvalue = 2.86E−4,FDRP值分别= 0.02)。
在科水平上,在十二指肠微生物组中确定了111个科,在空肠微生物组中确定了99个科,在FD微生物组中确定了101个科(补充图4)。在所有测试的小肠段中,最丰富的科是链球菌科(p_Firmicutes, c_bacillus, o_lactobacillus),占每段整个微生物组组成的26%以上,其次是肠杆菌科(p_Proteobacteria,c_Gammaproteobacteria,o_Enterobacteriales),细孔菌科(p_Firmicutes,c_Clostridia,o_Clostridiales)占各节段的21%,占各节段的9%。11个微生物家族的相对丰度随小肠位置的不同而不同(补充表3)。与空肠和FD相比,十二指肠微生物组的梭状芽孢杆菌科(Clostridiaceae)家族的相对丰度降低,而慢根瘤菌科(Bradyrhizobiaceae)家族的相对丰度增加(P< 0.05,补充表3)。与十二指肠和空肠相比,FD微生物组中Peptostreptococcaceae家族的相对丰度增加(P< 0.05,补充表3)。此外,FD中许多其他科的相对丰度与十二指肠不同,包括杆菌科(bacteraceae)、拟杆菌科(Bacteroidaceae)、肠球菌科(Enterococcaceae),以及变形菌门(Proteobacteria门)的许多科,如Enterobacteriaceae、Moraxellaceae、Neisseriaceae(补充表3)。
为了通过旋转PCoA组分来增强节段之间的分离,从而获得肠段之间的最大分离,在家族水平上进行PLS鉴别分析(PLS- da),结果显示空肠内的微生物特征与十二指肠和FD的微生物特征重叠(补充图5A)。相比之下,十二指肠样本的家族级微生物特征与FD中识别的特征显著不同(补充图5B),并且在PLS-DA图上观察到的与分离相关的投影(VIP)评分中排名前3的变量重要性分别为普雷沃菌科、Neisseriaceae和Moraxellaceae(补充图6)。
不同小肠节段与粪便的微生物图谱(第3组)
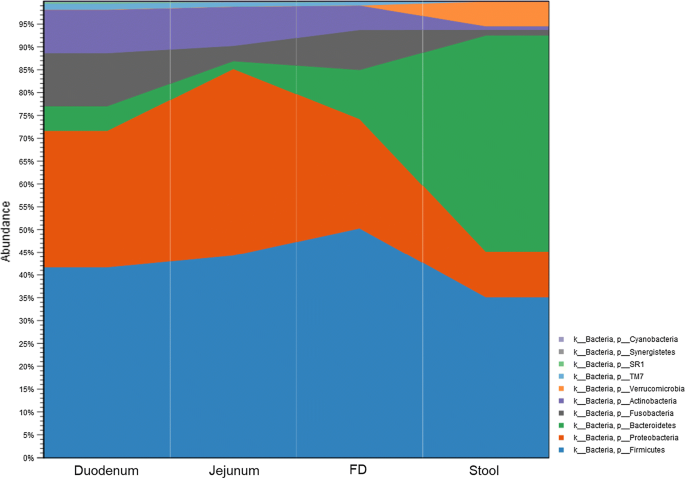
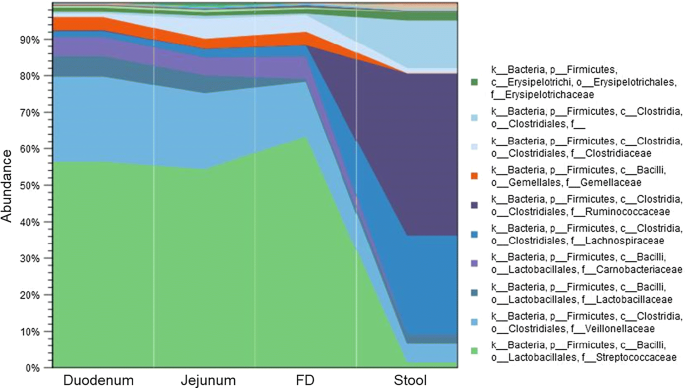
最后,我们比较了小肠不同部位(如十二指肠、空肠和FD样本)的微生物组谱,与来自同一个体的粪便样本(n= 8)。结果显示,从十二指肠到粪便的微生物组有明显的差异。3.厚壁菌门在小肠和粪便中都高度丰富。它是在小肠中检测到的主要门,代表了所有节段中总微生物相对丰度的50%以上,是在粪便中检测到的第二大门。总体而言,十二指肠厚壁菌门的相对丰度略高于粪便(表2).然而,与粪便相比,小肠段中该门内的微生物组成有明显不同(图1)。4).在小肠段,厚壁菌门主要由乳酸菌目(占所有厚壁菌门的68%)所代表,其中包括来自链球菌科、乳酸菌科和肉杆菌科的乳酸菌(兼性厌氧菌),而后者在粪便中是不存在的(图1)。4).相比之下,粪便中的厚壁菌门主要以梭状芽孢杆菌目(占所有厚壁菌门的93%)为代表,其中包括瘤胃球菌科、拉克诺螺旋菌科和克里斯滕森菌科的专性厌氧菌,而后者在小肠中是不存在的。与小肠的所有节段相比,这三个家族在粪便微生物组中的相对丰度增加了(补充表4)。与粪便相比,变形菌门在十二指肠微生物组中的相对丰度增加了(表4)2).在小肠中,该门的特征是具有较高丰度的奈瑟菌科(β -变形菌纲)(FC = 5.57, FDRP值= 2.13E−14),巴氏杆菌科和肠杆菌科(Gammaproteobacteria类)(FC = 4.93, FDRPvalue = 2.30E−12 and FC = 3.35, FDRPvalue = 1.03E−4)(补充图8A)。大便变形菌门以碱性细菌科(Alcaligenaceae)和H2S产菌来自Deltaproteobacteria (FC = 3.32, FDRPvalue = 2.10E−03,and FC = 1.82 FDR,P值分别= 0.03)(补充图8B)。
讨论
在过去的十年中,人们对人类微生物群在人类健康和疾病中的作用产生了极大的兴趣。人类微生物组项目开始了大量的工作,这些工作后来主导了文献[1].最初对人类微生物群的描述包括对嘴、鼻子、皮肤、阴道和肠道的分析。粪便被用作整个“肠道”的替代品,大量研究发现,粪便微生物群的两大门——厚壁菌门和拟杆菌门的比例存在差异。这些利用粪便进行的研究有两个关键发现,一是不同细菌种群相对丰度的不平衡很重要,二是微生物多样性的减少与各种疾病和条件之间的关联。例如,最初的数据表明,肥胖可能与拟杆菌门/厚壁菌门比值较高有关[6,7].就多样性而言,数据一致表明,炎性肠病等严重肠道疾病与粪便微生物群多样性的减少有关[8,9].
虽然与微生物组相关的发现在不断发展,但使用粪便作为整个肠道的替代品仍有局限性。粪便易于获取,细菌数量高,使DNA提取和扩增更容易。然而,结肠只占整个肠的4-5英尺。小肠长达20英尺,是胃肠道的重要组成部分。它是胃内容物(食物)、胆汁和胰腺分泌物汇集的地方,是肠道的主要吸收面。此外,考虑到肠绒毛和微绒毛,小肠的表面积很大。因此,小肠微生物的影响是潜在的巨大的,但目前还没有对整个小肠和大肠的微生物组进行系统的研究,尽管先前的研究基于健康志愿者和疾病状态下的活检和腔内内容物分析评估了小肠微生物组的组成[19,20.,21,22,23,24,25].这项研究是REIMAGINE研究的一部分,该研究是一项大规模的努力,旨在检查和了解小肠的微生物组成在人类健康和疾病中的作用,并提供了小肠和粪便微生物群之间巨大差异的详细分析。我们的研究结果表明,小肠微生物群是独特的,粪便并不是整个肠道微生物群的替代品。在小肠和粪便微生物群之间发现了许多差异。粪便微生物组的特点是两个主要门,拟杆菌门和厚壁菌门相对丰度高。小肠微生物群也以两大门为主,但它们是厚壁菌门和变形菌门,拟杆菌门相对丰度较低,可能是由于小肠中胆汁酸含量较高[26].虽然粪便和小肠微生物群之间的共同联系似乎是厚壁菌门,但仔细检查发现,小肠中的厚壁菌门的组成也与粪便中的有很大不同,其主要特征是链球菌科、细孔菌科和乳酸菌科。链球菌科和乳酸菌科产生乳酸作为葡萄糖发酵的最终产物[27,28].乳酸菌在肠道中的作用尚不完全清楚,但许多研究已将这些细菌与肠道和免疫系统的改善联系起来,并与调节肠道菌群对肥胖发展的影响有关[29,30.,31].
小肠中的变形菌门的组成也与粪便中的不同。来自三角洲变形菌纲的物种在粪便中更为丰富,包括亚硫酸氢产生者如脱磷孤菌属sp.和Bilophilasp。32,33].相比之下,在小肠中变形菌门以γ变形菌纲为主要代表,它包括几种具有医学和科学重要性的已知病原体[34].另外,变形菌属的种常分离于唾液等嗜血杆菌sp.和奈瑟氏菌属subflava,也大量存在于小肠中[35].小肠微生物组也包含不同的分类群,如TM7。候选门TM7主要存在于小肠中,占总细菌丰度的2%。在粪便中发现了微量的TM7。这一独特的门与人类疾病有关,如炎症性粘膜疾病,尽管它在全球流行,但仍然无法培养,使其成为已知的最神秘的门之一[36,37].同样,粪便微生物组显示出明显丰富的类群从疣菌门。
直到最近,深小肠还无法进入。这种被称为双气囊内窥镜的内窥镜技术几乎可以检查小肠的所有层面,在某些情况下还可以到达盲肠。这项技术可以对几乎整个肠道的微生物群进行评估。在这项研究中,最初似乎小肠有一致的细菌图谱。然而,进一步的分析显示,在小肠内更多远端位置的样本中,微生物组逐渐发生了变化。这是通过特定类群的微妙变化而凸显出来的,例如拟杆菌门的逐渐增加和变形菌门的减少,但这些仍然没有达到结肠中发现的水平。我们的许多发现与以前的研究结果一致,这些研究使用荧光原位杂交(FISH)、焦糖测序和16S rRNA基因测序等技术,对肠易激综合征(IBS)和乳糜泻等疾病和健康对照组的小肠微生物组进行了检查和比较。22,23,24,25].例如,Dlugosz等人[23]检测了肠易激综合征受试者和健康对照者空肠粘膜相关微生物组,发现在肠易激综合征受试者和对照者中,主要门的丰度顺序相似(厚壁菌门43/ %,变形菌门23%,细菌门15%,放线菌门9.3%),并确定了该属的高丰度链球菌而且韦永氏球菌属低,但埃希氏杆菌属.然而,Chung等人。24]发现在健康对照组的粘膜相关空肠菌群中,变形菌门(47.7%)的丰度较高,拟杆菌门(15.3%)的丰度相似,厚壁菌门(11%)的丰度较低。有趣的是,这项研究发现家庭Prevotellaceae更丰富,而且奈瑟氏菌科在肠易激综合征受试者与对照组的粘膜相关空肠微生物群中含量较低,并发现两者之间存在关联奈瑟氏菌科肠易激综合征症状的严重程度[24].李等人。[19]在一项针对9名健康志愿者的小型研究中检查了十二指肠管腔和粘膜相关的微生物群,以及粪便和直肠粘膜相关的微生物群,并发现厚壁菌门和变形菌门在十二指肠样本中占主导地位,属普氏菌,Stenotrophomonas,而且链球菌在十二指肠腔标本中占主导地位。最后,在一项对乳糜泻患者、无谷蛋白饮食的乳糜泻患者和对照组的十二指肠活检研究中,D 'Argenio等人[38发现了相对丰度最高的属链球菌,普氏菌、Propioibacterium而且不动杆菌在健康对照组中发现了这个属奈瑟氏菌属在乳糜泻患者的十二指肠样本中明显多于对照组。
由于本研究具有前瞻性,因此,回顾小肠取样的历史方法作为对照也很重要。虽然有新的新兴设备可以对小肠微生物组进行取样(电子胶囊),但沃森胶囊已经存在了几十年,主要用于小肠活检,但也被用于评估小肠微生物组[39].这种胶囊的挑战是它缺乏无菌性,因为它必须通过上部通道和胃。另一个挑战是定位,这需要放射学。另一种评估小肠微生物群的方法甚至更早。早在1969年就有人对回肠流出物进行微生物学研究,但这些研究主要是在回肠造口术患者中进行的[40,41].这里的挑战是结肠有大量的细菌可以影响回肠。在完整的肠道中,回肠流出物需要结肠镜检查和结肠镜准备,这众所周知会影响小肠和结肠微生物群。
尽管REIMAGINE研究旨在成为小肠微生物组成的决定性大规模研究,但目前的工作仍有局限性。该研究的一个缺陷是,这些受试者正在接受上消化道内窥镜检查,以评估肠道疾病,包括消化不良、胃食管反流症、贫血等。这些条件可能会影响研究结果。然而,反对这一观点的一个论点是假设足够的样本量回归均值的概念。另一个可能的限制是考虑生物量。在微生物组分析中,相对丰度是一个考虑因素,但另一个考虑因素是特定分类学组中细菌的绝对数量。例如,与粪便相比,小肠中变形菌门的相对丰度可能更大,但每毫升粪便中变形菌门的绝对数量可能更大。然而,这并没有考虑到与小肠非常大的表面积有关的生物量。因此,这种影响和真正的生物量很难计算。
总之,小肠微生物群在许多方面都是独特的。考虑到小肠的表面积和长度以及它对人类营养和免疫功能的重要性,分析小肠微生物群可能比分析粪便对我们了解人类疾病有更大的影响。正如这项研究所表明的,人类小肠菌群主要由厚壁菌门和变形菌门组成。深入分析发现,在主要的门内,特定的分类群在小肠的不同部位的分布是不同的。了解包括小肠在内的肠道所有部位微生物组的这些组成变化,对于发现未来与人类健康和疾病的联系将非常重要。
数据可用性
在当前研究中生成的数据集可在国家生物技术信息中心(NCBI)生物项目资源库中获得https://www.ncbi.nlm.nih.gov/bioproject生物项目ID PRJNA590519。
参考文献
特恩堡PJ,莱雷,哈马迪M,弗雷泽-利格特CM,奈特R,戈登JI。人类微生物组项目。自然.2007; 449:804 - 810。
科斯特洛·EK, Lauber CL, Hamady M, Fierer N, Gordon JI, Knight R.人体生境中细菌群落的时空变异。科学.2009; 326:1694 - 1697。
吕宏,姚涛,李晓燕,等。胃肠道微生物生态学研究进展。微生物环境。.2017; 32:300 - 313。
西尔斯CL。一个充满活力的伙伴关系:庆祝我们的肠道菌群。厌氧生物.2005; 11:247 - 251。
协会的高分子聚合物。健康人体微生物群的结构、功能和多样性。自然.2012; 486:207 - 214。
Turnbaugh PJ, Ley RE, Mahowald MA, Magrini V, Mardis ER, Gordon JI。一种与肥胖相关的肠道微生物群,其能量收集能力增强。自然.2006; 444:1027 - 1031。
Ley RE, Backhed F, Turnbaugh P, Lozupone CA, Knight RD, Gordon JI。肥胖会改变肠道微生物生态。美国国家科学研究院.2005; 102:11070 - 11075。
莫尼汉C, Rigottier-Gois L, Bonnaud E,等。宏基因组方法揭示克罗恩病粪便菌群多样性减少。肠道.2006; 55:205 - 211。
Durban A, Abellan JJ, Jimenez-Hernandez N,等。肠易激综合征中粪便和粘膜相关细菌群落的结构改变。环境Microbiol代表.2012; 4:242 - 247。
通过微生物作用改善生活:哺乳动物胃肠道菌群对宿主的益处。环境Microbiol.2009; 11:2194 - 2206。
王晓燕,王晓燕,王晓燕,等。sa1912 -揭示整个肠道菌群及其与遗传、免疫和神经内分泌生态系统的关系重新定义研究。胃肠病学.2019; 156: s - 450。
Sender R, Fuchs S, Milo R.对人体和细菌细胞数量的修正估计。公共科学图书馆杂志.2016; 14: e1002533。
Erdogan A, Rao SS, Gulley D, Jacobs C, Lee YY, Badger C.小肠细菌过度生长:十二指肠吸痰vs葡萄糖呼吸试验。Neurogastroenterol Motil.2015; 27:481 - 489。
Jacobs C, Coss Adame E, Attaluri A, Valestin J, Rao SS.运动障碍和质子泵抑制剂的使用是小肠细菌和/或真菌过度生长的独立危险因素。食物药物杂志.2013; 37:1103 - 1111。
Leite G, Morales W, Weitsman S,等。优化小肠吸出物微生物组测序:通过REIMAGINE研究验证新技术。BMC Microbiol.2019;。https://doi.org/10.1186/s12866-12019-11617-12861.
Klindworth A, Pruesse E, Schweer T,等。16S核糖体RNA基因PCR引物用于经典和下一代基于测序的多样性研究的评价。核酸Res.2013年,41:e1。
不浪费,不需要:为什么稀少的微生物组数据是不被接受的。公共科学图书馆第一版杂志.2014; 10: e1003531。
Weiss S,徐喆,pedada S,等。归一化和微生物差异丰度策略取决于数据特征。微生物组.2017年,27。
李G,杨米,周K, et al。健康志愿者十二指肠和直肠活检组织和管腔内容物中微生物群的多样性。生物科技J Microbiol》.2015; 25:1136 - 1145。
陈娟,陈建军,陈建军,等。小肠微生物失调是功能性胃肠疾病相关症状的基础。Nat Commun.2019; 10:2012。
Seekatz AM, Schnizlein MK, Koenigsknecht MJ,等。禁食健康人胃和小肠菌群的时空分析。mSphere.2019; 4: e00126-19。
柯克霍夫斯,本-阿莫尔,萨姆姆,等。粪便和十二指肠样本的分子分析显示明显较高的患病率和数量铜绿假单胞菌在肠易激综合症中。J地中海Microbiol.2011; 60:236 - 245。
Dlugosz A, Winckler B, Lundin E,等。肠易激综合征患者与健康对照组间小肠菌群无差异。Sci代表.2015; 5:8508。
钟春春,常鹏飞,廖春春,等。肠易激综合征患者与健康受试者小肠及粪便微生物菌群的差异。Scand杂志.2016; 51:410 - 419。
李G,杨米,金Y, et al。腹泻型肠易激综合征中十二指肠和直肠共有粘膜相关菌群的参与。J杂志.2018; 33:1220 - 1226。
Ridlon JM, Kang DJ, Hylemon PB, Bajaj JS。胆汁酸和肠道微生物群。当今杂志.2014; 30:332 - 338。
乳酸菌科。在:Holzapfel WH, Wood BJB, eds。乳酸菌:生物多样性与分类学.纽约:威利;2014.
du Toit M, uch M, Cho GS, Franz CMAP。链球菌属。在:Holzapfel WH, Wood BJB, eds。乳酸菌:生物多样性与分类学.纽约:威利;2014.
乳酸菌对肠道菌群复杂性的贡献:光与影。前端细胞感染微生物.2012; 2:86。
Perdigon G, Maldonado Galdeano C, Valdez JC, Medici M.乳酸菌与肠道免疫系统的相互作用。Eur J Clin Nutr.2002; 56: S21-S26。
蔡东涛,郑pc,潘天明。肠道菌群的抗肥胖作用与乳酸菌有关。生物科技:Microbiol》.2014; 98:1-10。
Warren YA, Citron DM, Merriam CV, Goldstein EJ。脱硫弧菌属与其他表型相似属的生化分化与比较。中国Microbiol.2005; 43:4041 - 4045。
Peck SC, Denger K, Burrichter A, Irwin SM, Balskus EP, Schleheck D.一种甘氨酸自由基酶使人类肠道细菌产生硫化氢Bilophila wadsworthia.美国国家科学研究院.2019; 116:3171 - 3176。
Rizzatti G, Lopetuso LR, Gibiino G, Binda C, Gasbarrini a .变形菌门:人类疾病的常见因素。生物医学Res Int.2017; 2017:9351507。
王晓燕,王晓燕,王晓燕,等。健康成人口腔菌群的焦磷酸测序分析。J削弱Res.2008; 87:1016 - 1020。
He X, McLean JS, Edlund A,等。培养人类相关的TM7系统型揭示了减少的基因组和表观寄生生活方式。美国国家科学研究院.2015; 112:244 - 249。
Ferrari B, Winsley T, Ji M, Neilan B.对无处不在的候选菌的分布和丰度的洞察Saccharibacteria门以下标签焦磷酸测序。Sci代表.2014; 4:3957。
D 'Argenio V, Casaburi G, Precone V,等。宏基因组学揭示了生物失调和潜在的致病性n刺蛾成人乳糜泻患者十二指肠菌株。是杂志.2016; 111:879 - 890。
苏立文,李志刚,李志刚,等。健康和疾病时小肠的菌群。厌氧生物.2003; 9:11-14。
Percy-Robb IW, Brunton WA, Jalan KN, McManus JP, Gould JC, Sircus W.回肠造口术患者回肠出水细菌含量与胆盐代谢的关系肠道.1969; 10:1049 - 1050。
boijink CC, El-Aidy S, Rajilic-Stojanovic M,等。在人类回肠菌群中检测到高时间和个体间变异。环境Microbiol.2010; 12:3213 - 3227。
作者信息
作者和联系
相应的作者
道德声明
的利益冲突
作者声明他们没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
电子辅料
下面是电子补充材料的链接。
权利和权限
开放获取本文遵循创作共用署名-非商业用途4.0国际许可协议(Creative Commons Attribution-NonCommercial 4.0 International License),该协议允许以任何媒介或格式进行任何非商业用途、分享、改编、发布和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by-nc/4.0/.
关于这篇文章
引用这篇文章
雷特,G.G.S,韦茨曼,S,帕罗迪,G。et al。测绘人类小肠节段菌群与粪便的比较:一项重新想象的研究。挖说科学65, 2595 - 2604(2020)。https://doi.org/10.1007/s10620-020-06173-x
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1007/s10620-020-06173-x
关键字
- 微生物组
- 十二指肠
- 空肠
- 凳子
- 16 s宏基因组分析