文摘
了解肠道微生物对人类健康和福祉的影响至关重要,评估他们的遗传潜力。在这里,我们描述了Illumina-based宏基因组测序、装配和330万微生物基因冗余特征,源于576.7碱基序列,从124欧洲人的粪便样本。基因集,∼人类基因补150倍,包含了绝大多数的流行(更频繁)微生物的基因群,可能包括一个大的比例普遍人类肠道微生物的基因。基因在很大程度上个人之间共享的队列。超过99%的基因是细菌,这表明整个队列港口1000年和1150年之间普遍的细菌种类,每个人至少160个这样的物种,也很大程度上共享。我们定义和描述最小肠道metagenome和最小的肠道细菌基因组的功能出现在所有个人和大多数细菌,分别。
主要
据估计,我们体内的微生物共同组成100万亿个细胞,十倍人类细胞的数量,建议他们独特的基因编码100倍比我们自己的基因组1。大多数居住在肠道微生物,对人体生理和营养产生深远影响,并为人类生活是至关重要的2,3。此外,肠道微生物导致的能量收获从食物,和肠道微生物的变化可能与肠道疾病和肥胖有关4,5,6,7,8。
理解和利用的影响肠道微生物对人类健康和福祉有必要解释内容,肠道微生物的多样性和功能的社区。16 s核糖体RNA基因(rRNA)序列的方法9显示两个细菌分裂,拟杆菌门和厚壁菌门,占超过90%的已知的系统分类和控制远端肠道微生物群10。研究还显示,大量的健康个体之间的肠道微生物的多样性4,8,10,11。虽然婴儿这种差异尤其明显12在今后的生活中,肠道微生物组收敛于更多类似的门。
宏基因组测序是一个强大的替代rRNA测序分析复杂的微生物群落13,14,15。应用于人类的肠道,这样的研究已经产生一些3 Gb (Gb)的粪便中微生物序列33个人从美国或日本8,16,17。得到更广泛的概述我们使用的人类肠道微生物基因Illumina公司基因组分析器(GA)技术进行深入的粪便中总DNA的测序124年欧洲成年人。我们生成576.7 Gb的序列,在所有先前的研究,近200倍组装成叠连群,并预测330万年独特的开放阅读框(orf)。这个基因目录包含了几乎所有的常见肠道微生物的基因在我们的群体中,提供了一个广泛的功能重要的细菌生活在肠道和表明,很多细菌物种是由不同的人共享。我们的研究结果还表明,短内容宏基因组测序可以用于遗传潜力的全球特性复杂生态环境。
肠道微生物宏基因组测序
MetaHIT的一部分(人类肠道)的宏基因组项目,我们收集粪便标本124健康成年人超重和肥胖的个体,以及炎症性肠病(IBD)患者,从丹麦和西班牙(补充表1)。从粪便标本中提取总DNA18和平均4.5 Gb(介于2和7.3 Gb)为每个生成的数据序列样本,允许我们去捕捉最新奇的(见方法和补充表2)。总的来说,我们获得了576.7 Gb的序列(补充表3)。
想要生成一个广泛的目录从人类肠道微生物的基因,我们第一次组装短Illumina公司读取到再叠连群,这可以通过标准方法分析和注释。使用SOAPdenovo19,德Bruijn图论工具专门设计的装配很短的读取,我们执行新创组装的所有Illumina公司GA序列数据。因为高多样性预计个人之间8,16,17,我们首先组装每个独立样本(补充图3)。多达42.7%的Illumina公司GA读取被组装成658万叠连群长度> 500个基点,给总叠连群长度10.3 Gb, 2.2 kb的将军长度(补充图4)和12.3 - 237.6 Mb的范围(补充表4)。几乎35%的读取来自任何一个样本可以从其他样本映射到叠连群,表明存在一个共同的核心序列。
Illumina公司的质量评估遗传大会我们样本的叠连群MH0006映射和MH0012桑格读取相同的样品(补充表2)。总共有98.7%的重叠群映射到至少一个桑格读是共线的绘制区域的99.6%以上。这是与454测序所产生的叠连群,两个样本(MH0006)的控制,其中97.9%是共线的绘制区域的99.5%以上。我们估计每megabase装配误差是14.2和20.7 (Mb)的Illumina公司——和454 -基于重叠群,分别为(见方法和补充图5),表明短期和long-read-based装配精度相当。
完成叠连群设置我们从所有的124个样本,池未装配的读取和重复了新创装配过程。大约040万因此产生更多的重叠群,拥有一个370 Mb的长度和一个将军939个基点的长度。我们最后重叠群的总长度是10.7 Gb。约80%的576.7 Gb的Illumina公司GA可以对齐序列重叠群的身份的阈值为90%,考虑到住宿的测序错误和应变变化在肠道(图1),几乎两倍的42.7%的序列被SOAPdenovo组装成叠连群,因为装配使用更严格的标准。这表明绝大多数的Illumina公司序列是由我们的叠连群表示。
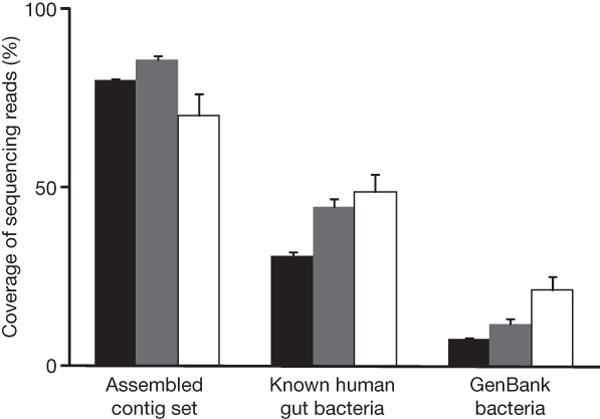
这三个人类微生物测序读sets-Illumina GA读取来自124人在这项研究中(黑色;n= 124),罗氏/ 454读取人类从18岁双胞胎和他们的母亲(灰色;n= 18)和桑格读取来自13个日本人(白;n= 13)-每个参考序列集的对齐。平均值±s.e.m.绘制。
人类肠道微生物组的比较表示在我们叠连群,从以前的工作,我们一致的读取最大肠道metagenome发表的两项研究(1.83 Gb的罗氏公司/ 454测序读来自18个美国成年人8和0.79 Gb的桑格读取来自13个日本成人和婴儿17),使用90%的身份门槛。共有70.1%的和85.9%的读取从日本和美国样本,分别可以对齐重叠群(图1),显示序列重叠群包括一个高分数的先前的研究。相比之下,85.7%和69.5%的重叠群没有覆盖的读取从日本和美国样本,分别突出我们捕获的新奇。
只有31.0 - -48.8%的读取两个先前的研究和本研究可以对齐到194年公共人类肠道细菌基因组(补充表5),7.6 - -21.2%的细菌基因组存入基因库(图1)。这表明参考基因集分离菌株的基因组测序获得的规模仍然有限。
一个基因人类肠道微生物组的目录
建立一套冗余人类肠道微生物基因我们第一次使用MetaGene20.项目预测子在我们叠连群,发现14048045 orf超过100个基点(补充表6)。他们占领了86.7%的叠连群,可比价值发现完全测序的基因组(∼86%)。三分之二的orf出现不完整,可能是因为我们重叠群的大小(将军2.2 kb)。我们接下来删除冗余子,通过两两比较,使用一个非常严格的标准95%的身份超过90%的ORF长度越短,可以融合orthologues但是避免通货膨胀的数据集可能由于测序错误(见方法)。然而,最终的冗余基因集包含多达3299822个orf平均长度为704个基点(补充表7)。
我们学期的基因刚好普遍的基因,因为它们编码的重叠群装配最丰富的读取(见方法)。最小的相对丰度普遍的基因∼6×107,估计从序列的最小覆盖的独特基因(接近3),和总Illumina公司生成的序列长度为每个单独的(平均4.5 Gb),假设平均基因长度0.85 kb(也就是说,3×0.85×103/ 4.5×109)。
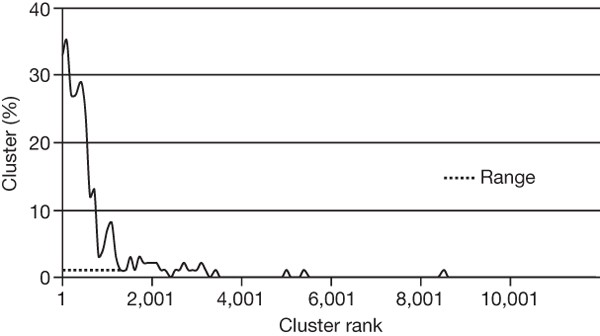
我们绘制了330万肠道orf的319812个基因(目标基因)89年频繁引用人类肠道中的微生物基因组。标识阈值90%,80%的目标基因至少有80%的长度由单个肠道ORF (图2 b)。这表明基因集包括大多数的已知人类肠道细菌基因。
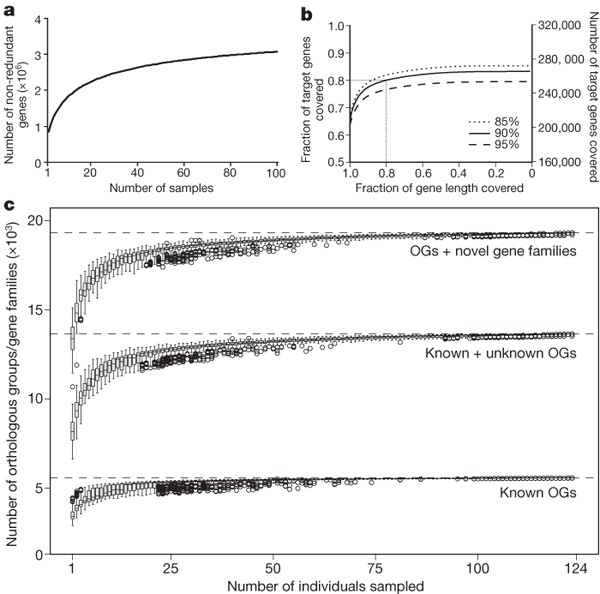
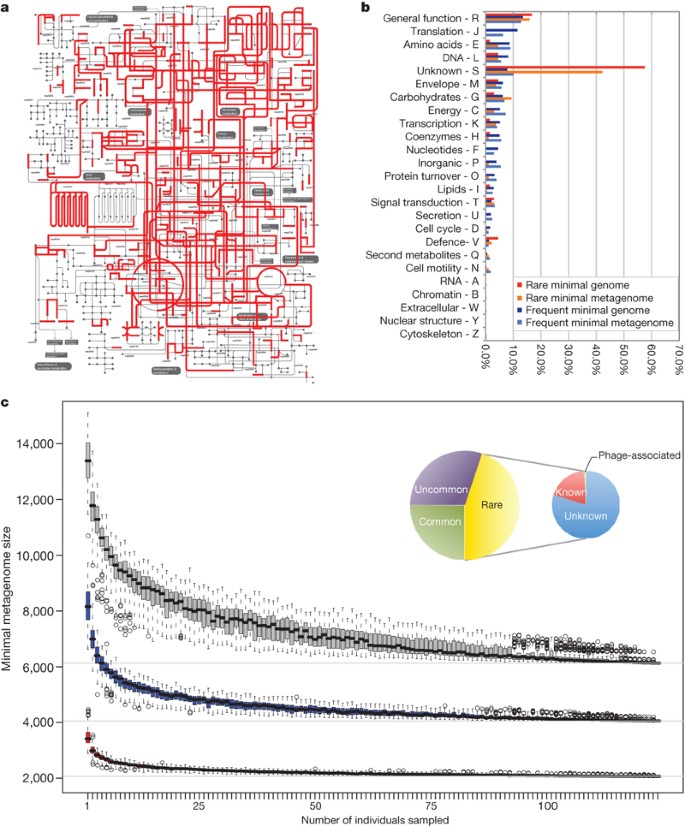
一个、独特的基因测序的程度的函数。基因累积曲线对应于年代奥林匹克广播服务公司毛(τ)值(观察到基因的数量),使用估计计算21(版本8.2.0)随机选择100个样本(由于内存限制)。b频繁的报道基因从89年肠道微生物物种(补充表12)。c,数量的函数被数量的样本调查,基于已知(特征)同源组(og;底),已知和未知同源组(包括,例如,假定的,预测,守恒假设函数;小说中)和同源组+基因家族(> 20蛋白质)恢复从metagenome(上)。框之间的四分位范围(差)表示第一和第三个四分位数(分别为25和75百分位数)和里面的线表示中位数。胡须表示最低和最高的值在1.5倍差从第一和第三个四分位数,分别。圆圈表示异常值超出了胡须。
我们检查的数量普遍基因识别所有个人的程度排序的函数,要求至少有两个支持读取基因调用(图2一个)。incidence-based报道丰富估计量(冰),决定在100人(最多的估计21程序可以适应),表明我们的目录中获得85.3%的普遍的基因。虽然这可能是低估了,不过它表示目录包含普遍的绝大多数人群的基因。
每个人携带536112±12167(平均±s.e.m)普遍的基因(补充图6 b),这表明大部分的330万个基因池必须共享。然而,大多数流行的基因被发现只有少数人:2375655中不到20%,而294110年发现至少有50%的人(我们这些常见的基因)。这些值取决于采样深度;测序MH0006和MH0012透露更多的目录基因,出席低丰度(补充图7)。然而,即使在我们常规采样深度,每个人怀有204056±3603(平均±s.e.m)共同的基因,表明大约38%的个人总基因库是共享的。IBD患者包庇,有趣的是,平均而言,比个人更少的基因25%患有炎症性肠病(补充图8),与观测一致,前者比后者细菌多样性较低22。
常见的细菌核心
深宏基因组测序提供了机会去探索存在的一组常见的微生物物种(共同核心)队列。为了这个目的,我们使用了非冗余的650集细菌和古细菌基因组测序(见方法)。我们对齐每个人类肠道微生物样本的Illumina公司GA读取到基因集,使用90%的身份门槛,并确定基因的比例由对齐到只有一个位置的读取。1%的覆盖率,这对于一个典型的肠道细菌基因组对应的平均长度约40 kb,一些25倍以上的16 s基因通常用于物种鉴定,我们发现18个物种在所有个人、57≥90%和75年在≥50%的个人(补充表8)。在10%的覆盖率,要求∼高10倍丰度在一个示例中,我们仍然发现13以上的物种在≥90%的个人和35≥50%。
当累积序列长度从3.96 Gb增加到8.74 Gb和4.41 Gb 11.6 Gb,样品MH0006和MH0012分别菌株的数量普遍在1%的覆盖率两个阈值增加了25%,从135年到169年。这表明存在明显比我们能够观测到较大的共同核心序列深度通常为每个单独的使用。
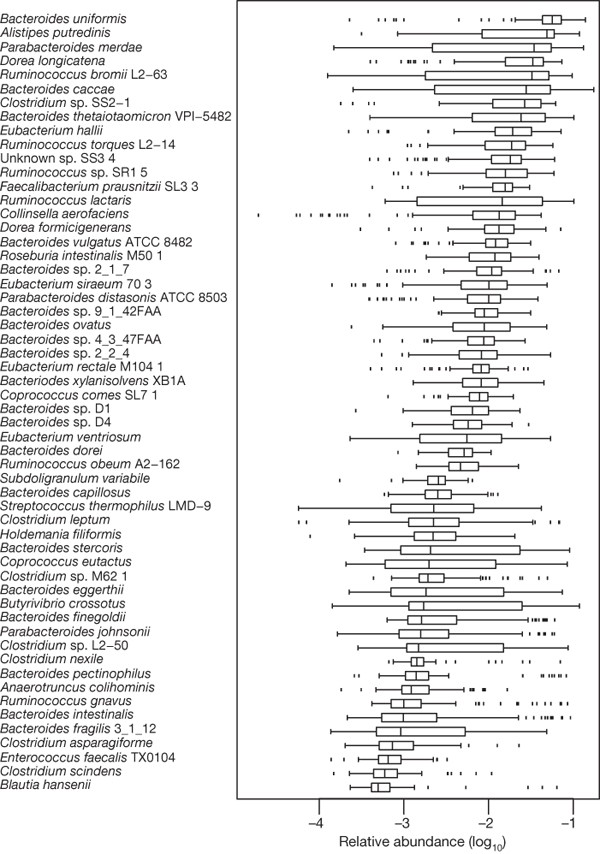
大量的微生物物种的变化在个人可以极大地影响识别常见的核心。想象这种可变性,我们比较了不同基因组测序读取次数一致在我们群的人。即使对最常见的57个物种出现在≥90%的个人基因组覆盖率> 1% (补充表8),inter-individual变异性与12 - 2187倍(图3)。正如预期的10,23拟杆菌门和壁厚菌门丰度最高。
看到图2 c定义的盒子,须阴谋。看到计算的方法。
一个复杂的亲缘物种的模式,以集群属和家庭层面,走出网络的分析基于成对皮尔逊相关系数的155个物种存在于至少一个个体(≥1%的覆盖率补充图9)。突出集群包括一些肠道最丰富的物种,如拟杆菌门和成员Dorea/真细菌/瘤胃球菌属组织和双歧杆菌,变形菌门和链球菌、乳酸杆菌组。这些观察结果表明,相似的细菌可能存在于不同的个人星座群,原因仍有待建立。
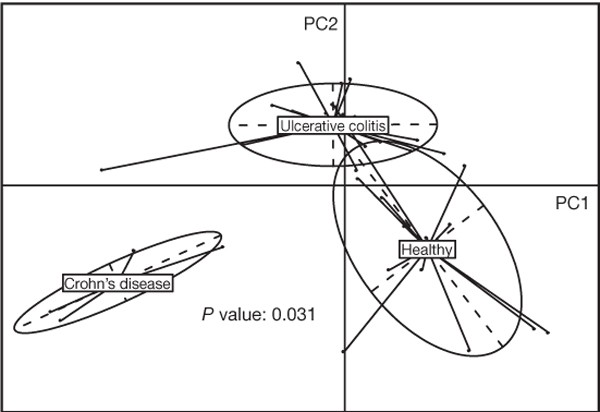
上述结果表明,Illumina-based细菌分析应该揭示健康个体和病人之间的差异。为了测试这个假说我们比较IBD患者和健康对照组(补充表1),因为它曾报道,两个具有不同的微生物群22。主成分分析,基于相同的155种,明确地分离患者与健康个体和溃疡性结肠炎与克罗恩病病人(图4),证实了我们的假设。
主成分分析与健康状况作为辅助变量,基于155个物种的丰度≥1%的基因组覆盖率的Illumina公司至少1个人的阅读群体,进行了14个健康个体和25 IBD患者(溃疡性结肠炎和克罗恩病4日)21日从西班牙(补充表1)。一分之二组件(PC1和PC2)策划和惯性占整体的7.3%。个人(由分)集群,为每个类重心计算;P价值的健康状况和物种丰度之间的联系是由蒙特卡罗试验(999复制)。
普遍的基因编码的功能集
我们通过调整分类预测基因的数据库进行集成NCBI-NR冗余蛋白质序列,基因在KEGG(京都基因和基因组的百科全书)24途径和齿轮(集群的同源组)25和蛋26数据库。有77.1%的基因分为phylotypes, 57.5%的蛋酒集群,47.0% KEGG orthology和18.7%的基因分配给KEGG通路,分别为(补充表9)。几乎所有(99.96%)的系统分配属于细菌和古菌的基因,反映出它们在肠道上的优势。基因没有映射到同源组被聚集到基因家族(见方法)。研究基因的功能内容普遍设置我们计算的总数同源组和/或基因家族的任意组合n个人(n= 2 - 124;看到图2 c)。这稀疏分析表明,“已知”功能(带注释的蛋酒或KEGG)迅速饱和(观察值5569组):当抽样的任何子集50个人,大多数都被发现。然而,四分之三的普遍肠道功能包括但一个个同源组和/或全新的基因家族(图2 c)。只包括这些团体时,稀疏曲线开始高原最后,在更高的水平上(19338组检测),确认大量的广泛的抽样个体是必要的,以抓住这大量的小说/未知的功能。
对生活在肠道细菌功能很重要
大量的冗余的人体肠道内的细菌基因提供了一个机会来识别对生活在这个环境中细菌的功能很重要。有功能所必需的一种细菌茁壮成长在肠道环境中(即“最小肠道基因”)和那些参与整个生态系统的体内平衡,编码在许多物种(肠道metagenome“微不足道”)。第一组功能预计将出现在大多数或所有肠道细菌物种;第二组在大多数或所有个人的直觉样本。
识别编码的函数最小的肠道基因组我们使用他们应该出现在大多数或所有肠道细菌物种,因此出现在基因的频率高于目录功能只出现在一些肠道细菌物种。可以推导出不同功能的相对频率的基因数量招募不同蛋集群,归一化后的基因长度和拷贝数(补充图。10 a, b)。我们排在所有的集群基因频率和确定的范围包括集群指定著名的重要细菌的功能,如确定实验的研究得到了,枯草芽孢杆菌27集群,并且假定附加在这个范围内同样重要。正如所料,包括大部分的范围枯草芽孢杆菌基本集群(86%)顶端的排名顺序(图5)。约76%的集群的重要基因大肠杆菌28是在这个范围内,确认我们的方法的有效性。这表明1244宏基因组范围内的集群发现(补充表10;称为“范围集群”以后)指定在肠道功能重要的生活。
集群被它们所包含的基因数量排名,归一化平均长度和拷贝数补充图10)和集群的比例至关重要枯草芽孢杆菌基因是决定100年连续组织集群。表示范围的一部分包含86%的集群分布枯草芽孢杆菌至关重要的基因。
我们发现两种类型的函数在集群范围:那些需要在所有细菌(管家)和潜在的特定的肠道。第一类是函数的许多示例的主要代谢途径(例如,中心碳代谢、氨基酸合成),和重要的蛋白复合物(RNA和DNA聚合酶、ATP合酶、通用分泌器)。毫不奇怪,投影的范围集群KEGG代谢途径给出了一个高度集成的全球肠道细胞代谢图6)。
一个、投影的肠道最小基因组KEGG通路使用iPath工具38。b、功能基因组组成的最小肠道和metagenome。罕见的和频繁的参考基因组测序蛋。c,估计的最小肠道metagenome大小。已知的同源组(红色),已知和未知同源组(蓝色)和同源组+新基因家族(> 20蛋白质;灰色)(见所示图2 c盒子,须情节的定义)。插图显示了构成肠道微生物组最小。大圈:分类最小metagenome根据同源组在STRING7发生39细菌基因组。常见(25%),不常见(35%)和罕见(45%)是指函数出现在> 50%,< 50%,但> 10%,<字符串细菌基因组的10%,分别。小圆:罕见的同源组组成。未知(80%)没有注释或糟糕的特征,而已知的细菌(19%)和phage-related(1%)同源组织功能描述。
假定的gut-specific功能包括那些参与粘附到宿主蛋白(胶原蛋白、纤维蛋白原、纤连蛋白)或globoseries收获糖的糖脂,进行血液和上皮细胞。此外,15%的范围集群编码函数出现在< 10%的蛋酒基因组(见补充图11),在很大程度上是(74.3%)没有定义(图6 b)。详细的研究这些应该导致肠道细菌生活的更深层次的理解。
识别编码的函数最小肠道metagenome,我们计算的同源组由个人共享我们的队列。这个最小集合,6313功能,远远大于估计在之前的一项研究8。只有2069个功能注释同源组,表明他们严重低估了真实规模的常见功能补充个体(图6 c)。最小的肠道metagenome包括相当大的一部分功能(∼45%),出现在< 10%的细菌基因组测序(图6 c插图)。否则这些罕见的功能,在每一个124人的肠道生态系统可能是必要的。百分之八十的同源组包含基因最多差特征函数,强调我们的肠道功能的知识有限。
已知的分数,代码(pro) phage-related蛋白质5%左右,这意味着噬菌体的普遍存在和可能的重要的生态作用在肠道内稳态。最引人注目的次生代谢最小metagenome关系是非常重要的,不是意外,生物降解复杂的糖类和聚糖收获从主机饮食和/或肠道粘膜。例子包括果胶的降解和吸收通路(及其单体,鼠李糖)和山梨糖醇、糖无处不在的水果和蔬菜,但是没有或者不被人体吸收。发现一些肠道微生物降解29日,30.这种能力似乎被选中的肠道生态系统作为一个非竞争性的能源。除了这些,发酵能力,例如,甘露糖、果糖、纤维素、蔗糖也是metagenome很小的一部分。在一起,这些强调肠道生态系统在复杂的强烈依赖糖降解功能。
功能基因组的互补性和metagenome
详细分析肠道metagenome之间的互补性和人类基因组是超出了目前的工作范围。概述,我们考虑两个因素:保护功能的最小metagenome和存在/没有函数在一个或另一个(补充表11)。肠道细菌使用主要发酵生成能量,转化糖,在某种程度上,短链脂肪酸,主机使用的能源来源。醋酸是重要的肌肉,心脏和大脑细胞31日丙酸,用于宿主肝neoglucogenic过程,然而,此外,丁酸对肠上皮细胞是重要的32。除了短链脂肪酸,氨基酸是人类不可或缺的33由细菌和可以提供的34。类似地,细菌可以提供某些维生素3(例如,生物素,维生素k1)主机。所有的这些分子的生物合成步骤最小metagenome编码。
肠道细菌似乎能够降低许多外源性物质,包括非转基因和卤代芳香族化合物(补充表11),即使大多数路径的步骤不是metagenome很小的一部分,只存在于一小部分人。苯甲酸是一个特别有趣的例子,这是一种常见的食品补充剂,称为E211。其辅酶a结扎通路降解,在最小metagenome编码,导致pimeloyl-coenzyme-A,生物素的前体,表明这种食物补充有可能对人类健康有益的作用。
讨论
我们使用广泛的Illumina公司GA short-read-based总粪便DNA的测序一群124人的欧洲(北欧和地中海)起源建立目录的冗余人类肠道微生物的基因。目录包含330万个微生物基因,150倍超过人类基因补,包括绝大多数基因(> 86%)的普遍怀有的队列。目录可能包含大多数普遍人类肠道微生物的基因,有以下原因:(1)超过70%的宏基因组读取从三个以前的研究,包括美国和日本人8,16,17,可以映射重叠群;(2)从89年约80%的微生物基因频繁肠道参考基因组存在在我们集合。原则上证明这个结果代表了一个短内容排序可以用来描述复杂的微生物。
每个单独的完整的细菌基因补不是我们采样的工作。尽管如此,我们发现一些536000流行独特基因,330万的由我们的队列。不可避免的是,个人主要分享共同的基因池。目前的深度测序,我们发现几乎40%的基因从每个共享至少一半的个体群。未来的研究全球跨度,设想在国际人类微生物组的财团,将完成,必要时,我们的基因样本,建立边界共享基因的比例。
几乎全部(99.1%)我们的目录的细菌基因的起源,其余大多是热点,只有0.1%的真核生物和病毒的起源。基因目录因此相当于大约1000与一般细菌物种基因组,编码约3364冗余基因。我们估计不超过15%的普遍的基因群体可能丢失的目录,并显示队列港口不超过∼1150个细菌物种丰富,足以检测到我们的抽样。鉴于大型微生物重叠序列在我们先前的研究表明,丰富的肠道细菌物种的数量不可能远高于观察我们的队列。每个单独的队列港口至少有160这样的细菌种类,平均普遍估计的基因数量,和许多因此必须共享。
我们分配12%的参考基因(404000)194肠道细菌基因组测序,因此可以将它们与细菌物种。至少1000 human-associated细菌基因组的测序中预见到国际人类微生物组的财团,通过人类微生物组计划和MetaHIT。这是符合优势种的数量我们在人类肠道的队列和预期的更广泛,而且应该让更广泛的基因物种任务。然而,我们使用了目前测序基因组进一步探索主要物种在我们群共享的概念,确定了75种常见> 50%的个人和57种常见> 90%。这些数字可能会增加测序参考菌株的数量和更深的抽样。事实上,增加2-3-fold测序深度提高25%的物种数量,我们可以发现两个人之间共享。大量的物种共享支持这样的观点,即普遍的人类微生物组是有限的而不是过于大尺寸。
如何与这个观点相当的人际无数的肠道细菌物种的多样性,因大多数以前的研究使用16 s RNA标志基因4,8,10,11吗?可能这些研究的取样深度不足以揭示常见物种当出席低丰度,并强调了不同成分的优势种相对较少。我们发现一个非常高的可变性的丰度(12 - 2200倍)57最常见的物种在我们群的人。然而,16 s rRNA-based最近的一项研究得出结论说,一个常见的细菌物种的“核心”,至少50%的人在研究之间共享,存在35。
详细比较的细菌基因的个体群将在未来,不断的上下文中MetaHIT临床研究的一部分。然而,聚类的基因在家庭允许我们捕获一个几乎普遍基因设置和显示功能的潜力相当大的新奇,扩展功能类别30%以前的工作8。同样,这种分析揭示了功能核心,守恒的每个单独的队列,它反映了人类肠道metagenome最小,编码可能在许多物种和肠道生态系统正常运转所必需的。这个最小的大小metagenome超过数倍的核心metagenome先前报道的8。它包括host-bacterial交互功能被认为是重要的,如降解复杂的多糖,短链脂肪酸的合成不可缺少的氨基酸和维生素。最后,我们还确定了功能属性最小的肠道细菌基因组,可能要求的任何细菌茁壮成长在这个生态系统。除了一般管家功能,未知函数的最小基因组包含了许多基因在基因组测序和可能特别需要在肠道。
除了提供人类肠道微生物组的全局视图,广泛的基因目录我们协会建立了使未来的研究微生物与人类表型和基因,更广泛地说,人类的生活习惯,考虑环境,包括饮食,从出生到老年。我们预计,这些研究将导致一个更完整的理解比我们目前人类生物学。
方法总结
人类粪便样本收集,立即冻结,DNA被标准方法纯化22。124人,paired-end图书馆是由不同大小的克隆插入并受Illumina公司GA测序。所有的阅读都使用SOAPdenovo组装19,与特定的参数- m 3的宏基因组数据。MetaGene用于基因预测。非冗余基因集构造成对比较的所有基因,用咩咩的叫声36身份的标准下> 95%和> 90%重叠。基因分类BLASTP的基础上进行了分配37搜索(e值< 1×105)NCBI-NR数据库和126种已知的肠道细菌基因组。基因功能注释是由BLASTP搜索(e值< 1×105蛋酒和KEGG数据库(v48.2)。同源组的总数量和共享和/或基因家族是计算使用的随机组合n个人(n= 2到124,每本100复制)。
网上的方法
人类粪便样本集合
丹麦人的国际米兰- 99组40根据BMI,不同表型对肥胖、糖尿病和地位,而西班牙人健康对照组或慢性炎性肠道疾病患者(克罗恩病和溃疡性结肠炎)在临床缓解。
患者和健康对照组被要求提供一个冰冻的粪便样本。在家新鲜粪便样品了,样品被立即冷冻储存在家里的冰箱。冷冻样本被送到医院使用绝缘泡沫聚苯乙烯容器,然后储存在-80°C到分析。
DNA提取
冰冻的整除(200毫克)的粪便样本悬浮在250μl硫氰酸胍,0.1米三羟甲基氨基甲烷(pH值7.5)和40μl 10%液N-lauroyl肌氨酸。然后,提取DNA进行了如前所述22。DNA浓度和其分子大小估计nanodrop(热科学)和琼脂糖凝胶电泳。
DNA库建设和测序
DNA库准备遵循制造商的指令(Illumina公司)。我们使用相同的工作流执行集群生成,其它地方描述的模板杂交,等温扩增,线性化、阻塞和变性和杂交测序引物。base-calling管道(illuminapipeline - 0.3版)是用于处理原始荧光图像和调用序列。
我们构建一个库(克隆插入大小200个基点)的第一个15个样品,并与不同大小的克隆插入两个库(135个基点和400个基点)为每个剩余的109个样本进行验证的实验重现性。
估计代小说之间的最优回报序列和测序深度,我们一致Illumina公司GA读从468335年样本MH0006和MH0012桑格读总计为311.7 Mb产生相同的两个样品(156.9和154.7 Mb,分别补充表2),使用短的寡核苷酸排列程序(SOAP)41和95%的匹配要求序列的身份。大约4 Gb的Illumina公司序列,94%和89%的桑格读取(分别为MH0006和MH0012)都淹没了。进一步的大规模测序,12.6和16.6 Gb MH0006和MH0012分别只适度增加覆盖率约95% (补充图1)。超过90%的桑格读取被Illumina公司序列覆盖到一个非常高的和统一的水平(补充图2),这表明有很少或没有偏见的Illumina公司GA序列。正如预期的那样,大部分Illumina公司序列(分别为57%和74% M0006和M0012)小说,不能映射到桑格读取。这个分数是4和12到16 Gb测序水平相似,确认最新奇已经被捕的4 Gb。
我们生成35.4 -.976亿读剩下的122个样本,平均6250万读。测序读第一批15个样品的长度是44 bp和第二批75个基点。
使用公共数据
基因组测序的细菌基因组(完全806)存入基因库从NCBI数据库下载(http://www.ncbi.nlm.nih.gov/2009年1月10日)。已知的人类肠道细菌基因组序列下载来自数据库(http://www.hmpdacc-resources.org/cgi-bin/hmp_catalog/main.cgi),基因库(67基因组),华盛顿大学圣路易斯(2009年4月,85个基因组,版本http://genome.wustl.edu/pub/organism/Microbes/Human_Gut_Microbiome/MetaHIT项目(17日),测序的基因组,2009年9月版,http://www.sanger.ac.uk/pathogens/metahit/)。在这个项目中使用的其他肠道metagenome数据包括:(1)人类肠道宏基因组测序数据从我们个人8,这是与入世SRA002775从NCBI下载;(2)从日本人人类肠道宏基因组数据17,从p·博克EMBL的集团(下载http://www.bork.embl.de)。我们构建的集成的NR数据库这个研究包括NCBI-NR数据库(2009年4月版)和所有从已知的人类肠道细菌基因组的基因。
Illumina公司GA短读新创组装
高质量短读的DNA样本被SOAPdenovo assembler组装19。总之,我们首先过滤低丰富的序列组装根据17-mer频率。深度小于5的17-mers筛选的大会,对于这些低频序列很可能进行组装,而删除它们将大大降低内存需求,使装配可行的在一个普通的超级计算机(512 GB的记忆在我们的研究所)。
然后序列加工,均de Bruijn图形数据格式是用于存储之间的重叠信息序列。重叠路径由一个读是不可靠和删除。短low-depth技巧和泡沫是由于测序错误或微生物菌株之间的遗传变异是修剪和合并,分别。被用来解决小重复阅读路径。
最后,我们打破了在重复连接边界,与明确的连接和输出连续序列重叠群。宏基因组的特殊模型选择和参数- k 21和23 - k”被用于44 bp和读取75个基点,分别指示所需的最小的序列重叠。
后新创大会为每一个独立样本,我们合并一起未装配的读取和执行组装,以最大化的使用数据和组装的微生物基因组低频率在每一读,但是有足够的深度序列组装一起把所有样品的数据。
使用桑格读取验证Illumina公司重叠群
我们使用BLASTN (WU-BLAST 2.0)映射桑格读取从样本MH0006 MH0012(分别为156.9 Mb和154.7 Mb) Illumina公司叠连群(最好打超过75个基点,超过95%的身份)相同的样本。每个校准扫描了破损的共线性序列都有至少50个基地左对齐的一端对齐。每一个这样的破坏被认为是一个装配误差在Illumina公司叠连群位置共线性断裂。错误在30 bp互相合并。一个错误被丢弃,如果存在一个桑格读到同意的重叠群结构60 bp两岸的错误。相比之下,我们重复这个Newbler2组装454钛读取MH0006 (550 Mb读取)。补充图5显示错误的数量/ Mb的组装Illumina公司/ 454重叠群。我们估计14.12错误Mb的叠连群Illumina公司大会,这是与454年大会(20.73 Mb)。98.7%的Illumina公司叠连群映射至少一个桑格读是共线的绘制区域的99.55%以上,相当于97.86%的454叠连群共线绘制区域的99.48%以上。
评价人类肠道微生物组报道
Illumina公司GA读取一致反对重叠群装配和已知的细菌基因组使用SOAP41最多允许两个身份不匹配第一个35-bp地区和90%的阅读顺序。罗氏公司/ 454和桑格测序读一致反对用BLASTN 1×10相同的参考890%,超过100个基点排列长度和最小单位截止。两个不匹配被允许读和身份是95%序列对齐到GA读MH0006和MH0012桑格读相同的样品用肥皂。
基因预测和建设冗余基因集
我们使用MetaGene20.——使用di-codon频率估计一个给定的序列的GC含量,并预测一系列的开放框架基于匿名的基因组序列来发现羊痘疮的重叠群的124个样本以及合并的重叠群装配。
预测的orf然后彼此对齐使用咩咩的叫声36。一对基因的身份和对齐的长度大于95%覆盖超过90%的短基因组合在一起。群体共享基因然后合并,每个合并组最长的子被用来代表集团和集团的其他成员作为冗余。因此,我们组织冗余基因集的预测基因排除冗余。最后,orf长度小于100 bp被过滤。我们翻译并使用遗传Codes11 NCBI成蛋白质序列。
识别的基因
识别low-abundance基因之间的平衡和降低识别错误率,我们设置的阈值的影响进行了探讨阅读覆盖率的要求确定一个基因在个体的微生物。的基因数量下降两倍当读取识别所需的数量从2增加到6,此后慢慢改变(补充图6)。然而,包括罕见的基因分析,我们选择2读取的阈值。
基因分类任务
预测基因的分类任务进行了使用集成BLASTP对齐对NR数据库。BLASTP对齐打击与e值大于1×105被过滤,每个基因定义的重大比赛的吗e值≤10×e价值最高的碰撞发生在区分分类群的保留。然后我们确定每个基因的分类水平最低的共同祖先(LCA)的算法实现梅根42。LCA-based算法分配基因分类单元的方式分配的分类单元的分类水平反映了基因的保护水平。例如,如果一个基因在许多物种中保存,这是分配给LCA的而不是一个物种。
基因功能分类
我们使用BLASTP搜索蛋白质序列预测基因的蛋酒数据库26和KEGG数据库24与e值≤1×105。基因被注解为支架的功能或KEGG同系物最低e价值。蛋酒数据库是一种集成的齿轮和KOG数据库。齿轮的基因注释被分为25齿轮类,和基因注释KEGG被分配到KEGG通路。
确定最小的肠道细菌基因组
冗余基因的数量分配给蛋酒集群是由基因长度和集群拷贝数(归一化补充图8)。集群被标准化的基因数量和排名的范围包括集群编码至关重要枯草芽孢杆菌基因决定,计算这些集群的比例在100年连续组织集群。分析所涉及的基因集群范围,除了iPath预测,使用KEGG和手动验证机械完整性的途径和蛋白质编码。
功能补充和最小metagenome总量的测定
我们计算的总数量和共享同源组和/或基因家族在随机的组合n个人(n= 2到124,每本100复制)。这种分析进行三组基因簇:(1)已知蛋酒同源组(也就是说,那些功能注释,排除那些条款的[Uu] ncharacteri(深圳),[Uu] nknown, (Pp) redicted或(Pp) utative发生);(2)所有蛋酒同源组;(3)所有直系同源组+基因家族由剩余的基因没有分配给两个以上类别。家庭聚集的“所有人针对所有BLASTP结果使用制程43通货膨胀因素的1.1和60的bit-score截止。
稀疏的分析
估计总基因丰富的使用100随机选择的样本估计由于内存限制。由于CV值> 0.5,chao2(经典)和冰丰富估计计算和更大的估计使用了两个(ICE)。这个样本大小的估计是3621646个基因(ICE)而年代奥林匹克广播服务公司毛(τ)3090575个基因,或85.3%。冰估计量曲线不完全饱和,(数据未显示)表明额外的样品需要被添加到实现最后一个结论性的估计。
常见的细菌核心
消除非常相似的菌株的影响和评估的存在已知微生物物种群体的个体中,我们使用650测序的细菌和古细菌基因组作为参考。一组是由932年公开的基因组,按相似性分组,使用90%的身份截止和相似性至少80%的长度。从每组只使用最大的基因组。Illumina公司读取从124个人被映射到组,对于物种分析来自同一物种的基因组分析和(不同大小> 20%)策划通过手动检查和通过使用16 s集群时可用的序列。
相对丰富的微生物基因组个体
我们独特的基因组覆盖率计算映射Illumina公司读和规范化的1 Gb的序列,为不同的测序正确的水平在不同的个体。冗余覆盖求和所有物种的细菌基因组设置为每个和每个物种的比例相对于总和计算。
物种共存的网络
155个物种的基因组覆盖率的Illumina公司读取≥1%至少在一个跨物种的皮尔森个人我们计算两两之间的相关性测序深度(丰度)在整个群124人。从11175年产生的跨物种的相关性,相关性低于-0.4或0.4以上(n= 342)使用Cytoscape在图形可视化44显示每个物种的基因组覆盖率平均图中节点的大小。
加入代码
存款数据
所有124个样本的原始Illumina公司读数据被存入EBI,根据入世ERA000116。重叠群和基因集可供下载从EMBL (http:///www.bork.embl.de/ arumugam / Qin_et_al_2010 /)和华大基因研究院(http://gutmeta.genomics.org.cn)网站。
引用
雷,r E。,Peterson, D. A. & Gordon, J. I. Ecological and evolutionary forces shaping microbial diversity in the human intestine.细胞124年837 - 848 (2006)
Backhed F。,雷,r E。,年代onnenburg, J. L., Peterson, D. A. & Gordon, J. I. Host-bacterial mutualism in the human intestine.科学307年1915 - 1920 (2005)
Hooper, l . V。,Midtvedt, T. & Gordon, J. I. How host-microbial interactions shape the nutrient environment of the mammalian intestine.为基础。启减轻。22283 - 307 (2002)
雷,r E。,Turnbaugh, P. J., Klein, S. & Gordon, J. I. Microbial ecology: human gut microbes associated with obesity.自然444年1022 - 1023 (2006)
恩伯,p . j . et al .肥胖相关的肠道微生物与增加能源产量的能力。自然444年1027 - 1031 (2006)
雷,r . e . et al .肥胖改变肠道微生物生态学。Proc。《科学。美国102年11070 - 11075 (2005)
张,h . et al .人类肠道微生物群在肥胖和胃旁路手术后。Proc。《科学。美国106年2365 - 2370 (2009)
恩伯,p . j . et al .核心肠道微生物在肥胖和精益双胞胎。自然457年480 - 484 (2009)
Zoetendal, e . G。,Akkermans, A. D. & De Vos, W. M. Temperature gradient gel electrophoresis analysis of 16S rRNA from human fecal samples reveals stable and host-specific communities of active bacteria.达成。环绕。Microbiol。64年3854 - 3859 (1998)
Eckburg, p . b . et al。人类肠道微生物菌群的多样性。科学308年1635 - 1638 (2005)
雷,r E。,Lozupone, C. A., Hamady, M., Knight, R. & Gordon, J. I. Worlds within worlds: evolution of the vertebrate gut microbiota.启Microbiol性质。6776 - 788 (2008)
帕默,C。,Bik, E. M., Digiulio, D. B., Relman, D. A. & Brown, P. O. Development of the human infant intestinal microbiota.公共科学图书馆杂志。5e177 (2007)
Riesenfeld c。,年代chloss, P. D. & Handelsman, J. Metagenomics: genomic analysis of microbial communities.为基础。启麝猫。38525 - 552 (2004)
冯·仅仅c . et al .定量系统的评估在不同环境中微生物群落。科学315年1126 - 1130 (2007)
Tringe, s . g . &鲁宾,e . m .宏基因组:环境样本的DNA测序。自然启麝猫。6805 - 814 (2005)
吉尔,s . r . et al。人类远端肠道微生物宏基因组分析。科学312年1355 - 1359 (2006)
Kurokawa, k等。一般比较宏基因组显示基因集富集在人类肠道微生物组。DNA Res。14169 - 181 (2007)
Suau, et al。直接分析的基因编码16 s rRNA从复杂的社区揭示了许多新颖的分子物种在人类肠道内。达成。环绕。Microbiol。65年4799 - 4807 (1999)
李,r·朱& H。新创组装的人类基因组大规模并行短阅读顺序。基因组Res。10.1101 / gr.097261.109(2009年12月17日)
野口勇,H。,P一个rk, J. & Takagi, T. MetaGene: prokaryotic gene finding from environmental genome shotgun sequences.核酸Res。345623 - 5630 (2006)
Colwell, r·k·估计:统计评估物种丰富度和物种样本共享,8.2版。<http://viceroy.eeb.uconn.edu/estimates> (1997)
Manichanh, et al。减少粪便微生物群的多样性在克罗恩病了一个宏基因组的方法。肠道55205 - 211 (2006)
王,X。,Heazlewood, S. P., Krause, D. O. & Florin, T. H. Molecular characterization of the microbial species that colonize human ileal and colonic mucosa by using 16S rDNA sequence analysis.j:。Microbiol。95年508 - 520 (2003)
Kanehisa, M。Goto, S。,Kawashima, S., Okuno, Y. & Hattori, M. The KEGG resource for deciphering the genome.核酸Res。32D277-D280 (2004)
Tatusov, r . l . et al .齿轮数据库:一个更新版本包括真核生物。BMC生物信息学441 (2003)
Jensen l . j .等人蛋酒:自动化建设和同源组基因的注释。核酸Res。36D250-D254 (2008)
小林,k . et al .至关重要枯草芽孢杆菌基因。Proc。《科学。美国One hundred.4678 - 4683 (2003)
爸爸,t . et al .建设大肠杆菌k - 12在坐标系,单基因敲除突变体:庆应义塾集合。摩尔。系统。医学杂志。210.1038 / msb4100050 (2006)
Dongowski G。,Lorenz, A. & Anger, H. Degradation of pectins with different degrees of esterification by叫多形拟杆菌人类肠道菌群分离。达成。环绕。Microbiol。66年1321 - 1327 (2000)
卡明斯,j . h &麦克法兰g . t .控制和人类结肠细菌发酵的结果。j:。Bacteriol。70年443 - 459 (1991)
黄,j . M。,de Souza, R., Kendall, C. W., Emam, A. & Jenkins, D. J. Colonic health: fermentation and short chain fatty acids.j .中国。杂志。40235 - 243 (2006)
哈默尔,h . m . et al .丁酸对结肠功能的作用。滋养品。杂志。其他。27104 - 119 (2008)
Elango, R。,Ball, R. O. & Pencharz, P. B. Amino acid requirements in humans: with a special emphasis on the metabolic availability of amino acids.氨基酸3719-27 (2009)
Metges, c . c的贡献微生物氨基酸,氨基酸宿主的体内平衡。j .减轻。130年1857年代- 1864年代(2000)
水龙头,j . et al。对人类肠道菌群系统的核心。环绕。Microbiol。112574 - 2584 (2009)
肯特,w . j . BLAT-the爆炸之类的校准工具。基因组Res。12656 - 664 (2002)
Altschul s . f . et al .缺口爆炸和PSI-BLAST:新一代的蛋白质数据库搜索程序。核酸Res。253389 - 3402 (1997)
Letunic,我。,Yamada, T., Kanehisa, M. & Bork, P. iPath: interactive exploration of biochemical pathways and networks.趋势生物化学科学。33101 - 103 (2008)
冯·仅仅c . et al .字符串7-recent集成和预测蛋白质相互作用的发展。核酸Res。35D358-D362 (2007)
小丘,等。基于人群的影响现有生活方式干预的变化在长期的饮食习惯:Inter99研究。上一页。地中海。47378 - 383 (2008)
李,r . et al . SOAP2:一种改进的超快的工具短读一致性。生物信息学251966 - 1967 (2009)
Huson, d . H。,Auch, A. F., Qi, J. & Schuster, S. C. MEGAN analysis of metagenomic data.基因组Res。17377 - 386 (2007)
van幅年代。通过流程模拟图聚类。博士论文,大学,乌特勒支(2000)
香农,p . et al . Cytoscape:软件环境生物分子相互作用网络的集成模型。基因组Res。132498 - 2504 (2003)
确认
我们的教职员工在深圳、北京基因组研究所的名字并不包括在作者列表,但谁导致了大规模测序的团队合作精神。研究导致这些结果已收到资金从欧洲共同体的第七框架计划(fp7/2007 - 2013): MetaHIT,赠款协议健康- f4 - 2007 - 201052,丹麦的Ole Rømer格兰特自然科学研究理事会Solexa项目(272-07-0196),中国的深圳市政府,中国国家自然科学基金(30725008)、国际科技合作项目(0806)、中国(CXB200903110066A;ZYC200903240076A),丹麦的战略研究委员会授予2106-07-0021 (Seqnet),应用医学基因组学中心和Lundbeck公司基础它是一家个性化疾病的预测、预防和护理。Ciberehd由研究院祝您健康卡洛斯三世(西班牙)。我们还要感谢x王从生物科学与生物工程学院,华南理工大学,因为他的本科生创新计划协调J.L.周益春加入。
作者的贡献所有作者是人类肠道的宏基因组的成员(MetaHIT)财团。So.L。,H.Y., Je.W., J.D., F.G., K.K., O.P., S.B., J.P., Ji.W., S.D.E. and Ju.W. managed the project. T.N., T.H. and K.S.B. performed clinical analyses; F.L. and C.M. performed DNA extraction. X.Z., B.W., J.C., H.L., Hu.Z., K.T., D.L.P., E.P. and M.J. performed sequencing. Ju.W., S.D.E, P.B., R.L., J.R., M.A. and J.Q. designed the analyses. J.Q., Sha.L., D.L., J.L., J.X., Y.X., Ho.Z., M.B., H.B.N., T.S.-P., C.Y., She.L., T.Y., N.P., J.-M.B., P.L., D.R.M., S.D.E. and Y.Z. performed the data analyses. S.D.E., P.B., J.R., J.Q., R.L. and Ju.W. wrote the paper. J.T., A.L., P.R., Y.L. and N.Q. revised the paper. The MetaHIT Consortium members contributed to design and execution of the study.
作者信息
作者和联系
财团
相应的作者
权利和权限
本文根据分布Creative Commons Attribution-Non-Commercial-Share都许可证(http://creativecommons.org/licenses/by-nc-sa/3.0/),它允许分布和繁殖在任何媒介,被认为提供了原作者和来源。这个许可证不允许商业开发,下许可的派生作品必须相同或相似的执照。
关于这篇文章
引用这篇文章
秦,J。,Li, R., Raes, J.et al。人类肠道微生物基因目录建立了宏基因组测序。自然464年59 - 65 (2010)。https://doi.org/10.1038/nature08821
收到了:
接受:
发行日期:
DOI:https://doi.org/10.1038/nature08821
进一步的阅读
眼睑的微生物和颊uveitic青光眼患者
BMC眼科(2022)
慢性腹泻的肠道微生物组和抗生素resistome恒河猴(解剖)及其对人类肠道微生物组相似
微生物组(2022)
阐明生物性别差异和microbiota-gut-brain轴:全面审查的角色在神经精神障碍
生物学上的性别差异(2022)
在结肠炎大鼠肠道微生物组的改变在双边Tianshu穴位艾灸
BMC胃肠病学(2022)
广泛的宏基因组分析的猪肠道resistome识别指标反映抗菌素耐药性
微生物组(2022)