文摘
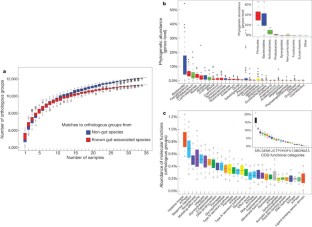
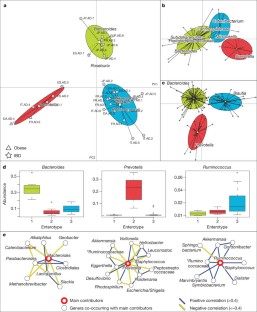
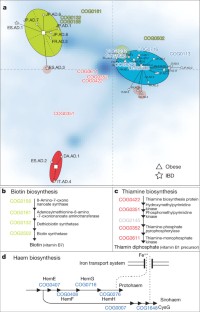
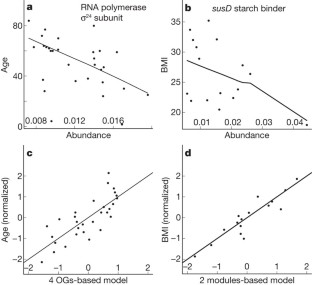
我们所知的人类肠道微生物的种类和功能成分迅速增加,但它仍然是基于很少的人群和对世界各地的变化。结合22新粪便基因组测序的个体从四个国家以前公布的数据集,我们确定三个强大的集群(称为菌群以后)没有特定国家或大陆。我们也证实了菌群在两个出版,更大的群组研究中,表明肠道菌群变化通常分层,不是连续的。这表明进一步的存在有限数量的平衡host-microbial共生状态,可能对饮食和药物的反应不同的摄入量。菌群主要是由物种组成,但丰富的分子功能并不一定提供了丰富的物种,突出的重要性理解微生物群落的功能分析。虽然单个主机属性如身体质量指数、年龄、或性别无法解释观察到的菌群,数据驱动的标记基因或功能模块为每个主机属性可以被识别。例如,12个基因明显与年龄和身体质量指数和三个功能模块,暗示一个诊断微生物标记的潜力。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
粪便微生物群组成影响体外发酵黑麦、燕麦、小麦面包
科学报告开放获取2023年1月3日
相关的粪便微生物生化概要文件在更年期期间:巴西队列研究
BMC女性健康开放获取2022年12月06
综合文献和可视化分析研究粪便微生物移植从2000年到2021年出版
生物医学工程在线开放获取2022年10月29日
访问选项
订阅杂志
得到完整的期刊访问1年
199.00美元
只有3.90美元的问题
所有价格是净价格。
增值税将被添加后在付款。
税计算期间将完成付款。
买条
时间有限或全文访问ReadCube。
32.00美元
所有价格是净价格。
改变历史
2011年6月08
一个作家被省略了。他的名字已经被添加到HTML和PDF和附带的勘误表中描述。
引用
Eckburg, p . b . et al。人类肠道微生物菌群的多样性。科学308年1635 - 1638 (2005)
Hayashi, H。,Sakamoto, M. & Benno, Y. Phylogenetic analysis of the human gut microbiota using 16S rDNA clone libraries and strictly anaerobic culture-based methods.Microbiol。Immunol。46535 - 548 (2002)
躺,c . et al .结肠微生物群签名在五个北欧国家。达成。环绕。Microbiol。71年4153 - 4155 (2005)
吉尔,s . r . et al。人类远端肠道微生物宏基因组分析。科学312年1355 - 1359 (2006)
恩伯,p . j . et al .核心肠道微生物在肥胖和精益双胞胎。自然457年480 - 484 (2009)
Kurokawa, k等。一般比较宏基因组显示基因集富集在人类肠道微生物组。DNA Res。14169 - 181 (2007)
Zoetendal, e . G。,Rajilic-Stojanovic, M. & de Vos, W. M. High-throughput diversity and functionality analysis of the gastrointestinal tract microbiota.肠道571605 - 1615 (2008)
秦,j . et al。人类肠道微生物基因目录建立了宏基因组测序。自然464年59 - 65 (2010)
Raes, j . &博克,p .分子生物学生态系统:对社区功能的理解。启Microbiol性质。6693 - 699 (2008)
纳尔逊,k . e . et al。人类微生物组参考基因组的一个目录。科学328年994 - 999 (2010)
MetaHIT财团。桑格研究所MetaHIT草案细菌基因组。<http://www.sanger.ac.uk/resources/downloads/bacteria/metahit/>(2010年7月9日)
穆勒,j .等人蛋酒v2.0:扩展基因的进化谱系与增强非同源组、种类和功能注释。核酸Res。38D190-D195 (2010)
帕默,C。,Bik, E. M., Digiulio, D. B., Relman, D. A. & Brown, P. O. Development of the human infant intestinal microbiota.公共科学图书馆杂志。5e177 (2007)
水龙头,j . et al。对人类肠道菌群系统的核心。环绕。Microbiol。112574 - 2584 (2009)
Jensen l . j . et al .字符串8 630年全球对蛋白质及其功能相互作用的生物。核酸Res。37D412-D416 (2009)
Dethlefsen, L。针对S。,Sogin, M. L. & Relman, D. A. The pervasive effects of an antibiotic on the human gut microbiota, as revealed by deep 16S rRNA sequencing.公共科学图书馆杂志。6e280 (2008)
沃克,答:我们的小的朋友问好。启Microbiol性质。5572 - 573 (2007)
Krogfelt, k .细菌粘附:遗传学、生物起源,作用在发病机理的fimbrial丰富大肠杆菌。启感染。说。13721 - 735 (1991)
Salonen, a . et al .粪便DNA提取方法的比较分析与系统发育微阵列:有效恢复使用机械细胞裂解细菌和古细菌的DNA。j . Microbiol。方法81年127 - 134 (2010)
Rajilic-Stojanovic, m . et al。人类肠道的芯片,开发和应用的系统芯片:分析普遍保守phylotypes丰富的微生物群的年轻人和老年人。环绕。Microbiol。111736 - 1751 (2009)
Rousseeuw, p . j .轮廓:图形援助聚类分析的解释和验证。j .第一版。达成。数学。20.53 - 65 (1987)
Vanhoutte, T。驾车,G。,Brandt, E., d & Swings, J. Temporal stability analysis of the microbiota in human feces by denaturing gradient gel electrophoresis using universal and group-specific 16S rRNA gene primers.《。生态。48437 - 446 (2004)
Tannock, g . w . et al .粪微生物区系的分析人体摄入益生菌产品包含乳杆菌DR20。达成。环绕。Microbiol。66年2578 - 2588 (2000)
Seksik, p . et al .改变占主导地位的粪便细菌组患者结肠的克罗恩病。肠道52237 - 242 (2003)
Costello, e . k . et al .细菌社区人体栖息地的变化在时间和空间。科学326年1694 - 1697 (2009)
Martens e . C。,Koropatkin, N. M., Smith, T. J. & Gordon, J. I. Complex glycan catabolism by the human gut microbiota: the Bacteroidetes Sus-like paradigm.生物。化学。284年24673 - 24677 (2009)
赖特,d . P。,Rosendale, D. I. & Roberton, A. M.普氏菌酶参与粘蛋白低聚糖降解和证据为一小操纵子基因表达了粘蛋白在增长。《。列托人。190年73 - 79 (2000)
Derrien, M。,Vaughan, E. E., Plugge, C. M. & de Vos, W. M.Akkermansia muciniphila11月,11月,将军sp.人类肠道mucin-degrading细菌。Int。j .系统。另一个星球。Microbiol。541469 - 1476 (2004)
雷,r E。,Turnbaugh, P. J., Klein, S. & Gordon, J. I. Microbial ecology: human gut microbes associated with obesity.自然444年1022 - 1023 (2006)
Schwiertz, a . et al .微生物群和SCFA精益和超重的健康受试者。肥胖18190 - 195 (2009)
游艇e . j .肠道细菌和老化。j:。Microbiol。102年1178 - 1186 (2007)
Kovacikova, g . &感知外界,k替代西格玛因子σE在肠道生存和毒性中起着重要的作用霍乱弧菌。感染。Immun。70年5355 - 5362 (2002)
Fujihashi, k & Kiyono h粘膜免疫衰老:新发展和控制传染病的疫苗。Immunol趋势。30.334 - 343 (2009)
恩伯,p . j . et al .肥胖相关的肠道微生物与增加能源产量的能力。自然444年1027 - 1031 (2006)
Raes, J。,Korbel, J. O., Lercher, M. J., von Mering, C. & Bork, P. Prediction of effective genome size in metagenomic samples.基因组医学杂志。8R10 (2007)
吉布森,g . r . et al .替代途径在发酵过程中氢处理的人类结肠。肠道31日679 - 683 (1990)
Godon, J·J。,Zumstein, E., Dabert, P., Habouzit, F. & Moletta, R. Molecular microbial diversity of an anaerobic digestor as determined by small-subunit rDNA sequence analysis.达成。环绕。Microbiol。63年2802 - 2813 (1997)
Arumugam, M。,Harrington, E. D., Foerstner, K. U., Raes, J. & Bork, P. Smash Community: a metagenomic annotation and analysis tool.生物信息学262977 - 2978 (2010)
王,问。,Garrity, G. M., Tiedje, J. M. & Cole, J. R. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy.达成。环绕。Microbiol。73年5261 - 5267 (2007)
Gianoulis, t . a . et al .量化环境适应宏基因组的代谢途径。Proc。《科学。美国106年1374 - 1379 (2009)
确认
作者感谢c·克里维,g . Falony和EMBL的博克小组的成员讨论和援助。我们感谢EMBL的核心设施和y元管理高性能计算资源。研究导致这些结果已收到资金从欧洲共同体的第七框架计划(fp7/2007 - 2013): MetaHIT,赠款协议健康- f4 - 2007 - 201052, EMBL, Lundbeck公司基础它是一家应用医学基因组学中心的个性化疾病的预测、预防和护理(LuCAMP),诺和诺德公司基金会和中国国际科技合作项目(0806)。肥胖和非肥胖志愿者的招募MicroObes SU.VI。马克斯队列研究协调由p·加兰和s . Hercberg和metagenome排序是由国家de la矫揉造作的(ANR);MicroAge研究招募的志愿者从CROWNALIFE队列研究协调Silvi和a . Cresci和metagenome排序由GenoScope资助。Ciberehd由de Salud研究所卡洛斯三世(西班牙)。jr研究所支持科学研究和创新的鼓励布鲁塞尔(ISRIB)和奥德修斯项目的科研基金的弗兰德斯(FWO)。我们感谢人类微生物组计划生成参考基因组从人类肠道微生物与国际人类微生物组的财团进行讨论和交换的数据。
作者信息
作者和联系
财团
贡献
所有作者是人类肠道的宏基因组的成员(MetaHIT)财团。小君W。,F.G.,开口保险单,W.M.d.V., S.B., J.D., Jean W., S.D.E. and P.B. managed the project. N.B., F.C., T.H., C.M. and T. N. performed clinical analyses. M.L. and F.L. performed DNA extraction. E.P., D.L.P., T.B., J.P. and E.U. performed DNA sequencing. M.A., J.R., S.D.E. and P.B. designed the analyses. M.A., J.R., T.Y., D.R.M., G.R.F., J.T., J.-M.B., M.B., L.F., L.G., M.K., H.B.N., N.P., J.Q., T.S.-P., S.T., D.T., E.G.Z., S.D.E. and P.B. performed the analyses. M.A., J.R., P.B. and S.D.E. wrote the manuscript. M.H., T.H., K.K. and the MetaHIT Consortium members contributed to the design and execution of the study.
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
额外的信息
生桑格读取数据从欧洲粪便基因组已经存入NCBI跟踪档案使用以下项目标识符:MH6 (33049), MH13 (33053), MH12 (33055), MH30 (33057), CD1(33059)、张(33061),UC4 (33113), UC6(33063),第一(33305),3号(33307)4号(33309年),NO8 (33311), OB2 (33313), OB1 (38231), OB6 (38233), OB8 (45929), (63073), (63075), C (63077)、D (63079), E (63081), G (63083)。叠连群,基因和注释是可供下载http://www.bork.embl.de/Docu/Arumugam_et_al_2011/。
权利和权限
关于这篇文章
引用这篇文章
Arumugam, M。Raes, J。,Pelletier, E.et al。人类肠道微生物菌群。自然473年,174 - 180 (2011)。https://doi.org/10.1038/nature09944
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature09944
本文引用的
土壤微生物和健康
自然评论微生物学(2023)
粪便微生物群组成影响体外发酵黑麦、燕麦、小麦面包
科学报告(2023)
机理的理解改变药物药物动力学的肠道微生物群
医药杂志》上的调查(2023)
奥利司他,竞争性的脂肪酶抑制剂作为典型药物疗法,能提高肠道炎症反应
应用生物化学(2022)
多糖的叶黄精属sibiricum红色。调节肠道微生物群,影响小鼠的短链脂肪酸的生产
AMB表达(2022)
