文摘
我们的肠道微生物群港口不同细菌社区需要对我们的健康,营养和健康1,2。肠道殖民始于出生,高潮迭起,与两个主导的收购集团严格的厌氧细菌属于厚壁菌门和拟杆菌门2。文化无关,基因组方法已经改变了我们对人类微生物组的角色的理解健康和许多疾病1。然而,由于普遍认为我们的土著细菌主要是顽固的文化,他们的许多功能和表型仍然未知3。在这里,我们描述一个小说工作流基于表型培养目标与大规模的全基因组测序,系统发育分析和计算模拟,表明大部分可耕种的肠道细菌。将这种方法应用于健康人,我们从特征和候选人小说的家庭分离出137种细菌,属物种被存档为纯粹的文化。全基因组和宏基因组测序,结合计算和表型分析,表明至少50 - 60%的细菌属一个健康的肠道微生物群的个体产生弹性孢子,专门用于主机到主机传输。我们的方法打开人类肠道微生物群的表型分析和揭示了如何显著比例的oxygen-sensitive肠道细菌可以人与人之间的传播,影响微生物群遗传。
主要
典型的人类肠道微生物群包含100 - 1000细菌物种个体间巨大的成分的多样性,这样每个人的指纹一样独特的微生物群1,4。尽管分类学多样性、宏基因组测序已经强调,health-associated肠道微生物组编码高度保守的基因家族和途径与基本细菌生理和生长有关2。然而,许多基本的微生物群功能相关的体内平衡,免疫系统开发、消化、抗病原体和微生物群继承尚未被发现5。这个艰巨的挑战来验证和解释功能微生物群的属性被阻碍,因为大多数肠道细菌被广泛认为是“unculturable”,从来没有被隔离在实验室3,6。
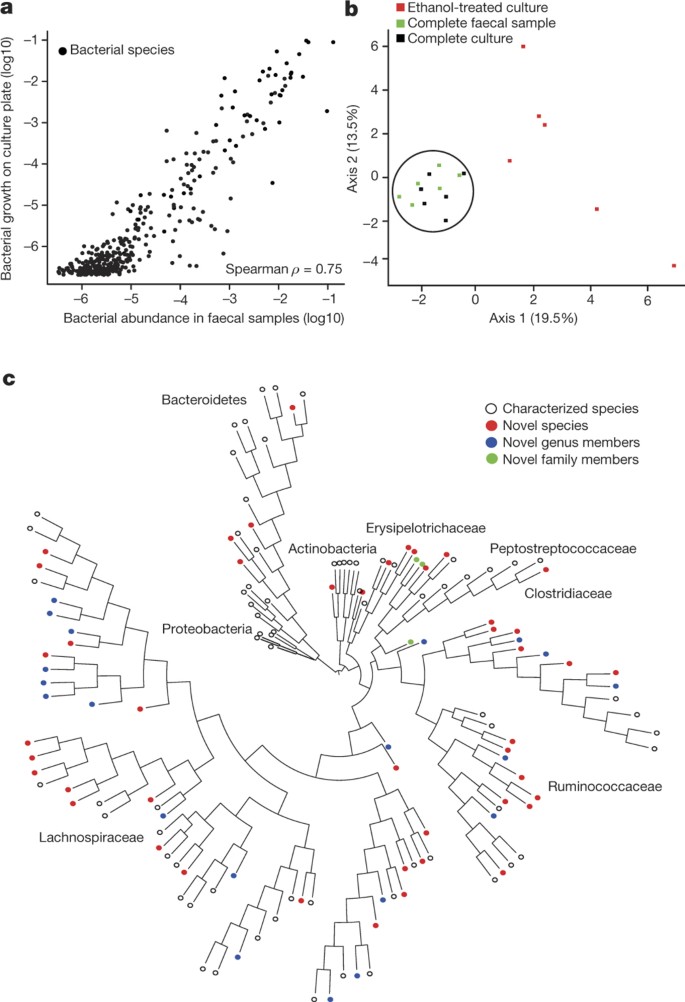
我们试图建立一个genomic-based工作流可以作为一个平台,有针对性的培养特定的细菌的表型(扩展数据图1)。因此,我们收集了六个健康的新鲜粪便样本人类和定义了细菌社区居民与宏基因组测序和细菌培养的方法。应用猎枪宏基因组测序,我们描述和比较细菌物种呈现在原始的粪便样本,增长不同的殖民地在琼脂板包含复杂,广泛细菌学的媒介,YCFA7。重要的是,我们观察到一个强大的物种的两个样本之间的相关性(枪兵的水平ρ= 0.75,P< 0.01)(图1一个)。测序时,最初的粪便样本和培养细菌社区共享的平均93%的原始读取六个捐助者。这个重叠是72%新创大会扩展数据图2)。比较全面的基因目录所派生的文化无关意味着从318人的肠道菌群4更大的数据库中发现39.4%的基因都代表我们的队列和73.5%的741计算导出宏基因组物种通过这种分析也确定培养样本检测。
一个相对大量的粪便样品中细菌(x轴)相比,相对丰富的细菌生长在YCFA琼脂板(y轴),由宏基因组测序。细菌生长在YCFA琼脂是代表完整的粪便样本的枪兵ρ= 0.75 (n= 6)。b、主成分分析的16 s rRNA基因序列从六个捐赠者粪便样本中发现(n= 6),代表完整的粪便样品中细菌(绿色),粪便细菌菌落恢复YCFA琼脂板没有乙醇预处理(黑色)或用乙醇预处理选择ethanol-resistant孢子形成的细菌(红色)。培养没有乙醇的选择是代表完整的粪便样本,乙醇治疗转变形象,丰富ethanol-resistant孢子形成的细菌,并允许他们随后的隔离。c,系统发育树的细菌培养六个捐助者由全长16 s rRNA基因序列。新型候选物种(红色),属(蓝色)和家庭(绿色)所示的点的颜色。主要类群和家庭的名字。变形菌门没有培养,但包括上下文。
总之,这些结果表明,相当比例的粪便中的细菌可以与单个生长培养基培养的微生物群。然而,超过8×106不同的殖民地需要从YCFA琼脂板与宏基因组测序的物种的检测灵敏度。因此,我们建立了一个广泛培养方法,结合高通量存档或特定表型选择时,可以用来分离和识别小说从胃肠道细菌。
人类肠道微生物群是由严格的厌氧细菌,对周围的氧气非常敏感,所以不知道如何将这些细菌生存环境暴露在个体间传播。厚壁菌门的某些成员门,包括腹泻病原艰难梭状芽胞杆菌殖民期间,产生新陈代谢休眠和高度耐孢子能同时促进持久性宿主和环境中的传播8,9,10。相对很少有肠道孢子形成的细菌培养到目前为止,虽然宏基因组研究表明其他意想不到的肠道微生物群的成员拥有潜在的孢子形成基因,这些细菌特征仍然不佳11,12,13,14。
我们假设孢子形成是一个未被欣赏的基本人类肠道微生物群的表型可能对微生物群产生深远影响持久性和人类之间传播。孢子从梭状芽孢杆菌对乙醇和这种表型可以选择用于孢子混合人口的孢子和ethanol-sensitive营养细胞15。粪便样本有或没有乙醇治疗处理使用我们的结合文化和宏基因组工作流(扩展数据图1)。主成分分析表明,乙醇治疗可耕种的细菌组成和发生了深刻的变化,相比原来的形象,有效地丰富ethanol-resistant细菌,促进他们的隔离(图1 b)。我们选择~ 2000个体细菌菌落ethanol-treated和non-ethanol-treated条件,re-streaked他们纯洁,并执行完整的16 s rrna启用分类特征基因测序。独特的类群被存档冻结的股票未来的表型分析。
总的来说,我们存档细菌占96%的细菌丰度在属级和90%的细菌丰度在物种水平基于平均相对丰度在六个捐助者(扩展数据图3 a, b)。甚至属出席平均相对丰度较低(< 0.1%)被孤立的(扩展数据图3 c)。总的来说,我们存档的137种不同的细菌包括45候选人小说物种(图1 c,扩展数据图3 d和补充表1),隔离代表20候选人小说属2候选人小说的家庭。我们集合包含90种人类微生物组项目的“通缉犯”名单之前未耕作的和unsequenced微生物16(补充表1)。因此,我们广泛YCFA-based培养方法导致大量细菌的发现,和挑战的概念,大多数unculturable肠道菌群。
我们分离和纯化细菌代表66种不同ethanol-resistant物种分布在5知道家庭和2新发现候选人家庭(扩展数据图3 d和扩展数据图4)。识别这些新的和意外spore-formers亮点的广泛的分类分布表型在肠道壁厚菌门的物种。定义中的守恒的基因通路基础孢子形成和萌发肠道微生物群,我们测序,组装和注释的整个基因组234存档ethanol-resistant ethanol-sensitive细菌。以前,这种基因标记用于识别孢子形成细菌物种都是基于潜在基因的假设13,17,18;这里我们应用客观的计算方法来定义66守恒和一个ethanol-resistance有关的基因表型(扩展数据图5和补充表1)。这组基因允许预测的孢子形成细菌物种隔离来自不同环境的能力,高度的准确性(扩展数据图6和补充表1),由基因从一个广泛的功能类(扩展数据图6 b和补充表1)。
测试是否共生的孢子形成促进长期的环境生存,我们暴露了过去的同桌的孢子形成和non-spore-forming细菌和多样化的选择梭状芽孢杆菌为增加时间周围的氧气。在这种情况下,non-spore-forming细菌仍然可行的2 - 6天(48 - 144 h) (图2一个)。相比之下,同桌的孢子形成的细菌,梭状芽孢杆菌和兼性厌氧菌大肠杆菌能够稳定地生存实验的最后21天(504小时)。此外,孢子形成的共生体梭状芽孢杆菌,但不是non-spore-forming共生体,幸存下来的长时间暴露于公共消毒剂乙醇(扩展数据图7所示)。这些结果说明共生体spore-formers和梭状芽孢杆菌分享一组核心的孢子形成基因赋予一个高度耐药表型与环境相关联在人类之间传播。
一个,比non-spore-formers aero-tolerant Spore-formers,预计可以实现主机到主机的信息传播。一旦暴露于氧气,只有1%的原培养液non-spore-forming 96 h后细菌(虚线)是可行的(4天)和144 h后没有一个可行的(6天)。孢子形成的细菌(实线)持续由于孢子的形成。实验504 h(21天)后停止。分类的家庭每一物种测试显示在方括号(n为每个应变)= 3生物复制。b肠道spore-formers回应胆汁酸发芽的。菌落的数量(c.f.u)。(表示发芽孢子)出现在盘子的存在表示为一个特定的开端的褶皱变化对c.f.u.恢复的数量在盘子里没有发芽的。Spore-formers和non-spore-formers受到乙醇冲击镀前(n为每个应变)= 6生物复制。只有spore-formers幸存下来。的褶皱变化(虚线)表明,发芽的没有影响c.f.u.恢复的数量。示意图总结了cholate-derived哺乳动物肠道中胆汁酸代谢。意味着和范围,韦尔奇的未配对的双尾t以及(*P< 0.05,* *P< 0.01,* * *P< 0.001,* * * *P< 0.0001)。
梭状芽孢杆菌孢子进化机制恢复新陈代谢和营养生长发芽后肠道殖民的反应消化胆汁酸释放到小肠的胆囊9。我们接触肠spore-formers和non-spore-formers胆酸(牛磺胆酸盐,glycocholate和胆盐)来评估他们的反应后发芽的ethanol-shock治疗(图2 b)。牛磺胆酸盐是一种强有力的开端的spore-formers,增加从共生的细菌孢子的culturability(8 -至70000倍P所有spore-formers测试< 0.05),而其他的胆盐衍生物有不同功效的发芽共生体spore-formers (图2 b)。牛磺胆酸盐和其他胆汁酸non-spore-formers culturability没有影响,证明效果是特定于spore-formers (扩展数据图8所示)。我们建议这个bile-acid-triggered殖民萌发的机理是守恒的在活的有机体内提示促进肠道孢子形成的细菌的入侵。因此,存在一个二元性的目的做法肠道细菌孢子形成的;孢子形成确保他们的生存和传播而萌发的回应在活的有机体内线索可以确保他们坚持人类的人口。
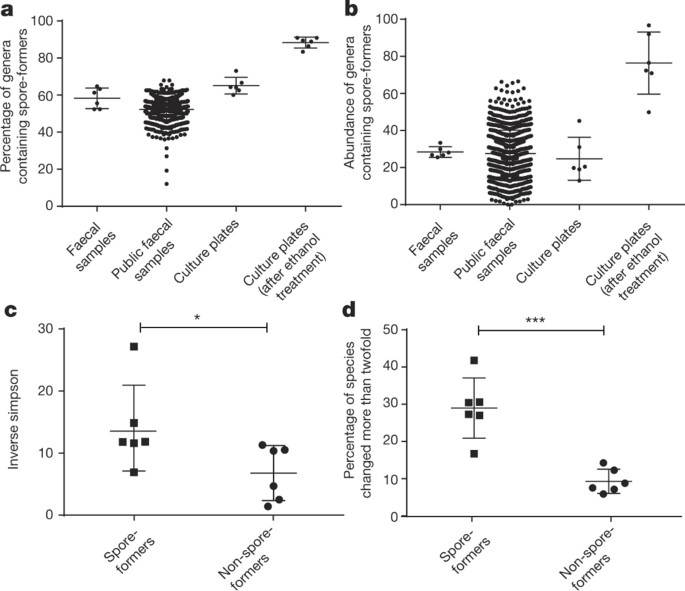
我们下一个试图估计孢子形成的细菌在肠道菌群的比例。审讯孢子基因签名的宏基因组数据集的预测,平均60%的属包含孢子形成的细菌(图3)。肠道菌群(这些属代表总数的30%图3 b)。我们独立验证这些观察与16 s rRNA基因扩增子测序(扩展数据图9所示)。重要的是,这些孢子形成的细菌的比例也观察到在1351年公开的粪便从健康个体生成的宏基因组数据集19(图3 a, b)。我们还发现同样比例的spore-formers(61.3%)在“宏基因组物种”源自318年健康个体4。
一个,b使用基因签名询问公共(n= 1351)和完整的粪便样本宏基因组数据集从这个研究(n= 6)显示的比例spore-formers数总数的属(一个)和总微生物丰度(b)。c,d宏基因组测序的捐赠者粪便样本(n= 6)1年之后表明,孢子形成的细菌比non-spore-forming更多样的细菌(c),比例显著增加显示两个或更多的物种变化在同一时期(d)。平均值±标准偏差(其中),双尾配对t以及(*P< 0.05,* * *P< 0.001)。
而肠道菌群被认为是相对稳定的20.家庭成员的密切接触,证据表明,促进共享Ruminococcaceae和Lachnospiraceae细菌21家庭,我们描述spore-formers (扩展数据图4)。我们注意到在我们的群体中,孢子形成的细菌微生物群的多样性明显多于non-spore-forming细菌(图3 c)。孢子形成的动态测试和non-spore-forming细菌随着时间的推移,我们分析了宏基因组资料收集的粪便样本相同的健康受试者一年后原始采样。有趣的是,我们注意到变化的显著增加孢子形成的细菌的比例与non-spore-forming细菌在此期间。这意味着更高的物种周转率或更大的转变在孢子形成细菌物种相对多度(图3 d)。综上所述,我们的表型和基因分析表明,孢子形成和non-spore-forming细菌代表专业,不同的微生物群的表型组件,每个国家都有独特的殖民动力学。
我们表明,芽孢形成普遍,尽管此前未被欣赏的人类肠道微生物群的函数,与微生物群的传播和继承的重要意义。的基础上共同的发展史和共同进化和孢子形成和萌发的表型特征,我们认为丰富的共生体肠道spore-formers确定这里依靠相同的传输和殖民策略梭状芽孢杆菌22。总之,环境梭状芽孢杆菌孢子是高度传染性长时间后,通常在当地环境中传输也有可能快速远距离传播23。传输动力学和地理范围的共生体spore-formers尚未确定,但我们预计,这种类型的信息将提供伟大的见解遗传和形状的选择性因素人类肠道菌群的组成。
我们的工作流支持大规模培养,归档,基因组测序和小说细菌的表型出现人类肠道微生物群,以前认为是unculturable。我们有相当whole-genome-sequence生成数据集对应于肠道细菌基因组的总数的39%由人类微生物组计划生成。我们的流线型,单一交易媒介的方法,建立在别人的相当大的努力24,25和打开人类肠道微生物群的表型鉴定。
方法
培养
新鲜粪便样本来自六的健康成年人捐助者(1粪便样本/捐助:最低0.5 g)和1 h内被放置在厌氧条件下通过保持厌氧细菌的可行性。所有样品处理和厌氧条件下培养发生在惠特利DG250工作站在37°C。文化媒体、PBS和所有其他材料用于培养被安置在厌氧内阁24小时使用前降低厌氧条件。粪便样本被一分为二。均质在减少一部分PBS(0.1克每毫升凳子PBS)和连续稀释,直接到YCFA镀7与0.002克毫升琼脂补充−1每个葡萄糖,麦芽糖和纤维二糖在大培养皿(直径13.5厘米)。这个示例还受到宏基因组测序整个社区。另一部分是处理同等体积的70% (v / v)乙醇4 h在室温环境有氧条件下杀死营养细胞。然后,固体材料与PBS洗了三次,最终在PBS resuspended。镀了如前所述。
ethanol-treated样本,媒介是补充钠0.1%牛磺胆酸盐刺激孢子萌发。殖民地了72 h从培养皿ethanol-treated和镀后non-ethanol-treated条件窝藏non-confluent增长,(也就是说,盘子的殖民地是截然不同的,而不是触摸)。选的殖民地被re-streaked证实纯度。没有统计方法被用来预先确定样本容量。实验并不是随机的。调查人员没有被分配在实验和结果评估。
微生物群分析和排序
识别每个隔离是由PCR扩增全长16 s rRNA基因(使用7 f (5′-AGAGTTTGATYMTGGCTCAG-3′)正向引物和1510 r (5′-ACGGYTACCTTGTTACGACTT-3′)反向引物毛细管测序紧随其后。全长16 s rRNA基因序列读取对齐在核糖体数据库项目(RDP),手动在ARB策划26和mothur27被用来读取操作分类单位(辣子鸡)进行分类。R包seqinr 3.1版本是用于确定序列相似性辣子鸡和98.7%被用作了解截止28,29日。每个了解的全长16 s rRNA基因序列OTU与RDP参考数据库分配属级分类名称30.和BLASTn搜索定义特征或候选人小说的物种31日。
与人类微生物组计划(HMP)进行了使用97%序列相似性的16 s rRNA培养细菌的基因序列来定义一个物种,因为只有部分16 s rRNA基因序列。HMP数据关于通缉类群和测序项目下载完成http://hmpdacc.org/most_wanted/的数据和http://hmpdacc.org/HMRGD/,分别。
基因组DNA提取从至少一个代表每一个独特的OTU使用phenol-chloroform-based DNA分离过程。DNA测序Illumina公司HiSeq平台上生成读长度为100个基点,这些都是组装和注释进行进一步分析。粪便样本中直接提取的DNA为整个社区宏基因组和16 s rRNA使用MP基因扩增子测序生物医学FastDNA旋转工具的土壤。能够比较完整的社区样本,non-confluent文化从琼脂板刮72 h与最初的粪便样本和接种后从这个社区使用相同的DNA提取的DNA分离过程。16 s rRNA基因扩增子库是由PCR扩增的可变区域1和2的16 s rRNA使用Q5高保真聚合酶基因工具箱提供的新英格兰生物学实验室。引物27 f AATGATACGGCGACCACCGAGATCTACAC(第一部分,Illumina公司适配器)TATGGTAATT(第二部分,向前引物垫)CC(第三部分,向前底漆链接器)AGMGTTYGATYMTGGCTCAG(第四部分,正向引物)和338 r CAAGCAGAAGACGGCATACGAGAT(第一部分,反向互补的3′Illumina公司适配器)ACGAGACTGATT(第二部分,戈利条码)AGTCAGTCAG(第三部分,反向引物垫)AA(第四部分,反向引物链接器)GCTGCCTCCCGTAGGAGT(第五部分,反向引物)。4每样品进行PCR扩增反应;产品被汇集并结合克分子数相等的金额使用Illumina公司MiSeq测序平台,生成150个基点。
微生物群分析
的最大似然发展史而产生的细菌是对齐的RDP序列使用FastTree 2.1.3版本(ref。32)使用以下设置:广义倒流(GTR)模型的核苷酸替换和猫近似率变化的跨站点与20个类别。ethanol-resistant发展史是直接来自整个文化发展史。所有系统发育树在ITOL编辑33。
分析部分16 s rRNA基因序列产生的16 s rRNA基因进行了扩增子库使用mothur MiSeq SOP342014年8月29日,生成7549辣子鸡所有样本。97%的序列相似性阈值被用来定义一个OTU。
宏基因组序列读取分析使用挪威海怪35分类基于自定义数据库的序列分类方法包括完整、高质量的参考细菌,病毒和古细菌基因组DNA除了基因组测序研究。分类结果读取日志2改变总丰度和标准化。宏基因组样本相比在属和种的水平和相对丰富的在基因水平对齐使用bowtie2算法36到适当的目录的基因。序列被认为是目前实现平均双重的覆盖在考虑序列的长度。截止100独特的读取应用于确定宏基因组物种检测。在适当情况下,斯皮尔曼等级相关系数是申请相关分析。逆辛普森多样性指数计算从巨妖输出R版本3.2.1使用素食:社区生态包版本2.3 0。
孢子形成的基因签名
基于启发式双向最佳冲击分析确定21342年保守基因内的694300个基因在234测序的基因组注释。基于机器的支持向量,对比组关联挖掘应用于确定最优,加权66个基因组成的基因签名。使用BLAST-based物种分类进行基因检测的检测比例加权签名贡献和扩展生成一个总分在0和1之间。分数超过0.5被认为是真正的spore-formers基于已知spore-formers相比。基于签名的丰度评估反对1351年公开可用的宏基因组数据集从健康人19挪威海怪后分类任务使用数据库。属被认为spore-formers当所有已知物种内,属孢子形成得分大于0.5。
透射电子显微镜法
孢子图像生成使用透射电子显微镜(TEM)如前所述37。细菌分离成像是由裸奔纯文化从冷冻甘油股票和后,全长16 s rRNA基因测序证实纯度一轮获得可见的文化孤立单一的殖民地。TEM图像在接种后准备从文化板块72 h。孢子体的数量明显在TEM图像表示为一个百分比的营养细胞的数量现在和这个范围从1%瘤胃球菌属flavefaciens_93% 4%Turicibacter杂志。
氧气敏感性分析
纯粹的文化是在YCFA汤在厌氧培养条件如前所述,文化被发现在一系列稀释到YCFA包含0.1%牛磺胆酸盐钠琼脂。盘子被孵化环境(有氧)条件下室温为指定的时间之前回到了厌氧内阁。菌落(c.f.u。) 72 h后计算。文化,是孵化厌氧,因此能不暴露于氧气,作为对照组。试验前,所有物种受到乙醇冲击和厌氧培养以确定他们形成孢子的能力。oxygen-exposed文化的可行性被表示为一个百分比的可行性厌氧控制文化。
萌发对肠道胆汁酸测定
纯文化是生长在YCFA肉汤在厌氧条件下,然后被洗通过反复离心法颗粒和re-suspending PBS。营养细胞被杀使用乙醇休克疗法如前所述,文化被连续稀释和镀YCFA琼脂,没有0.1%肠道胆汁盐(牛磺胆酸盐、胆盐和glycocholate)。菌落(c.f.u)被数72 h后的褶皱变化c.f.u.出现在盘子的数量在一个特定的开端的关于c.f.u.出现在盘子的数量没有发芽的计算。检测极限(200 c.f.u.毫升−1)是用于c.f.u.恢复的数量hathewayi梭状芽胞杆菌镀没有发芽的允许叠化计算。实验以确定non-spore-formers开端的是同样的反应,除了营养细胞不是乙醇处理而是连续稀释,直接镀后清洗。
加入代码
主要登记入册
欧洲核苷酸存档
存款数据
组装和注释基因组序列数据存入欧洲核苷酸档案加入号码ERP012217。细菌隔离已经沉积莱布尼兹研究所DSMZ-German Microorganims集合和细胞培养(http://www.dsmz.de),CCUG-Culture收集、瑞典哥德堡大学(http://www.ccug.se),比利时联合主办的集微生物实验室微生物在根特大学(BCCM /液化沼气)(http://bccm.belspo.be/)和日本收集的微生物(JCM;http://jcm.brc.riken.jp/en/)。加入隔离数字中列出补充表1。没有加入任何隔离数字可按照客户要求定制。
引用
秦,j . et al。人类肠道微生物基因目录建立了宏基因组测序。自然464年59 - 65 (2010)
Lozupone, c。,Stombaugh, J. I., Gordon, J. I., Jansson, J. K. & Knight, R. Diversity, stability and resilience of the human gut microbiota.自然489年220 - 230 (2012)
沃克,a·W。,邓肯,s . H。,Louis, P. & Flint, H. J. Phylogeny, culturing, and metagenomics of the human gut microbiota.Microbiol趋势。22267 - 274 (2014)
尼尔森,h . b . et al .识别和组装基因组和遗传因素在复杂的宏基因组样本不使用参考基因组。生物科技自然》。32822 - 828 (2014)
纳尔逊,k . e . et al。人类微生物组参考基因组的一个目录。科学328年994 - 999 (2010)
斯图尔特·e·j·unculturable细菌增长。j . Bacteriol。194年4151 - 4160 (2012)
邓肯,s . H。,,g . L。,Harmsen, H. J., Stewart, C. S. & Flint, H. J. Growth requirements and fermentation products of梭菌属prausnitzii,并提议重新分类Faecalibacterium prausnitzii11月,将军梳子。11月。Int。j .系统。另一个星球。Microbiol。522141 - 2146 (2002)
域名,t . d . et al .抗生素治疗艰难梭状芽胞杆菌载体老鼠触发supershedder状态,spore-mediated传播,并在宿主免疫功能不全的严重疾病。感染。Immun。77年3661 - 3669 (2009)
弗朗西斯,m . B。艾伦,c。,Shrestha, R. & Sorg, J. A. Bile acid recognition by the艰难梭状芽胞杆菌发芽的受体、中海壳牌、建立感染是很重要的。公共科学图书馆Pathog。9e1003356 (2013)
Janoir, c . et al .自适应策略和发病机理艰难梭状芽胞杆菌从在活的有机体内转录组。感染。Immun。81年3757 - 3769 (2013)
Rajilić-Stojanović,m &德·沃斯·w·m·第一1000种培养的人类肠道微生物群。《。牧师。38996 - 1047 (2014)
加尔佩林,m . y . et al .基因组孢子形成的决定因素杆菌和梭状芽胞杆菌:对sporulation-specific基因的最小集合。环绕。Microbiol。142870 - 2890 (2012)
Abecasis, A . b . et al .基因签名和新孢子形成的识别基因。j . Bacteriol。195年2101 - 2115 (2013)
米,c . j . & Beiko r . g . phylogenomic生态专业的看法Lachnospiraceae,一个家庭的消化tract-associated细菌。基因组医学杂志。另一个星球。6703 - 713 (2014)
莱利,t . V。,Brazier, J. S., Hassan, H., Williams, K. & Phillips, K. D. Comparison of alcohol shock enrichment and selective enrichment for the isolation of艰难梭状芽胞杆菌。论文。感染。99年355 - 359 (1987)
Fodor, a . a . et al。“通缉”从人类全基因组测序的微生物类群。《公共科学图书馆•综合》7e41294 (2012)
•德•胡恩m . J。,Eichenberger, P. & Vitkup, D. Hierarchical evolution of the bacterial sporulation network.咕咕叫。医学杂志。20.R735-R745 (2010)
Onyenwoke r U。布里尔,j . A。,Farahi, K. & Wiegel, J. Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch (Firmicutes).拱门。Microbiol。182年182 - 192 (2004)
福斯特,s . c . et al . HPMCD:人类微生物群落的宏基因组数据库的数据集和微生物基因组参考。核酸Res。44D604-D609 (2016)
信仰,j。j。人类肠道微生物群的长期稳定。科学341年1237439 (2013)
城堡,p D。,Iverson, K. D., Petrosino, J. F. & Schloss, S. J. The dynamics of a family’s gut microbiota reveal variations on a theme.微生物组225 (2014)
Paredes-Sabja D。,Shen, A. & Sorg, J. A.艰难梭状芽胞杆菌芽孢生物学:孢子形成、萌发和芽孢结构蛋白质。Microbiol趋势。22406 - 416 (2014)
他,m . et al .疫情罹患卫生保健相关。病的出现和全球传播艰难梭状芽胞杆菌。自然麝猫。45109 - 113 (2013)
古德曼,a . l . et al .广泛个人人类肠道微生物群文化集合的特点和操作在无菌的老鼠。Proc。《科学。美国108年6252 - 6257 (2011)
Lagier j . c . et al .微生物文化组学:人类肠道微生物研究的范式转变。中国。Microbiol。感染。181185 - 1193 (2012)
路德维希·w . et al . ARB:序列数据的软件环境。核酸Res。321363 - 1371 (2004)
城堡,p . d . et al .引入mothur:开源,独立于平台的,支持的软件描述和比较微生物群落。达成。环绕。Microbiol。75年7537 - 7541 (2009)
博萨德,P P。亚伯,S。,Zbinden, R., Böttger, E. C. & Altwegg, M. Ribosomal DNA sequencing for identification of aerobic gram-positive rods in the clinical laboratory (an 18-month evaluation).j .中国。Microbiol。414134 - 4140 (2003)
Clarridge, j . e . III。的影响16 s rRNA的细菌基因序列分析鉴定临床微生物学和传染病。中国。Microbiol。牧师。17840 - 862 (2004)
王,问。,Garrity, G. M., Tiedje, J. M. & Cole, J. R. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy.达成。环绕。Microbiol。73年5261 - 5267 (2007)
Altschul, s F。、《W。,Miller, W., Myers, E. W. & Lipman, D. J. Basic local alignment search tool.j·摩尔,杂志。215年403 - 410 (1990)
价格,m . N。,Dehal, P. S. & Arkin, A. P. FastTree 2—approximately maximum-likelihood trees for large alignments.《公共科学图书馆•综合》5e9490 (2010)
Letunic, i &博克,p .互动生命之树v2:在线注释和显示的系统发育树容易。核酸Res。39W475-W478 (2011)
Kozich, J·J。,Westcott, S. L., Baxter, N. T., Highlander, S. K. & Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform.达成。环绕。Microbiol。79年5112 - 5120 (2013)
木头,d . e . &扎尔茨贝格,s . l .巨妖:超速的宏基因组序列分类使用精确对齐。基因组医学杂志。15R46 (2014)
Langmead, b &扎尔茨贝格,s . l .快gapped-read符合领结2。自然方法9357 - 359 (2012)
域名,t . d . et al .蛋白质组和基因组特征的高度传染性艰难梭状芽胞杆菌630个孢子。j . Bacteriol。191年5377 - 5386 (2009)
博萨德,P P。Zbinden, R。&Altwegg, M.Turicibacter肝病杂志11月,11月,将军sp.小说厌氧的革兰氏阳性细菌。Int。j .系统。另一个星球。Microbiol。521263 - 1266 (2002)
邓肯,s . H。,,g . L。,Barcenilla, A., Stewart, C. S. & Flint, H. J.Roseburia intestinalis小说saccharolytic sp. 11月,butyrate-producing细菌从人类粪便。Int。j .系统。另一个星球。Microbiol。521615 - 1620 (2002)
Iino, T。,Mori, K., Tanaka, K., Suzuki, K. & Harayama, S.Oscillibacter valericigenes11月,11月,将军sp. valerate-producing厌氧细菌隔绝的消化道日本蚬蛤。Int。j .系统。另一个星球。Microbiol。571840 - 1845 (2007)
Paredes-Sabja D。,Setlow, P. & Sarker, M. R. Germination of spores ofBacillales和梭菌属的物种:机制和蛋白质参与。Microbiol趋势。1985 - 94 (2011)
确认
这项工作是支持的威康信托基金会(098051);英国医学研究理事会(PF451 T.D.L.);澳大利亚国家卫生和医学研究委员会(1091097 S.C.F.)和维多利亚政府运营基础设施支持计划(S.C.F.)。我们感谢杜德恒和a·沃克输入。我们也愿意承认资金维尔康姆基金会桑格研究所的技术翻译团队,测序和生物信息学病原体信息学的支持团队。
作者信息
作者和联系
贡献
合,B.O.A. and M.D.S. developed culturing procedures; H.P.B. carried out anaerobic culturing and bacterial isolation; D.G. prepared TEM images; S.C.F., N.K. and H.P.B. performed bioinformatics analyses; H.P.B., S.C.F. and T.D.L. designed the study. H.P.B., S.C.F., B.A.N. and T.D.L. analysed data and wrote the paper.
相应的作者
道德声明
相互竞争的利益
作者提出了专利申请相关工作描述。
扩展数据数据和表
扩展数据图1为培养一个工作流,归档以及肠道微生物群的表征。
一个- - - - - -d原理图的工作流程,包括细菌培养和基因分离和描述细菌物种从人类肠道微生物群。过程包括几个步骤,文化,re-streak、归档和表型。一个、新鲜粪便样品不及时治疗或治疗选择的细菌所需的表型(如孢子形成)。凳子是均质连续稀释然后整除的匀浆YCFA琼脂培养细菌接种。b通过选择单一的殖民地,隔离标识,有纯洁和全长16 s rRNA基因扩增和测序。c,每一个独特的,小说和所需的隔离存档冻结在一个文化集合和一个全基因组序列生成。d宏基因组研究的表型特征和功能验证可以执行在体外和在活的有机体内。
图2扩展数据序列的比较阅读内容的粪便样品和培养样品六个捐助者。
大多数的序列读取从原始捐赠者粪便样本(n= 6)出现在文化既是原始读取样品(93%平均共享整个6捐助者)之后新创组装(72%平均共享整个6捐助者)。
图3扩展数据存档通过厌氧培养细菌的多样性和新颖性。
一个,b21的代表性物种25最丰富的细菌属(一个)和23 24最丰富的物种(b)被孤立和存档(丰度是由宏基因组测序和基于平均相对丰度六个捐助者(n= 6))。这代表了96%的平均相对丰度在属级和90%的平均相对丰度在物种水平六个捐助者。一个红点一个表明从每个属的物种数量存档。Lachnospiraceae incertae基准,不保密的Lachnospiraceae,梭状芽胞杆菌第四,梭状芽胞杆菌习不是严格的属和代表目前非保密的物种。Odoribacter splanchnicus在b是唯一一种没有存档。c肠道菌群成员,卑微的代表也讲究的。至少一个从显示每个属的代表性物种是有文化的。值和范围提出了上面的排名与分类单元的平均价值。d在这项研究中,细菌培养的数量。从每个类别至少40%是未知的。
扩展数据图4种系发生肠道孢子形成的细菌。
全长16 s rRNA基因发展史说明ethanol-resistant细菌的分类关系内的硬壁菌门菌培养捐赠者粪便样本。分支的颜色表示不同的家庭。阴影文字表示物种培养从ethanol-treated粪便样品和无遮蔽的文字表示物种培养从non-ethanol-treated粪便样本。百分数表示最近的身份特征的物种。透射电子显微图(显微镜)的孢子超微结构的系统多样化的选择培养细菌在图片和箭头所示包括候选人小说家庭有86%身份的16 s rRNA基因序列thermocellum梭状芽胞杆菌。典型的孢子结构定义和说明在相同的图像。显微镜是根据箱子旁边的物种名称排序。规模酒吧每个图像的底部所示。梭状芽孢杆菌包含上下文。细菌显示ethanol-resistant表型代表物种之前列为non-spore-formers (Turicibacter肝病杂志38小说和密切相关的候选物种),物种non-spore-formers密切相关(Roseburia intestinalis39和Oscillibacter valericigenes40小说和密切相关的候选物种)或物种形成孢子的嫌疑,据我们所知,从来没有证明这样做直到现在(真细菌eligens,真细菌rectale,Coprococcus来41和相关候选人小说物种)。
扩展数据图5中的孢子形成人类肠道微生物基因组签名。
识别孢子形成细菌物种的基因签名包含孢子形成,germination-associated基因和基因不是先前与孢子形成有关。外圆上孢子形成的特征基因,基因与特定的孢子形成周期或无特征基因在矩形内。梭状芽孢杆菌菌株630个基因的名字在可能的情况下使用,否则轨迹标记标识符显示。枯草芽孢杆菌基因名字当没有被使用梭状芽孢杆菌同系物是可用的。签名是富含sporulation-associated基因从孢子形成和萌发阶段电流-电压周期(重要问< 3.0×10−37,确切概率法)。与监管相关的基因存在至少有10个基因编码的监管或dna结合角色(问< 1.4×10−5,确切概率法)。与孢子形成有关的基因不是以前也存在这些假定的角色作为热休克,膜相关蛋白和DNA-polymerase-associated蛋白质。
图6扩展数据验证和孢子形成的表征签名。
一个,签名准确区分孢子形成和non-spore-forming细菌培养从这项研究和在不同环境(spore-formersn= 57,non-spore-formersn= 50,乙醇处理后培养n= 69,没有乙醇处理后培养n= 149)。指补充表1签名测试分数的细菌。意味着南达科他州±。b,功能类分配给签名揭示了一个广泛的功能和孢子形成过程,regulation-associated基因控制。
扩展数据图7比non-spore-forming细菌孢子形成的细菌更有弹性的消毒剂等环境压力。
纯细菌培养都沉浸在乙醇4 h在洗前和接种到YCFA增长中牛磺胆酸盐钠作为开端的。只有孢子形成的细菌活了下来。分类姓氏在括号中所示。虚线表示的文化检测极限c.f.u. 50毫升−1。均值±南达科他州。n= 3生物复制每一物种测试。
扩展数据图8 non-spore-forming细菌增长反应肠道发芽的。
c.f.u.出现在盘子的数量在一个特定的开端的表示为一个褶皱变化对c.f.u.出现在盘子的数量没有发芽的。没有事先进行乙醇休克疗法。的褶皱变化(虚线)表明一个开端的数量没有影响c.f.u.从细菌中恢复过来。没有基于一个未配对的统计上的显著差异t以及每个发芽的条件对没有发芽的条件。意味着和范围,n= 3生物物种的复制。
扩展数据图9的验证的比例估计spore-formers肠道菌群。
全长16 s rRNA基因扩增子序列被用来确定细菌的分类比例从六个捐赠者粪便样本。从每个捐赠者孢子形成细菌培养和分类分类被分配的主要文本中描述。属(圆)和家庭(广场)分类排名被指定为上下极限计算的比例spore-formers分类级别。具体属和家庭如果他们包含一个物种,包括培养后乙醇休克疗法。意味着南达科他州±。
补充信息
补充表
这个文件包含补充表1。(XLSX 129 kb)
权利和权限
这项工作是基于知识共享署名4.0国际4.0 (CC)许可证。本文中的图片或其他第三方材料都包含在本文的创作共用许可证,除非另有说明在信贷额度;如果材料不包括在Creative Commons许可下,用户需要获得许可执照持有人繁殖材料。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
布朗,H。,Forster, S., Anonye, B.et al。培养的unculturable人类微生物群揭示小说类群和广泛的孢子形成。自然533年,543 - 546 (2016)。https://doi.org/10.1038/nature17645
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature17645
本文引用的
比较微生物签名之间的配对粪便和直肠活检样本使用下一代测序和文化组学健康志愿者
微生物组(2022)
口腔生态环境修改硬奶酪:从pH值到微生物:基于16 s rRNA metabarcoding前瞻性队列研究的方法
转化医学杂志》(2022)
Sediment-associated微生物群落分析:通过连续膜过滤样品预处理16 s rRNA扩增子测序
BMC微生物学(2022)
在继承共生微生物的微生物的影响
BMC生态学与进化(2022)
Species-targeted排序和培养肠道微生物的共生的细菌在厌氧条件下使用流式细胞仪
微生物组(2022)