文摘
当前sequencing-based粪便微生物群的分析量化微生物类群和代谢途径的分数样本序列库生成的每个分析1,2。虽然这些相对的方法允许检测抵抗微生物变异,他们的能力是有限的,揭示了微生物群与宿主相互作用的健康3,4。相对的比较分析微生物数据不能提供信息的程度或方向性类群丰度变化或代谢潜力5。如果微生物样本之间的负载变化显著,相对分析将会阻碍试图链接微生物特性定量数据等生理参数或代谢物浓度5,6。近年来,相对方法忽略的可能性改变整体丰富微生物群本身可能是疾病相关生态系统的一个关键标识符配置7。使真正的host-microbiota交互的特性,微生物研究必须为计数交换比率4,8,9。我们建立一个工作流的粪便定量微生物分析材料,通过并行化微生物细胞的扩增子序列和流仪枚举。我们观察到十倍的差异健康个体的微生物负载和将这种变化与菌群分化却能合成更多形成联系起来。我们展示了微生物丰度支撑微生物群变异个体间和共变主机表型。定量分析绕过组合性影响肠道微生物群的交互网络的重建和显示分类之间的权衡拟杆菌和普氏菌是相对的人工制品微生物分析。最后,我们确定微生物负载作为一个观察到的微生物群的关键驱动因素改变的克罗恩病的患者10,这与low-cell-count有关拟杆菌菌群(定义通过却能合成更多形成相对分析)11,12。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
运输设备网络分析:增值贡献
杂志的经济结构开放获取2022年12月06
一种改进的工作流程准确使用宏基因组和健壮的医疗环境监测
微生物组开放获取2022年12月02
长期生活历史预测当前肠道微生物在以人群为基础的队列研究
自然老化开放获取2022年10月14日
访问选项
订阅自然+
得到直接在线访问自然和其他55个自然杂志
29.99美元
每月
订阅杂志
得到完整的期刊访问1年
199.00美元
只有3.90美元的问题
所有价格是净价格。
增值税将被添加后在付款。
税计算期间将完成付款。
买条
时间有限或全文访问ReadCube。
32.00美元
所有价格是净价格。
加入代码
引用
Zhernakova, a . et al。基于宏基因组分析显示标记为肠道微生物组成和多样性。科学352年565 - 569 (2016)
Falony, g . et al .肠道微生物群体分析变异。科学352年560 - 564 (2016)
Valles-Colomer, m . et al . Meta-omics在炎症性肠病的研究:应用程序,挑战,和指导方针。j·克罗恩氏结肠炎10735 - 746 (2016)
Satinsky, b . M。,Gifford, S. M., Crump, B. C. & Moran, M. A. Use of internal standards for quantitative metatranscriptome and metagenome analysis.Enzymol方法。531年237 - 250 (2013)
莫顿,j . t . et al .平衡树揭示微生物生态位分化。mSystems2e00162-16 (2017)
Gloor, g B。吴,j . R。,P一个wlowsky-Glahn, V. & Egozcue, J. J. It’s all relative: analyzing microbiome data as compositions.安。论文。26322 - 329 (2016)
Harmsen h·j·M。Pouwels, s D。凡克,。,Bos, N. A. & Dijkstra, G. Crohn’s disease patients have more IgG-binding fecal bacteria than controls.中国。疫苗Immunol。19515 - 521 (2012)
道具、r . et al .微生物丰度分类单元的绝对量化。ISME J。11584 - 587 (2017)
Stammler, f . et al .调整微生物资料供激增细菌微生物负载的差异。微生物组428 (2016)
落羽杉,j . et al .原发性硬化性胆管炎的特点是肠道失调独立于炎症性肠病。肠道65年1681 - 1689 (2016)
福尔摩斯,我。,Harris, K. & Quince, C. Dirichlet multinomial mixtures: generative models for microbial metagenomics.《公共科学图书馆•综合》7e30126 (2012)
Arumugam, m . et al .人类肠道微生物菌群。自然473年174 - 180 (2011)
刘易斯,美国j . &希顿k . w .粪便形成规模作为一个有用的指南肠转运时间。Scand。j .杂志。32920 - 924 (1997)
Vandeputte D。Falony, G。D 'hoe, K。,Vieira-Silva, S. & Raes, J. Water activity does not shape the microbiota in the human colon.肠道66年1865 - 1866 (2017)
Vandeputte, d . et al .粪便一致性与肠道微生物群的丰富性和组成密切相关,菌群和细菌的增长速度。肠道65年57 - 62 (2016)
Tigchelaar e . f . et al .肠道微生物群组成与粪便一致性。肠道65年540 - 542 (2016)
Fuller, b . j .冷冻保护剂:必要的防冻液保护生命在冷冻状态。低温。25375 - 388 (2004)
Hugon, p . et al .分子研究忽视显然革兰氏阴性人群在人类肠道微生物群。j .中国。Microbiol。513286 - 3293 (2013)
本·阿莫,k . et al .可行的遗传多样性,受伤,死亡粪便细菌评估fluorescence-activated细胞分类和16 s rRNA基因分析。达成。环绕。Microbiol。71年4679 - 4689 (2005)
Roager, h . m . et al .结肠转运时间与细菌新陈代谢和肠道粘膜营业额。Microbiol Nat。116093 (2016)
McMurdie, p . j . &福尔摩斯,不浪费,要不为什么纯化微生物数据是不可接受的。公共科学图书馆第一版。医学杂志。10e1003531 (2014)
古德里奇,j . k . et al .进行微生物研究。细胞158年250 - 262 (2014)
Magurran a E。测量生物多样性(布莱克威尔,2004)
浮士德,k & Raes j .微生物的相互作用:从网络模型。启Microbiol Nat。10538 - 550 (2012)
Lozupone, c。,Stombaugh, J. I., Gordon, J. I., Jansson, J. K. & Knight, R. Diversity, stability and resilience of the human gut microbiota.自然489年220 - 230 (2012)
帕斯卡,诉et al。克罗恩病的微生物签名。肠道66年813 - 822 (2017)
Kozich, J·J。,Westcott, S. L., Baxter, N. T., Highlander, S. K. & Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform.达成。环绕。Microbiol。79年5112 - 5120 (2013)
普雷斯特,我大肠。哈姆,F。,Kötzsch, S., van Loosdrecht, M. C. M. & Vrouwenvelder, J. S. Monitoring microbiological changes in drinking water systems using a fast and reproducible flow cytometric method.水Res。477131 - 7142 (2013)
拉姆齐,c . a . et al .病原体和宿主反应的识别标记与牙周疾病相关。j . Periodontol。80年436 - 446 (2009)
铁托,r . y . et al。Dialister作为微生物spondyloarthritis疾病活动的标志。关节炎Rheumatol。69年114 - 121 (2017)
Girardot C。,Scholtalbers, J., Sauer, S., Su, S. Y. & Furlong, E. E. Je, a versatile suite to handle multiplexed NGS libraries with unique molecular identifiers.BMC生物信息学17419 (2016)
Magocˇt &扎尔茨贝格,s . l .闪光:快速调整长度短读提高基因组装配。生物信息学272957 - 2963 (2011)
埃德加,r . C。,Haas, B. J., Clemente, J. C., Quince, C. & Knight, R. UCHIME improves sensitivity and speed of chimera detection.生物信息学272194 - 2200 (2011)
王,问。,Garrity, G. M., Tiedje, J. M. & Cole, J. R. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy.达成。环绕。Microbiol。73年5261 - 5267 (2007)
斯托达德,美国F。史密斯,b . J。嗯,R。,Roller, B. R. K. & Schmidt, T. M. rrnDB: improved tools for interpreting rRNA gene abundance in bacteria and archaea and a new foundation for future development.核酸Res。43D593-D598 (2015)
McMurdie, p . j . &福尔摩斯s phyloseq: R包可再生的交互式分析微生物人口普查数据和图形。《公共科学图书馆•综合》8e61217 (2013)
Oksanen, j .等人素食:社区生态包。R包版本2.2-1https://CRAN.R-project.org/package=vegan(2015)
眉目传情,d·h·FSA:渔业股票分析。R包版本0.8.13https://cran.r-project.org/package=FSA(2017)
Hothorn, T。,Hornik, K., van de Wiel, M. A. & Zeileis, A. A Lego system for conditional inference.点。统计。60257 - 263 (2006)
摩根,m . DirichletMultinomial: Dirichlet-multinomial混合微生物组数据的机器学习模型。R包版本1.18.0https://cran.r-project.org/package=dirmult(2017)
Wolak m .刑事法庭:促进组内相关系数的估计。R包版本tripwirehttps://cran.r-project.org/package=ICC(2016)
Delignette-Muller, m . l . & Dutang fitdistrplus: R包拟合分布。Softw j . Stat。64年猴(2015)
业务Benjamini y & y控制错误发现率:一个实用和强大的多个测试方法。j . r . Stat。Soc。B57289 - 300 (1995)
Vieira-Silva, s . et al . Species-function塑造人类肠道微生物的生态属性的关系。Microbiol Nat。116088 (2016)
史密斯,c . j . &奥斯本,a . m .优点和局限性定量PCR (Q-PCR)的方法在微生物生态学。《。生态。67年6 20 (2009)
哈姆,f . et al . Flow-cytometric总细菌细胞计数作为一个描述性的微生物参数对饮用水处理工艺。水Res。42269 - 277 (2008)
Habtewold, T。,Duchateau, L. & Christophides, G. K. Flow cytometry analysis of the microbiota associated with the midguts of vector mosquitoes.Parasit。向量9167 (2016)
确认
我们感谢所有参与者,f·吉拉尔多为使样本收集PXL特,l . Rymenans和c . Verspecht粪便DNA提取和图书馆准备,促进水分含量决定韦贝克k, p . Goncalves咨询关于模拟微生物数据为基准QMP和RMP方法。这项研究的主要资金来自一个KU鲁汶克雷亚格兰特。的官员支持科技创新的机构(IWT)。G.K.,K.D., M.V.-C., S.V.-S., and J.W. are funded by the Research Foundation Flanders (FWO-Vlaanderen). This work is further supported through funding by VIB, the Rega Institute for Medical Research, KU Leuven, FP7 METACARDIS (HEALTH-F4-2012-305312), and H2020 SYSCID (grant agreement 733100).
作者信息
作者和联系
贡献
这项研究是由G.F.实验设计的官员,S.V.,G.F.,和J.R. Sampling of cohorts was set up and carried out by D.V., G.K., K.D., S.V.-S., M.V.-C., J.S., J.W., R.Y.T., L.D.C., and G.F. Optimization of sequencing protocols was performed by R.Y.T.; data pre-processing by D.V., M.V.-C., J.S., J.W., and Y.D.; flow cytometry analyses by G.K. and K.D.; statistical analyses by D.V., G.K., K.D., S.V.-S., M.V.-C., J.S., J.W., and G.F.; network analyses by S.V.-S.; and simulation experiments by D.V. and S.V.-S. G.F. developed the QMP protocol. S.V.-S., G.F., and J.R. drafted the manuscript. All authors revised the article and approved the final version for publication.
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
额外的信息
审核人信息自然由于w . m . de Vos,另一个匿名的评论家(s)为他们的贡献的同行评审工作。
出版商的注意:施普林格自然保持中立在发表关于司法主权地图和所属机构。
扩展数据数据和表
图1扩展数据量化的微生物负载冷冻粪便样本。
一个,微生物细胞计数在新鲜和冷冻粪便整除强烈相互关联(研究对象、n= 39;皮尔森的r= 0.91,两面P= 4.9×10−16)。数据点代表平均值。误差线代表重复的金丝(新鲜),一式三份(冻)细胞计数。b,比较流仪评估微生物负载和细菌丰度估计的基础上qPCR(研究对象、n= 40)。数据点代表平均值。误差线代表一式三份的美国价值观(qPCR和细胞计数)。虽然比较细胞和分子枚举方法不推荐18(皮尔森,测量相关r=−0.53,两面P= 4.7×10−4)。在一个复杂的生态系统,列举细菌qPCR将引入偏差的基础上,通过提取、纯化、扩增的DNA, 16 s rRNA基因拷贝数变异,和社区复制率(如图所示,菌群之间的不同44),等等。此外,qPCR据报道只有足够灵敏检测两个基因浓度或微生物负载的变化45。流式细胞术不太具体,结果可能会影响聚合物的形成8。对于QMP分析,我们选择了流式细胞术方法由于其技术坦率(有限数量的技术操作;看到方法),再现性46,47,和吞吐量。
扩展数据图2个体内部和inter-individual人类粪便微生物细胞计数的变化。
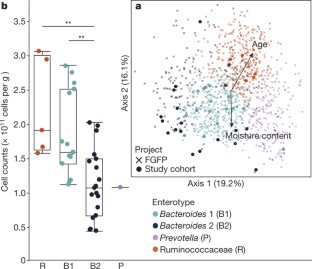
20个健康个体采样每日在一周(每天最多一个样本;纵向队列)。健康个体细胞计数明显高于克罗恩病的患者。病人组疾病群体,n= 29 (CD;紫色)与整体日常样品,n= 132(印第安纳州;灰色)或个人的平均细胞计数(市的av;橙色)。双面Wilcoxon rank-sum测试,* * *P< 0.001。箱线图的主体代表第一和第三个四分位数的分布和中间线。四分位数的胡须扩展到最后1.5×四分位范围内数据点,与异常值。
扩展数据图3粪便微生物负载与样品水分含量和种类丰富。
一个、微生物细胞数和水分含量在纵向和负相关验证组,虽然不是在这项队列研究(研究对象、n= 37,枪兵的ρ=−0.12,两面罗斯福= 0.56;纵向队列,n= 132,枪兵的ρ=−0.52,两面罗斯福= 1.6×10−9;验证组,n= 54,枪兵的ρ=−0.56,两面罗斯福= 9.1×10−5)。b、微生物细胞数和观察丰富相关温和(研究对象、n= 40;斯皮尔曼的ρ= 0.36,两面P= 2.3×10−2)。
扩展数据图4粪便菌群的微生物负载不同。
微生物负载之间的差异的四个菌群疾病组(n= 95)。箱线图表示微生物负载(每克粪便细胞)分布在四个菌群。箱线图的主体代表第一和第三个四分位数的分布和中间线。四分位数的胡须扩展到最后1.5×四分位范围内数据点,与异常值。双面Dunn的调整测试,* *P< 0.01,* * *P< 0.001。
扩展数据图5微生物负载不与测序深度有关。
测序深度没有反映出的总微生物负载(枪兵的一个示例ρ= 0.17,两面P= 0.28)。排名是根据样品减少细胞计数。
扩展数据图6插图RMP和QMP方法之间的差异。
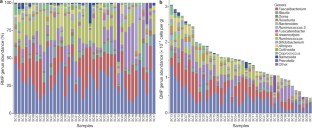
两个样品,每个片段包含4个属,分析(数据说明)。属大量分布在A和B是明显不同的样本,样本的微生物负荷B A负载样本内的两倍多属“紫色”16 s rRNA基因的两个副本。(1)制备DNA提取和图书馆。无论是RMP还是QMP纠正偏差引入的DNA提取、引物特异性PCR扩增或其他库制备步骤。结果测序深度是独立的微生物负载。(2)通过纯化偶数个读取每个样本,RMP假定类似属丰度分布样本A和B:示例B因此测序比样品更密集。结果概要文件因此差反映了属分布在原始样本。鉴于16 s rRNA基因的多个副本,“紫色”的相对丰度是高估了。(3)的第一步QMP 16 s rRNA拷贝数变异的纠正。在结果copy-corrected概要(CCP),每个读符合一个细菌进行测序。(4)中国共产党读总(R)除以微生物负载(X)为每个样本采样深度估计。 For sample A and B, sampling depth is [R]一个除以[X]一个和[R]B除以[X]B,分别。采样深度B是两个的最低(3.33%);同一水平的样本是稀薄的。这意味着“橙色”不再是发现。“橙色”也同样丰富A和B,它包含在示例RMP可以被认为是一个人工制品的强度不均匀采样。由此产生的稀薄属丰度与样品微生物负载和比例可以外推到生成QMPs表示为每克细胞的数量。
扩展数据图7 RMP和QMP丰度分布拟杆菌和普氏菌在健康对照组。
样品(n= 66)根据降低排名拟杆菌丰富RMP和QMP面板(堆叠酒吧)。之间的权衡拟杆菌和普氏菌(RMP);斯皮尔曼的ρ10 =−0.59,两面罗斯福= 2−4修正后)不再是重要的微生物负载(QMP;斯皮尔曼的ρ=−0.33,两面罗斯福= 1)。
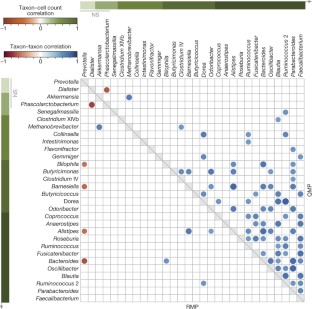
扩展数据图8错误发现率和灵敏度在网络重建基于QMP和RMP模拟数据。
QMP导致增加灵敏度和减少罗斯福相比RMP(两面t以及,P< 10−15)。为两个、四个,8倍最大不同微生物负载,QMP罗斯福是11%,15%,和22%低,分别比RMP;和QMP真阳性发现率(灵敏度)增长了10%,12%,15%。数据点描述重复(n= 50)。箱线图的主体代表第一和第三个四分位数的分布和中间线。四分位数的胡须扩展到最后模拟数据点在1.5×四分位范围。
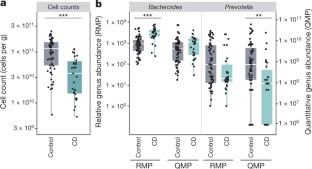
扩展数据图9 RMP低估了减少微生物丰富与克罗恩病相关。
观察到的丰富健康对照组(n= 66)和克罗恩病的患者(n= 29)。丰富的减少与克罗恩病相关QMP更加明显。箱线图的主体代表第一和第三个四分位数的分布和中间线。四分位数的胡须扩展到最后数据点在1.5×四分位范围。双面Wilcoxon测试,* * *P< 0.001,* *P< 0.01。
图10扩展数据流式细胞术控制策略。
一个固定的闸门和染色方法应用28。空白和样品的解决方案都沾SYBR绿色的我。一个,FL1-A / FL3-A收购的一个空白样品生理溶液0.85% (w / v)门边界表示。2000年一个阈值应用于FL1通道。b,进行二次浇注FSC-A / SSC-A通道进一步区分碎片或背景和微生物活动。c,dFL1-A / FL3-A计数采集的粪便样本(c)与二次浇注FSC-A / SSC-A通道在空白的基础上分析(d)。总数量被定义为事件在FL1-A注册/ FL3-A控制区域,不包括碎片或背景事件观察FSC-A / SSC-A R1门。每分钟流量被设定为14μl和收购率不超过每秒10000事件。每个面板反映事件注册在30年代收购。
补充信息
补充表
这个文件包含补充表1 - 11。(XLSX 90 kb)
权利和权限
关于这篇文章
引用这篇文章
Vandeputte D。Kathagen, G。D 'hoe, K。et al。定量微生物分析链接肠道微生物负载社会变化。自然551年,507 - 511 (2017)。https://doi.org/10.1038/nature24460
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature24460
本文引用的
低温贮藏介质条件对经济增长的影响和隔离肠道厌氧菌从人类粪便样本
微生物组(2022)
一种改进的工作流程准确使用宏基因组和健壮的医疗环境监测
微生物组(2022)
定量细菌共生体的微生物种群的研究显示地理差异Ixodes蓖麻
微生物组(2022)
运输设备网络分析:增值贡献
杂志的经济结构(2022)
Species-targeted排序和培养肠道微生物的共生的细菌在厌氧条件下使用流式细胞仪
微生物组(2022)
