文摘
技术变化在宏基因组分析必须最小化自信地对人类健康评估微生物群的贡献。我们测试了21个代表DNA提取协议相同的粪便样本和量化的差异观察微生物群落组成。我们比较差异由于图书馆准备和样本存储,我们与观察到的生物变异在同一标本或一个人。我们发现DNA提取的最大影响宏基因组分析的结果。对DNA提取协议,我们认为产生的DNA数量和质量,我们确定偏差估计的社会多样性和革兰氏阳性和革兰氏阴性细菌之间的比率。我们建议一个标准化的人类粪便样本的DNA提取方法,建立了在实验室可转让性和进一步的基准测试使用模拟群落的组成。它的采用将提高人类肠道微生物研究的可比性,促进荟萃分析。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
肠道微生物群在多发性硬化随疾病活动
基因组医学开放获取2023年1月05
肠道微生物群在克罗恩病的早期阶段独特的特点
肠道病原体开放获取2022年12月14日
一种改进的工作流程准确使用宏基因组和健壮的医疗环境监测
微生物组开放获取2022年12月02
访问选项
订阅自然+
得到直接在线访问自然和其他55个自然杂志
29.99美元
每月
订阅杂志
得到完整的期刊访问1年
99.00美元
只有8.25美元的问题
所有价格是净价格。
增值税将被添加后在付款。
税计算期间将完成付款。
买条
时间有限或全文访问ReadCube。
32.00美元
所有价格是净价格。

加入代码
引用
迈耶,f . et al .宏基因组拉斯特服务器——公共资源的自动系统发育和功能分析基因组。BMC生物信息学9386 (2008)。
拉森:et al .肠道微生物群在成人2型糖尿病患者与非糖尿病成年人。《公共科学图书馆•综合》5e9085 (2010)。
秦,j . et al . metagenome-wide协会研究肠道微生物群的2型糖尿病。自然490年,则高达55 - (2012)。
Forslund, k . et al .理清和二甲双胍治疗2型糖尿病的签名在人类肠道微生物群。自然528年,262 - 266 (2015)。
Manichanh, et al。减少粪便微生物群的多样性在克罗恩病了一个宏基因组的方法。肠道55,205 - 211 (2006)。
卡罗尔,贝聿铭等。分子分析鲁米那,mucosal-associated肠道微生物群的腹泻型肠易激综合症。点。j .杂志。Gastrointest。肝脏杂志。301年G799-G807 (2011)。
西,g . et al .潜在的粪便微生物群结直肠癌的早期检测。摩尔。系统。医学杂志。10766 (2014)。
Dethlefsen, L。,McFall-Ngai, M. & Relman, D.A. An ecological and evolutionary perspective on human-microbe mutualism and disease.自然449年,811 - 818 (2007)。
Dominguez-Bello M.G. et al .交付模式形状的收购和结构跨多个新生儿身体栖息地最初的微生物群。Proc。国家的。学会科学。美国107年,11971 - 11975 (2010)。
Yatsunenko, t . et al .人类肠道微生物组在年龄和地理。自然486年,222 - 227 (2012)。
勒夏特列原理,大肠et al。丰富的人类肠道微生物与代谢标记。自然500年,541 - 546 (2013)。
Wesolowska-Andersen, a . et al。从粪便细菌的DNA提取方法的选择影响群落结构评估的宏基因组分析。微生物组2,19 (2014)。
McOrist, A.L.,Jackson, M. & Bird, A.R. A comparison of five methods for extraction of bacterial DNA from human faecal samples.j . Microbiol。方法50,131 - 139 (2002)。
史密斯,B。李,N。,Andersen, A.S., Slotved, H.C. & Krogfelt, K.A. Optimising bacterial DNA extraction from faecal samples: comparison of three methods.Microbiol开放。J。5,14 - 17 (2011)。
Maukonen, J。,Simões, C. & Saarela, M. The currently used commercial DNA-extraction methods give different results of clostridial and actinobacterial populations derived from human fecal samples.《。生态。79年,697 - 708 (2012)。
肯尼迪,附加的影响等。不同DNA提取试剂盒和实验室的评估人类肠道微生物群组成16 s rRNA基因测序。《公共科学图书馆•综合》9e88982 (2014)。
Salonen, a . et al .粪便DNA提取方法的比较分析与系统发育微阵列:有效恢复使用机械细胞裂解细菌和古细菌的DNA。j . Microbiol。方法81年,127 - 134 (2010)。
Ariefdjohan,分子量,Savaiano, D.A. & Nakatsu, C.H. Comparison of DNA extraction kits for PCR-DGGE analysis of human intestinal microbial communities from fecal specimens.减轻。J。9,23 (2010)。
Sunagawa, s . et al .宏基因组物种分析使用通用系统发育标记基因。Nat方法。10,1196 - 1199 (2013)。
Manichanh C。Borruel, N。,Casellas, F. & Guarner, F. The gut microbiota in IBD.Nat,启杂志。乙醇。9,599 - 608 (2012)。
Lozupone, c.a et al。人类微生物群的荟萃分析的研究。基因组Res。23,1704 - 1714 (2013)。
Raes, j . &博克,p .分子生物学生态系统:对社区功能的理解。启Microbiol Nat。6,693 - 699 (2008)。
沃伊特A.Y. et al。时间和人类肠道基因组的技术变化。基因组医学杂志。1673 (2015)。
Franzosa,电子等。涉及的metatranscriptome和metagenome人类肠道。Proc。国家的。学会科学。美国111年E2329-E2338 (2014)。
歌,中华民国et al .保存方法不同粪便微生物稳定性,影响是否适合现场研究。mSystemshttps://dx.doi.org/10.1128/mSystems.00021-16(2016)。
Gohl D.M. et al。扩增子标记基因的系统改进的方法增加了微生物研究的准确性。生物科技Nat。》。34,942 - 949 (2016)。
Claassen、s等。比较效率的五个不同的商业DNA提取试剂盒提取粪便的DNA样本。j . Microbiol。方法94年,103 - 110 (2013)。
元,年代。,Cohen, D.B., Ravel, J., Abdo, Z. & Forney, L.J. Evaluation of methods for the extraction and purification of DNA from the human microbiome.《公共科学图书馆•综合》7e33865 (2012)。
Kultima jr et al . MOCAT:宏基因组大会和基因预测工具包。《公共科学图书馆•综合》7e47656 (2012)。
秦,j . et al。人类肠道微生物基因目录建立了宏基因组测序。自然464年59 - 65 (2010)。
Huttenhower, c . et al .人类微生物组项目财团。结构、功能和人类健康的微生物多样性。自然486年,207 - 214 (2012)。
Franzosa,电子等。确定个人使用的微生物宏基因组编码。Proc。国家的。学会科学。美国112年E2930-E2938 (2015)。
鲍威尔,美国等人蛋酒v3.0:同源组覆盖1133生物在41个不同分类范围。核酸Res。40D284-D289 (2012)。
Lozupone, c.a,Stombaugh, J.I., Gordon, J.I., Jansson, J.K. & Knight, R. Diversity, stability and resilience of the human gut microbiota.自然489年,220 - 230 (2012)。
圣地亚哥,a . et al .处理粪便样本:在微生物群落分析标准的一步。BMC Microbiol。14112 (2014)。
InhibitEx平板电脑——试剂盒的网上书店。可以在:https://www.qiagen.com/fr/shop/lab-basics/buffers-and-reagents/inhibitex-tablets/。
亨德森,g . et al。DNA提取方法和抽样技术的效果明显的奶牛和绵羊瘤胃微生物群落结构。《公共科学图书馆•综合》8e74787 (2013)。
琼斯,M.B. et al。库制备方法可以影响人类微生物组研究的基因组和功能预测。Proc。国家的。学会科学。美国112年,14024 - 14029 (2015)。
索尔特,中华民国et al .试剂和实验室污染会严重影响基于微生物分析。BMC医学杂志。1287 (2014)。
确认
我们感谢美国Burz和k . Weizer编辑和网络声明安抚。我们感谢d Ordonez和N.P.加洛佩兹建议在流式细胞仪上,流式细胞仪提供的核心设施,EMBL的。这项研究是由欧盟第七框架计划通过国际人类微生物组标准(健康- f4 - 2010 - 261376)资助。我们也收到了来自苏格兰政府支持农村和环境科学以及EMBL和分析服务。
作者信息
作者和联系
贡献
P.I.C.,S.S. and G.Z. analyzed data and drafted and finalized the manuscript. E.P. and A.A. analyzed data, sequenced samples and wrote the manuscript. F.L., J.R.K., M.R.H., L.P.C. and E.A.-V. analyzed data and wrote the manuscript. M.T., M. Driessen, R.H., F.-E.J. and K.R.P. created and quantified the mock community. M.B., J.R.M.B., L.B., T.C., S.C.-P., M. Derrien, A.D., M. Daigneault, R.A.L., W.M.d.V., B.B.F., H.J.F., F.G., M.H., H.H., J.v.H.V., J.J., I.K., P.L., E.L.C., V.M., C. Manichanh, J.C.M., C. Mery, H.M., C.O., P.W.O., J.P., S.P., N.P., M.P., A.S., D.S., K.P.S., B.S., K.S., P.V., J.V., L.Z. and E.G.Z. extracted samples and wrote the manuscript. S.D.E., J.D. and P.B. designed the study and wrote the manuscript.
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
综合补充信息
补充图1 Inter-individual距离依赖研究。
类似于图3,我们显示不同参数的估计效果大小的上下文中inter-individual距离评估在不同的研究使用。很明显,虽然不大,但在中等距离是有明显区别的研究,与高分子聚合物样品出现MetaHIT更同质的。
补充图2在两个样品提取的偏见。
提取偏差在两个样品是一致的,独立的距离测量使用。(一个)显示PCoA投影的物种丰度对于每一个样本,独立,使用枪兵排名相关性以及欧氏距离。大部分的变异是被前两个主坐标和提取方法的聚类很容易观察到的。(b)显示了一个PCoA投影功能的距离,斯皮尔曼排名和欧几里得。
补充图3革兰氏阳性细菌的溶菌作用积极与香农多样性。
革兰氏阳性细菌的复苏与整体香农多样性。只考虑前20名最丰富的物种在每个样本中,比率计算之间都是革兰氏阳性和革兰氏阴性细菌和革兰氏阴性细菌的革兰氏阴性。前面板显示这些比率与香农多样性指数的相关性,而较低的面板是这种相关性在最丰富的革兰氏阳性和革兰氏阴性细菌,是常见的两种样品A和B,表明积极的关系强劲复苏的革兰氏阳性细菌和观察香农多样性。
补充图4香农多样性的样本组成。
观察香农多样性一直受到提取方法,如两个样本。此外,有一个相当大的多样性两个样品之间的差异,提取不覆盖的偏见。
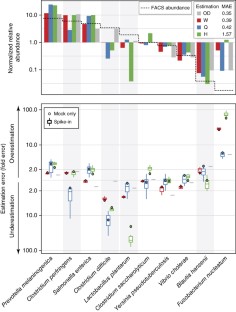
补充图5提取偏差表现最好的协议被认为是在第二阶段。
提取的变化是相同的在第二阶段复制的阶段我(酒吧1和2,分别)。此外,已合并成三个协议协议Q为第二阶段,即6、9和15产生类似的结果,目前提取偏差低于生物复制变异。树二期协议(H, W和Q),当应用在不同的实验室,没有先前的经验所使用的特定协议,产生类似的丰度估计,错误水平以下的生物变异在一个标本。
权利和权限
关于这篇文章
引用这篇文章
Costea, P。,Zeller, G., Sunagawa, S.et al。对人类粪便样本处理宏基因组研究标准。生物科技Nat》35,1069 - 1076 (2017)。https://doi.org/10.1038/nbt.3960
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nbt.3960
本文引用的
肠道微生物群在多发性硬化随疾病活动
基因组医学(2023)
肠道微生物的影响消化系统自身免疫性疾病
自然评论免疫学(2023)
一种改进的工作流程准确使用宏基因组和健壮的医疗环境监测
微生物组(2022)
开发完整的细胞微生物标准字段
微生物组(2022)
Ecosystem-specific微生物群和微生物数据库大数据的时代
环境微生物(2022)
