文摘
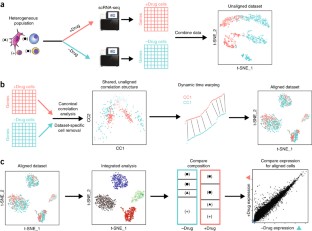
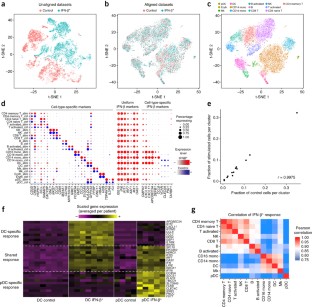
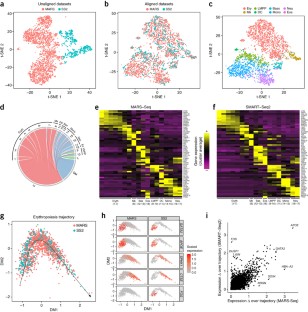
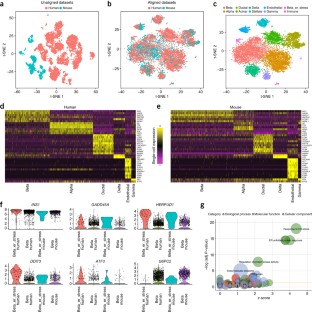
计算单细胞RNA-seq (scRNA-seq)方法已经成功地应用于实验代表一个单一的条件,技术,或物种的发现和定义细胞表型。然而,确定亚种群的细胞,存在多个数据集仍然是具有挑战性的。在这里,我们介绍一种分析战略整合scRNA-seq数据集基于共同来源的变异,使识别共同的人口数据集和下游比较分析。我们应用这种方法,实现在我们的R工具箱修(http://satijalab.org/seurat/),使scRNA-seq外周血单核细胞的数据集在休息和刺激条件下,使用两种分析技术造血祖细胞测序,胰腺细胞来自人类和小鼠胰岛地图册。在每种情况下,我们共同学习不同的或移行细胞状态的数据集,而提高通过综合分析统计能力。我们的方法促进scRNA-seq数据集的一般比较,可能加深我们的理解不同的细胞状态如何应对微扰,疾病,和演化。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
CDK8/19抑制起着重要的作用在胰腺β-cell归纳人类万能
干细胞研究与治疗开放获取2023年1月05
32-gene签名使用机器学习的发展对准确预测炎症性肠病
细胞再生开放获取2023年1月05
RNA单细胞测序揭示glioma-associated巨噬细胞极化的变化和细胞的恶性神经胶质瘤高AQP4的表达
癌症基因治疗开放获取2023年1月04
访问选项
订阅自然+
得到直接在线访问自然和其他55个自然杂志
29.99美元
每月
订阅杂志
得到完整的期刊访问1年
99.00美元
只有8.25美元的问题
所有价格是净价格。
增值税将被添加后在付款。
税计算期间将完成付款。
买条
时间有限或全文访问ReadCube。
32.00美元
所有价格是净价格。
引用
克莱恩,上午et al .液滴条码单细胞转录组应用于胚胎干细胞。细胞161年,1187 - 1201 (2015)。
Zilionis, r . et al .单细胞条码使用液滴微流体和测序。Protoc Nat。1244 - 73 (2017)。
Macosko E.Z. et al。高度并行的单个细胞的全基因组表达分析使用只滴。细胞161年,1202 - 1214 (2015)。
郑,G.X.Y. et al .大规模并行数字单个细胞的转录分析。Commun Nat。814049 (2017)。
Shekhar、k等。全面由单细胞转录组视网膜双极神经元的分类。细胞166年,1308 - 1323。e30 (2016)。
同时,A.-C。et al .单细胞RNA-seq揭示了新类型的人类血液树突细胞,单核细胞和祖细胞。科学356年eaah4573 (2017)。
杰尔et al。细胞命运决定的动力学和监管者pseudotemporal排序显示的单个细胞。生物科技Nat。》。32,381 - 386 (2014)。
韦尔奇,法学博士,Hartemink, A.J. & Prins, J.F. SLICER: inferring branched, nonlinear cellular trajectories from single cell RNA-seq data.基因组医学杂志。17106 (2016)。
Satija, R。,Farrell, J.A., Gennert, D., Schier, A.F. & Regev, A. Spatial reconstruction of single-cell gene expression data.生物科技Nat。》。33,495 - 502 (2015)。
Achim, k等。高通量的空间映射单细胞RNA-seq数据组织的起源。生物科技Nat。》。33,503 - 509 (2015)。
DeLaughter D.M. et al。单细胞解决时序基因表达在心脏发育。Dev细胞。39,480 - 490 (2016)。
Bendall,南卡罗来纳州et al .单细胞轨迹检测揭示发展和监管协调人类B细胞的发展。细胞157年,714 - 725 (2014)。
布莱克利正在,p . et al .定义三个由单细胞RNA-seq人类胚泡的细胞谱系。发展142年3613 (2015)。
约翰逊,M.B. et al。单细胞分析揭示转录在人类大脑皮层神经祖细胞的异质性。Nat。>。18行(2015)。
Regev, a . et al .人类细胞图谱。Elife6行(2017)。
Kharchenko,票面价值,Silberstein, L. & Scadden, D.T. Bayesian approach to single-cell differential expression analysis.Nat方法。11,740 - 742 (2014)。
Finak g . et al .桅杆:灵活的统计框架来评估转录变化和特征异质性RNA单细胞测序数据。基因组医学杂志。16278 (2015)。
王,B。,Zhu, J., Pierson, E., Ramazzotti, D. & Batzoglou, S. Visualization and analysis of single-cell RNA-seq data by kernel-based similarity learning.Nat方法。14,414 - 416 (2017)。
Kiselev V.Y. et al . SC3:共识单细胞RNA-seq数据的聚类。Nat方法。14,483 - 486 (2017)。
林,P。,Troup, M. & Ho, J.W.K. CIDR: Ultrafast and accurate clustering through imputation for single-cell RNA-seq data.基因组医学杂志。1859 (2017)。
普拉巴卡兰,S。,Azizi, E. & Pe'er, D. Dirichlet process mixture model for correcting technical variation in single-cell gene expression data.Proc。第33 Int,相依。马赫。学习。48,1070 - 1079 (2016)。
Ntranos, V。,Kamath, G.M., Zhang, J.M., Pachter, L. & Tse, D.N. Fast and accurate single-cell RNA-seq analysis by clustering of transcript-compatibility counts.基因组医学杂志。17112 (2016)。
徐,c &苏z细胞类型的识别从单细胞转录组使用一种新的聚类方法。生物信息学31日,1974 - 1980 (2015)。
Lei, Z。,Bai, Q., He, R. & Li, S.Z. Face shape recovery from a single image using CCA mapping between tensor spaces.26日设计。第一版。CVPR粘度模式识别doi: 10.1109 / CVPR.2008.4587341 (2008)。
周,F &老爹,F。先进的神经信息处理系统22;少量的2009 (eds。Y Bengio Y。,Schuurmans, D., Lafferty, J.D., Williams, C.K.I. & Culotta, A.)https://papers.nips.cc/paper/3728-canonical-time-warping-for-alignment-of-human-behavior(神经信息处理系统的基础上,Inc ., 2009)。
王,c &马哈,sProc。第二十二国际联合会议上人工智能,卷2 (ed。沃尔什,t .)1541 - 1546 (AAAI, 2011)。
黄,H。他,H。,Fan, X. & Zhang, J. Super-resolution of human face image using canonical correlation analysis.Recognit模式。43,2532 - 2543 (2010)。
霍特林,h .两组变量之间的关系。生物统计学28,321 - 377 (1936)。
哈同,是由,Szedmak, S. & Shawe-Taylor, J. Canonical correlation analysis: an overview with application to learning methods.神经第一版。16,2639 - 2664 (2004)。
威滕,D.M.,Tibshirani, R。&Hastie, T. A penalized matrix decomposition, with applications to sparse principal components and canonical correlation analysis.生物统计学10,515 - 534 (2009)。
Le曹K.-A。,Martin, P.G., Robert-Granié, C. & Besse, P. Sparse canonical methods for biological data integration: application to a cross-platform study.BMC生物信息学1034 (2009)。
Waaijenborg。,Verselewel de Witt Hamer, P.C. & Zwinderman, A.H. Quantifying the association between gene expressions and DNA-markers by penalized canonical correlation analysis.Stat .。麝猫。摩尔。杂志。7e3 (2008)。
Kettenring, j .的几组典型相关分析变量。生物统计学58,433 - 451 (1971)。
尼尔森,多重集典型相关性分析和多光谱,真正多瞬时遥感数据。IEEE反式。图像的过程。11,293 - 305 (2002)。
Berndt, d & Clifford j .使用动态时间扭曲在时间序列中找到模式。工作。"。"。越是加大。数据库398年,359 - 370 (1994)。
Kang莫莱森et al。多路复用滴单细胞RNA-sequencing使用自然的遗传变异。生物科技Nat。》。36,89 - 94 (2018)。
Nestorowa, s . et al .单细胞决议小鼠造血干细胞和祖细胞分化的地图。血128年e20-e31 (2016)。
保罗,f . et al .转录髓系祖细胞的异质性与血统的承诺。细胞163年,1663 - 1677 (2015)。
Adolfsson, j . et al .识别Flt3 + lympho-myeloid干细胞缺乏erythro-megakaryocytic潜在修订的路线图成人血血统的承诺。细胞121年,295 - 306 (2005)。
Lacar b . et al .正误表:核RNA-seq单一神经元激活的分子签名。Commun Nat。815047 (2017)。
波里,a . et al . CD56bright自然杀伤(NK)细胞:一个重要的NK细胞的子集。免疫学126年,458 - 465 (2009)。
男爵,m . et al .单细胞转录组的人类和小鼠胰腺地图揭示了国米,intra-cell人口结构。细胞系统。3,346 - 360。e4 (2016)。
Scheuner d &考夫曼R.J.展开蛋白质反应:有关β-cell衰竭和糖尿病患者胰岛素的需求。Endocr。牧师。29日,317 - 333 (2008)。
沃尔特,W。,Sánchez-Cabo, F. & Ricote, M. GOplot: an R package for visually combining expression data with functional analysis.生物信息学31日,2912 - 2914 (2015)。
江,H.-Y。et al .激活转录因子3是积分的真核起始因子2激酶压力反应。摩尔。细胞。医学杂志。24,1365 - 1377 (2004)。
爸爸,F.R.内质网压力,胰腺β-cell变性,和糖尿病。冷泉哈布。教谕。地中海。2a007666 (2012)。
Conesa, A . et al . RNA-seq的最佳实践的调查数据分析。基因组医学杂志。1713 (2016)。
约翰逊,真实再现,Li, C. & Rabinovic, A. Adjusting batch effects in microarray expression data using empirical Bayes methods.生物统计学8,118 - 127 (2007)。
•里奇、等人limma权力分析RNA-sequencing与微阵列研究微分表达式。核酸Res。43e47 (2015)。
湖,博et al .神经元亚型和多样性了single-nucleus RNA序列的人类大脑。科学352年,1586 - 1590 (2016)。
Ziegenhain, c . et al。RNA单细胞测序方法的比较分析。摩尔。细胞65年,631 - 643。e4 (2017)。
Svensson诉et al。权力分析单细胞RNA-sequencing实验。Nat方法。14,381 - 387 (2017)。
破车,摩根大通(J.P. et al。全基因组RNA断层在斑马鱼胚胎。细胞159年,662 - 675 (2014)。
李,J.H. et al .高度多路亚细胞RNA原位测序。科学343年,1360 - 1363 (2014)。
斯特尔,P.L. et al .可视化和分析空间转录组基因表达的组织部分。科学353年,78 - 82 (2016)。
Scialdone, a . et al .解决早期中胚层多元化通过单细胞分析表达式。自然535年,289 - 293 (2016)。
Tirosh, i . et al .解剖的多细胞生态系统由单细胞RNA-seq转移性黑素瘤。科学352年,189 - 196 (2016)。
Ilicic, t . et al。从单细胞RNA-seq数据分类的低质量的细胞。基因组医学杂志。17,29岁(2016年)。
Dudoit, S。,Fridlyans, J. & Speed, T.P. Comparison of discrimination methods for the classification of tumors using gene expression data.j。统计协会。97年,77 - 87 (2002)。
Tibshirani, R。Hastie, T。,Narasimhan, B. & Chu, G. Class prediction by nearest shrunken centroids, with applications to DNA microarrays.统计科学。18,104 - 117 (2003)。
Baglama, j . & Reichel l .增强隐式地重启兰索斯bidiagonalization方法。暹罗j .科学。第一版。(2005)。
Giorgino, t .计算和可视化动态时间扭曲排列在R: dtw包。Softw j . Stat。31日- 24 (2009)。
Waltman l . & Van Eck,新泽西州一个聪明的地方移动大规模modularity-based社区检测算法。欧元。理论物理。j·B86年1-33 (2013)。
Van Der Maaten, l .加速t-SNE使用基于树的算法。j·马赫。学习。Res。1521 (2014)。
理查兹,j . diffusionMap:扩散映射。(2014)https://cran.r-project.org/package=diffusionMap。
Hastie, t & Stuetzle w .主曲线。j。统计协会。84年502 (1989)。
年代最初是特雷弗Hastie R由Andreas Weingessel港口。princurve:适合主曲线在任意维度。https://cran.r-project.org/package=princurve(2013)。
曾,G.C.,Ghosh, D. & Feingold, E. Comprehensive literature review and statistical considerations for microarray meta-analysis.核酸Res。40,3785 - 3799 (2012)。
Kuleshov号et al . Enrichr:一个全面的基因集富集分析web服务器2016年更新。核酸Res。44W90-W97 (2016)。
Mayer, c . et al .皮质抑制性中间神经元的发展多元化。自然555年,457 - 462 (2018)。
Picelli, s . et al .全身使用Smart-seq2 RNA-seq从单个细胞。Protoc Nat。9,171 - 181 (2014)。
确认
我们感谢Satija实验室的成员,以及p . Roelli m . Stoeckius g . Fishell c . Desplan r·邦e . Macosko和a . Corvelo宝贵的反馈,和f . Hamey HM Kang j .你们寻求帮助与数据集出版。这项工作是由一个国家卫生研究院新创新者奖(1 dp2hg009623-01)和R01 (5 r01mh071679-12)和一个NSF研究生奖学金(DGE1342536)学士
作者信息
作者和联系
贡献
学士和R.S.构思研究。他还是,P.H., and R.S. implemented the alignment procedure, performed all data analysis, and wrote the manuscript. E.P. performed the PBMC validation experiments, and P.S. performed the ddSeq experiments.
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
权利和权限
关于这篇文章
引用这篇文章
巴特勒。,Hoffman, P., Smibert, P.et al。集成单细胞转录组数据在不同的条件下,技术,和物种。生物科技Nat》36,411 - 420 (2018)。https://doi.org/10.1038/nbt.4096
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nbt.4096
本文引用的
CDK8/19抑制起着重要的作用在胰腺β-cell归纳人类万能
干细胞研究与治疗(2023)
32-gene签名使用机器学习的发展对准确预测炎症性肠病
细胞再生(2023)
RNA单细胞测序揭示glioma-associated巨噬细胞极化的变化和细胞的恶性神经胶质瘤高AQP4的表达
癌症基因治疗(2023)
删除PD-1撼动了家族身份和肿瘤浸润调节性T细胞的代谢健身
自然免疫学(2023)
描述肿瘤微环境在单细胞水平揭示了小说在骨肉瘤免疫逃避机制
骨的研究(2023)
