摘要
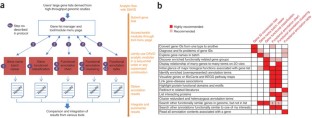
DAVID生物信息学资源包括一个集成的生物知识库和分析工具,旨在系统地从大型基因/蛋白质列表中提取生物学意义。该协议解释了如何使用DAVID,一个高通量和集成的数据挖掘环境,来分析从高通量基因组实验中获得的基因列表。该过程首先需要上传包含任意数量的通用基因标识符的基因列表,然后使用一个或多个文本和路径挖掘工具(如基因功能分类、功能注释图或聚类和功能注释表)进行分析。通过遵循这一协议,研究人员能够深入了解基因组规模研究中丰富的基因列表中的生物学主题。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
亮菌脂肪替代大豆油对肉仔鸡生产性能、消化率、盲肠微生物组、肝脏转录组及肝脏和血浆脂组的影响
动物科学与生物技术杂志开放获取2023年3月1日
microRNA对稻鱼一体化养殖鲫鱼金背突变体皮肤色素沉着的调控
BMC基因组学开放获取2023年2月10日
ERdj5在炎症条件下保护杯状细胞免受内质网应激介导的凋亡
实验与分子医学开放获取2023年2月9日
访问选项
订阅这本杂志
收到12个印刷问题和在线访问
每年209.00美元
每期只要17.42美元
租或购买这篇文章
只要这篇文章,只要你需要它
39.95美元
价格可能受当地税收的影响,在结账时计算


参考文献
黄达,W.等。DAVID生物信息学资源:扩展注释数据库和新算法,以更好地从大型基因列表中提取生物。核酸测定。35, w169-w175(2007)。
Dennis, G. Jr.等。DAVID:用于注释、可视化和集成发现的数据库。基因组医学杂志。4, p3(2003)。
Hosack, d.a., Dennis, G. Jr., Sherman, b.t., Lane, H.C. & Lempicki, R.A.在EASE基因列表中识别生物学主题。基因组医学杂志。4, r70(2003)。
齐伯格,B.R.等人。高通量GoMiner,用于解释多微阵列实验的“工业强度”集成基因本体工具,应用于常见可变免疫缺陷(CVID)的研究。BMC生物信息学6, 168(2005)。
Beissbarth, T. & Speed, T.P. GOstat:在一组基因中发现统计上过度代表的基因本体论。生物信息学20., 1464-1465(2004)。
Khatri, P., Bhavsar, P., Bawa, G. & Draghici, S. Onto-Tools:用于高通量基因表达实验功能设计和解释的网络可访问、基于本体的工具集成。核酸测定。32, w449-w456(2004)。
马丁等人。基于基因本体的基因数据集功能分析。基因组医学杂志。5, r101(2004)。
Al-Shahrour, F., Diaz-Uriarte, R. & Dopazo, J. FatiGO:一个用于发现基因本体论术语与基因组显著关联的网络工具。生物信息学20., 578-580(2004)。
Masseroli, M., Galati, O. & Pinciroli, F. GFINDer:遗传疾病和表型定位的统计分析和动态注释基因列表的挖掘。核酸测定。33, w717-w723(2005)。
Lee, j.s., Katari, G. & Sachidanandam, R. GObar:基于基因本体的基因集分析和可视化工具。BMC生物信息学6, 189(2005)。
萨勃拉曼尼亚,A.等。基因集富集分析:解释全基因组表达谱的一种基于知识的方法。Proc。国家的。学会科学。美国102, 15545-15550(2005)。
Khatri, P. & Draghici, S.基因表达数据的本体论分析:当前工具,局限性和开放问题。生物信息学21, 3587-3595(2005)。
谢尔曼,B.T.等。DAVID知识库:以基因为中心,整合异构基因注释资源,促进高通量基因功能分析。BMC生物信息学8, 426(2007)。
黄达,W.等。DAVID基因功能分类工具:一种以生物模块为中心的新型算法,用于功能分析大型基因列表。基因组医学杂志。8, r183(2007)。
黄,d.w., Sherman, B.T.和Lempicki, R.A. DAVID基因ID转换工具。信息学手段2, 428-430(2008)。
Cicala, C.等。HIV包膜在非增殖靶细胞中诱导一系列有利于病毒复制的细胞信号。Proc。国家的。学会科学。美国99, 9380-9385(2002)。
确认
我们非常感谢裁判的建设性意见,并感谢ABCC组的Robert Stephens, David Bryant和David Liu对Web服务器的支持。感谢免疫发病机制与生物信息学实验室(LIB)组的郑鑫和杨军的讨论。我们也感谢Bill Wilton和Mike Tartakovsky在信息技术和网络方面的支持。该项目由美国国家过敏和传染病研究所(NIAID)和国立卫生研究院(NIH)提供的联邦资金资助,合同编号为:一号门将- co - 56000。本工具和出版物的注释不一定反映美国卫生与公众服务部的观点或政策,也不意味着提及商品名称、商业产品或组织就意味着得到美国政府的认可。
作者信息
作者及隶属关系
相应的作者
补充信息
补充数据
的集合∼68个类似的富集分析工具。这些工具根据其后端算法大致分为三类。提供了关于每个工具的更多信息的参考链接。(xls101kb)
补充数据
∼400 Affymetrix IDs16用于本文(XLS 71 kb)
补充数据
从微阵列研究获得的基因列表与随机生成的相同大小基因列表之间的富集p值的比较。一个“好的”基因列表应该始终包含比相同大小的随机列表更丰富的生物。(PDF 383kb)
补充数据
DAVID系统支持的基因标识符类型和注释类别摘要。(xls38kb)
补充数据
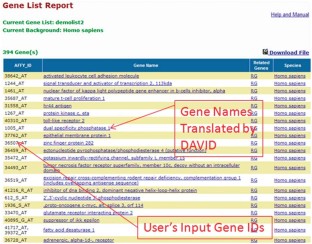
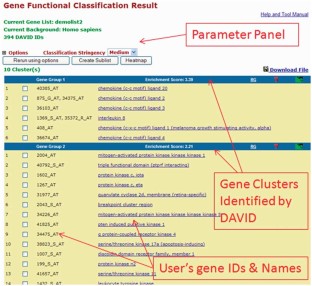
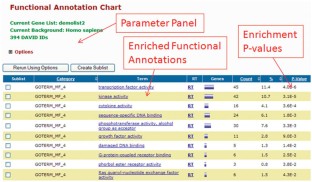
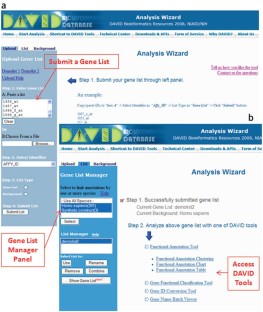
根据手稿中的描述,各主要分析步骤的截图。(PDF 1600kb)
补充数据
基因列表的输入格式示例。(PDF 69kb)
权利和权限
关于本文
引用本文
黄D,谢尔曼,B. & Lempicki, R.利用DAVID生物信息学资源对大型基因列表进行系统和综合分析。Nat Protoc4, 44-57(2009)。https://doi.org/10.1038/nprot.2008.211
发表:
发行日期:
DOI:https://doi.org/10.1038/nprot.2008.211
这篇文章被引用
空间相关性对甲基化熵的影响及其在小鼠脑甲基化组中的应用
表观遗传学与染色质(2023)
亮菌脂肪替代大豆油对肉仔鸡生产性能、消化率、盲肠微生物组、肝脏转录组及肝脏和血浆脂组的影响
动物科学与生物技术杂志(2023)
microRNA对稻鱼一体化养殖鲫鱼金背突变体皮肤色素沉着的调控
BMC基因组学(2023)
间皮瘤相关成纤维细胞通过c-Met/PI3K和WNT信号通路增强胸膜间皮瘤细胞的增殖和迁移,但对顺铂没有保护作用
实验与临床癌症研究杂志(2023)
ERdj5在炎症条件下保护杯状细胞免受内质网应激介导的凋亡
实验与分子医学(2023)
