摘要
数以百计的临床研究已经证明了人类微生物组和疾病之间的联系,但基本的问题仍然是我们如何概括这些知识。个别研究的结果可能不一致,由于缺乏标准的处理和分析方法,比较已发表的数据更加复杂。在这里,我们介绍MicrobiomeHD数据库,其中包括28项已发表的病例对照肠道微生物组研究,涵盖10种疾病。我们使用标准化方法对这些研究进行了跨疾病荟萃分析。我们发现了与疾病相关的微生物组变化的一致模式。一些疾病与50多个属相关,而大多数疾病仅显示10-15个属级的变化。一些疾病以潜在致病微生物的存在为特征,而另一些疾病则以健康相关细菌的耗尽为特征。此外,我们表明,与个别研究相关的大约一半属是对一种以上疾病有反应的细菌。因此,在病例对照研究中发现的许多关联可能不是疾病特异性的,而是对健康和疾病的非特异性、共同反应的一部分。
简介
人的胃肠道消化食物,吸收营养物质,并在维持代谢稳态中发挥重要作用。居住在我们肠道中的微生物从我们所吃的食物中获取能量,训练我们的免疫系统,分解异种生物和其他外来产品,释放对调节我们生理机能很重要的代谢物和激素1,2,3..来自我们微生物群的化学信号可以在肠道内局部起作用,也可以产生更大的系统性影响(例如,“肠道-大脑轴”)。4,5,6.
由于人类和我们的微生物群落之间的生理相互作用,许多疾病被假设与偏离“健康”的肠道微生物组有关。这些疾病包括代谢紊乱、炎症和自身免疫疾病、神经系统疾病和癌症等1,3.,7,8,9.某些肠道相关疾病(如肥胖和炎症性肠病)已在人类队列和动物实验中进行了广泛研究,其中已显示出显著的、有时是因果性的微生物关联。这些研究刺激了对一些病因不明的复杂疾病的研究,这些疾病被怀疑与微生物组有关。
总的来说,我们目前对人类肠道微生物组和疾病之间精确关系的理解仍然有限。现有的病例对照研究经常报告发现与疾病相关的微生物“生态失调”。然而,术语“生态失调”是不一致的,往往是模糊的定义,可以有广泛的解释10,11.因此,我们缺乏对微生物群落以及这些群落中的特定微生物是如何引起、响应或促进疾病的全面了解。不同的疾病是否以肠道微生物群的不同变化为特征?是否有些疾病以病原体入侵为特征,而另一些疾病则表现为有益细菌的耗尽?我们能否识别某些条件下的微生物生物标记物,这些标记物在许多患者队列中在一种疾病中持续增加或减少?最后,某些细菌是否属于非特定的“健康”或“患病”微生物群,并始终与健康或疾病有关?
综合现有知识的一种方法是通过元分析确定各研究之间的一致性,这使研究人员能够发现并消除可能掩盖潜在生物学模式的假阳性和假阴性。然而,先前对病例对照肠道微生物组研究的荟萃分析得出了复杂的结果,并且没有将其发现纳入多种疾病的背景12,13,14.对于一些疾病,如炎症性肠病(IBD),在几项研究中发现了肠道微生物群的整体差异,但在所有研究中,没有单个微生物与IBD一致相关12.对于肥胖等其他疾病,多项荟萃分析发现,肥胖患者和瘦人患者的肠道微生物群几乎没有差异12,13,14尽管在小鼠模型中,微生物组与肥胖有因果关系3.,15.这些荟萃分析仅限于关注一种或两种疾病,因此不能将其发现扩展到更广泛的人类疾病领域,以回答有关疾病相关微生物群变化的总体模式的更一般的问题。
在本文中,我们收集了28个已发表的病例对照16S扩增子测序肠道微生物组数据集,涵盖10种不同的疾病状态。我们为每项研究获取原始数据和疾病元数据,并系统地重新处理和重新分析数据。我们调查了是否可以在同一疾病的多项研究中确定肠道微生物群落中一致和特定的疾病相关变化。某些疾病(如结直肠癌)以疾病相关细菌的富集为特征,而其他疾病(如IBD)则以健康相关细菌的耗尽为特征。一些疾病(如腹泻)表现出与许多相关微生物的大规模群落转移,而大多数疾病只表现出少数几个关联。然而,许多关联不是特定于个别疾病,而是对多种疾病状态作出反应。在大多数研究中,大多数与疾病相关的个体微生物都是这组细菌的一部分,它们对健康和患病状态没有特异性反应。因此,来自个别病例对照研究的相关性应谨慎解释,因为这些微生物可能表明对疾病的共同反应,而不是疾病特异性差异的一部分。总之,这些发现揭示了不同类型的生态失调,这可以为基于微生物的诊断和治疗的发展提供信息。
结果
大多数疾病表现为微生物群的改变
为了回答关于已报道的人类微生物组与疾病之间关联的可重复性和可泛化性的问题,我们收集、重新处理和重新分析了来自微生物组数据集的原始数据。我们纳入了至少15例病例患者粪便样本中公开可用的16S扩增子测序数据(即FASTQ或FASTA)的研究,这些研究也具有相关的疾病元数据(即病例或对照疾病标签)。专门针对5岁以下儿童的研究被排除在我们的分析之外。我们确定了超过50个合适的病例对照16S数据集,其中28个成功下载、处理并包含在一个公开可用的数据库中,我们称之为MicrobiomeHD16.这些数据集的特征,包括样本量、疾病和状况以及参考文献见表1及补充表1.对于每个下载的研究,我们通过我们的16S处理管道处理原始测序数据(https://github.com/thomasgurry/amplicon_sequencing_pipeline)(见补充表格2而且3.详细的数据来源和处理方法)。100%新生OTUs用RDP分类器进行分类17(c= 0.5),除以总样品读数折算为相对丰度,折算为属水平。未在属水平分配的OTUs被丢弃。通过将数据压缩到属水平,我们失去了在病例组和对照组之间检测物种或菌株丰度细微差异的敏感性,但我们最小化了在研究之间进行比较的某些批量效应。因此,我们采取了一种粗粒度的方法,以牺牲系统发育分辨率为代价,优化我们比较各研究数据的能力。
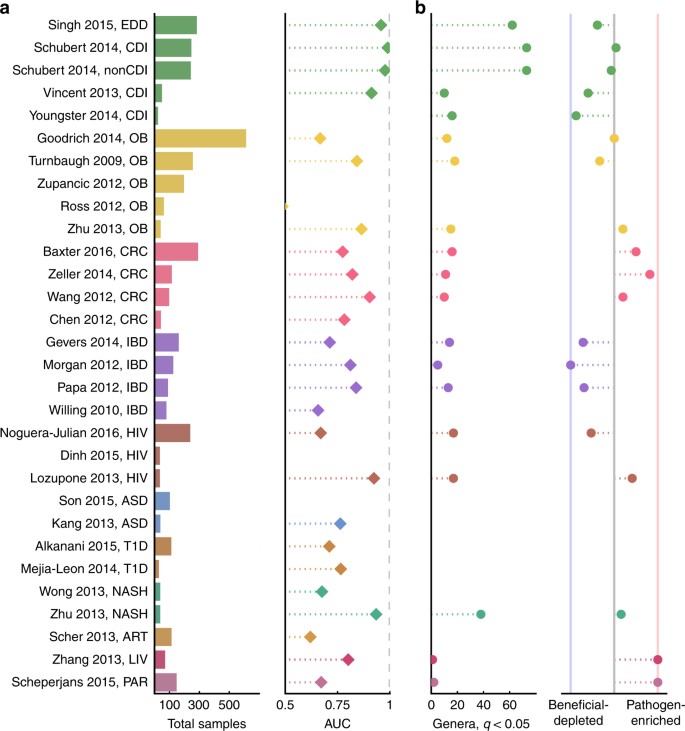
我们首先提出的问题是,一旦我们控制了处理和分析方法,报道的肠道微生物组和疾病之间的联系是否会被重现。为了测试肠道微生物组是否在各种疾病状态下发生改变,我们建立了属级随机森林分类器,以将每项研究中的病例从对照中分类。我们比较了各个研究中受试者工作特征(ROC)曲线(AUC)下的结果面积。1和补充图。1).我们可以对除关节炎和帕金森病以外的所有疾病的至少一个数据集的对照病例(AUC > 0.7)进行分类,每种疾病都只有一项研究。值得注意的是,所有腹泻数据集(除了young et al。18(只有4名不同的对照患者,因此未纳入本分析)具有非常高的可分类性(AUC > 0.9)。我们在四项IBD研究中的三项和所有四项CRC研究中成功地将患者与对照组(AUC > 0.7)进行了分类,这与之前的工作一致,表明使用监督分类方法可以很容易地将这些患者与对照组区分开来12,19,20.,21.因此,在许多不同的疾病中,微生物群确实发生了改变。
大多数疾病都表现出微生物组的改变,一致的疾病相关的变化在程度和方向上有所不同。一个左:这些分析中包含的每个研究的总样本量。关于每个数据集的其他信息可以在表中找到1.研究y-轴按疾病分组,并按样本量递减排序(从上到下)。右:属级随机森林分类器的ROC曲线下面积(AUC)。X-axis从0.5开始,随机分配标签的分类器的期望值,并且不显示小于0.5的auc。所有数据集的ROC曲线见补充图。1.注意young等人。18只有四个不同的对照患者被排除在随机森林分析之外。b左:属数问对于每个数据集< 0.05 (Kruskal-Wallis (KW)检验,Benjamini-Hochberg FDR校正)。如果一项研究没有显著的关联,就没有意义。右:微生物群转移的方向,即在患病患者中富集的总相关属的百分比。在最左边蓝线的数据集中,100%的相关(问< 0.05, FDR KW检验)属与健康相关(即患者相对于对照组减少)。在最右边红线的数据集中,100%的相关(问< 0.05, FDR KW检验)属与疾病相关(即患者相对对照组丰富)。补充无花果。14而且15显示问每项研究中每个属的值和影响
有益微生物的丧失或病原体的增多
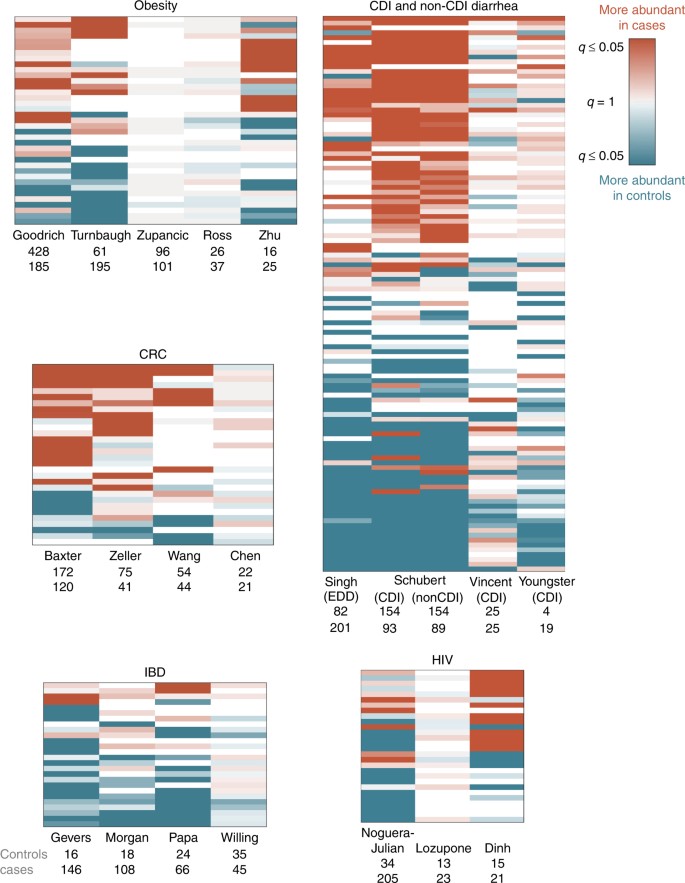
接下来,我们想知道这种特定类型的改变在患有同一疾病的独立队列患者中是否一致。我们对每个数据集分别进行了属级相对丰度的单变量检验,并比较了各研究的结果(Kruskal-Wallis (KW)检验与Benjamini-Hochberg错误发现率(FDR)校正)22).我们对这些研究的重新分析在很大程度上与最初报道的结果一致。尽管数据处理方法不同,但相同的分类组显示出与原始出版物相似的趋势(见补充说明)1对于我们的重新分析与以前发表的结果的完整比较)。此外,我们发现微生物组中与疾病相关的变化可以分为有意义的组,这为不同类型疾病的可能病因或治疗策略提供了见解。
在某些疾病中,微生物群的变化主要是由少数“致病性”细菌的富集所主导的。在这些情况下,微生物可能发挥了因果作用,可以用窄谱抗微生物药物靶向它们。结直肠癌的特征就是这种转变,我们发现四项CRC研究中的三项具有显著的一致性8,20.,21,23(无花果。1b和2,补充图中的属标签。2).与结直肠癌相关的生态失调通常以已知病原或病原相关的发病率增加为特征梭菌属,Porphyromonas,消化链球菌属,Parvimonas,肠杆菌属属(即,在两个或多个研究中,这些属在CRC患者中较高,图。2而且3,补充图中的属号。2而且3.).梭菌属与广泛的人类疾病和Porphyromonas是否有已知的口腔病原体24,25.
比较来自同一疾病的多项研究的结果,揭示了疾病相关微生物组改变的模式。显示日志的热图10(问(KW检验,Benjamini-Hochberg FDR校正)。行包括在每种疾病的至少一个数据集中具有显著性的所有属,列为数据集。问数值按效果的方向着色,其中红色表示疾病患者的平均丰度较高,蓝色表示对照组的平均丰度较高。不透明度范围为问= 0.05-1,其中问小于0.05的值是最不透明的问接近1的值为灰色。白色表示该属在该数据集中不存在。在每个热图中,行顺序从最与疾病相关的(上)到最与健康相关的(下)(即,按日志各行之和排序)10(问值),根据效果的方向进行标记)。疾病相关微生物群的变化程度可以通过每个疾病热图中的行数来可视化;变化的方向性可以从每种疾病中红行与蓝行的比例中看出。见补充图。2用于属(行)标签
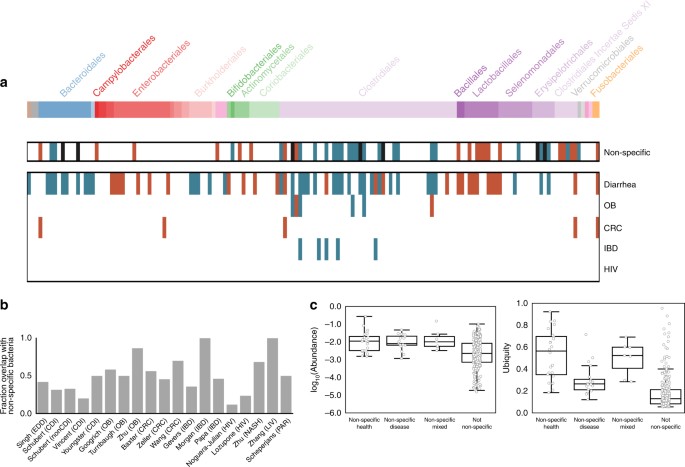
大多数与疾病相关的微生物组关联与对疾病的非特异性微生物反应重叠。一个非特异性和疾病相关属。属以列表示,根据由属级NCBI id建立的PhyloT树进行系统发育排列(http://phylot.biobyte.de).非特异性属与至少两种不同疾病的健康(或疾病)有关(问< 0.05, KW检验,Benjamini-Hochberg FDR校正)。在至少两项对同一疾病的研究中,疾病特异性属在同一方向上具有显著意义(问< 0.05, FDR KW试验)。如Fig。2,蓝色表示对照组的平均丰度较高,红色表示患者的平均丰度较高。黑色条形表示混合属,与两种疾病的健康相关,也与两种疾病的疾病相关。至少有三项研究显示疾病特异性属。门,从左到右:真古菌门(棕色),Verrucomicrobia Subdivision 5(灰色),Candidatus Saccharibacteria(灰色),拟杆菌门(蓝色),变形菌门(红色),辅菌门(粉红色),放线菌门(绿色),厚壁菌门(紫色),Verrucomicrobia(灰色),Lentisphaerae(粉红色),梭杆菌门(橙色)。见补充图。3.属标签。b每项研究的属级关联与共享反应重叠的百分比(问< 0.05, FDR KW试验)。只显示至少有一个重要关联的数据集。c总体而言,在所有数据集中的所有患者中,非特异性属的丰度和普遍性。非特异性属上x-轴的定义如上所述
相比之下,其他与疾病相关的微生物群变化的特征是患者相对于对照组的健康相关细菌的减少。在这些情况下,替代缺失类群的益生菌可能是比抗微生物药物更好的治疗策略。在我们的四项IBD研究中,相对于对照组,患者的微生物群落以消耗属为主,特别是丁酸盐产生梭菌属的19,26,27,28(无花果。1 b而且2,补充图中的属标签。2).具体来说,有五个属Ruminococcacaea而且Lachnospiracaea至少在两项研究中,IBD患者相对于对照组的家族性持续减少(图2)。3,补充图中的属标签。3.).而不是所有属内Ruminococcacaea而且Lachnospiracaea是短链脂肪酸(SCFA)的生产者,这些家族中的主要属是否含有短链脂肪酸的生产基因29通常与结肠健康有关30.,31,32.我们在分别比较克罗恩病和溃疡性结肠炎患者与对照组时发现了类似的结果,但在数据集上没有任何一致的模式来区分IBD亚型(补充说明)2;补充无花果。4而且5).
一些疾病的特点是肠道微生物群落的广泛重组。在这些情况下,完整的社区恢复策略,如粪便微生物群移植,可能更合适。例如,腹泻持续导致肠道微生物组组成的大规模重排,这可能反映了粪便运输时间的减少(图2)。1而且2).我们发现许多微生物都与两者有关艰难梭状芽胞杆菌感染(CDI)和非CDI腹泻(图;2而且3)18,33,34,35.一般来说,变形菌门在腹泻患者中的患病率增加,同时拟杆菌门和一些厚壁菌门的相对丰度降低。特别是,我们看到了产生丁酸盐的梭状芽孢杆菌的减少,包括其中的属Ruminococcaceae而且Lachnospiraceae家庭,这与健康的肠道有关36.我们还看到,含有与低pH值和上肠道高氧水平有关的生物体的属的流行度有所增加,比如乳杆菌科而且肠杆菌科,在腹泻患者(图;3)37.此外,在所有研究中,CDI和非CDI腹泻患者的alpha多样性(衡量整体社区结构的指标)均低于健康对照组(补充图)。6- - - - - -8).与CDI和非CDI腹泻研究一致的是,我们还发现与上肠道有关的生物体,比如乳酸菌而且肠杆菌科,在IBD患者中似乎丰富,他们可以表现出腹泻症状(补充图。2)37,38.IBD患者的α多样性也往往低于对照组(3项研究中克罗恩病与对照组,2项研究中溃疡性结肠炎与对照组;补充无花果。6- - - - - -8),尽管这种差异没有在所有患者都有活动性腹泻的腹泻研究中那么明显。
在一些研究中,混杂变量可能会导致关联。例如,在艾滋病毒研究中,病例和对照组之间没有一致的差异,因为证明了混杂因素39,40,41(无花果。2而且3).就像最初的Lozupone等人。40学习中,我们发现充实普氏菌,Catenibacterium,Dialister,脱磷孤菌属在hiv阳性患者中,除了8个其他属(图。2和补充图。2).我们还发现了拟杆菌,Odoribacter,Anaerostipes,Parasutterella,Alistipes艾滋病毒阳性患者相对于对照组。然而,诺格拉-朱利安等人。39研究表明,Lozupone论文中与HIV显著相关的属与性行为密切相关(例如,与男性发生性关系的男性与更高的性行为相关普氏菌水平),我们的重新分析也发现这两项研究的结果相互矛盾(图。2).因此,在哪些属与艾滋病毒相关方面没有共识。肥胖是另一个混杂变量可能导致微生物群改变的例子。最近的三项荟萃分析发现,与肥胖相关的微生物群没有可重复的变化12,13,14这与我们的分类结果是一致的,我们只能在五分之二的研究中准确地将肥胖患者和对照组患者进行分类(Zhu et al。1Turnbaugh等人。42;无花果。1).我们的属级再分析确实发现了瘦和肥胖患者之间的一些一致的属级关联1,42,43,44,45.两个属,Roseburia而且Mogibacterium,在两项肥胖研究中,在肥胖个体中显著增加(图2)。3).此外,Anaerovorax,Oscillibacter,Pseudoflavonifractor,四梭状芽胞杆菌在其中两项研究中,肥胖患者相对于对照组的脂肪消耗殆尽。然而,五项研究中有两项没有显著的属级关联(问< 0.05),尽管有一个大样本量(Zupancic et al。44).这表明,饮食等混杂因素可能导致了我们在重新分析中发现的某些关联,以及之前在文献中报道的某些关联14.对于肥胖和艾滋病等疾病,需要进行更多控制潜在混杂因素(如宿主行为和饮食)的研究,因为这些疾病与微生物群的关系尚不清楚。最后,病例对照组的患者经常服用其他药物,如抗生素,这可能会混淆与疾病相关的微生物群变化。我们的六个数据集包含抗生素元数据,其中只有一个数据集(Schubert et al。33)中有5个以上的对照组在服用抗生素。因此,经常使用抗生素治疗的疾病相关属(如腹泻、IBD)很可能与抗生素使用混淆。未来的病例对照研究应该通过收集抗生素和其他药物使用的详细元数据,或者通过招募接受各种治疗的对照组,来更好地分离治疗和疾病变量。
共有的vs.疾病特有的微生物反应
最后,我们试图确定是否可以确定一个统一的微生物组对一般健康和疾病的反应。先前的研究提出,alpha多样性的降低是疾病相关生态失调的可靠指标34,42,46.在我们的重新分析中,我们发现除了腹泻和可能的IBD之外,病例患者的alpha多样性没有一致的降低(补充图)。6- - - - - -8).这些结果与之前的荟萃分析一致,后者发现α多样性与疾病之间的关系不一致,在非腹泻疾病中效应值非常小12,13.为了进一步解决我们是否可以为患病微生物群找到一个强大的、广义的信号而不管疾病类型的问题,我们构建了随机森林分类器来区分健康患者和任何类型的病例患者。来自这些一般健康与疾病分类器的auc与原始单一数据集分类结果密切相关,这表明确实存在一个甚至可以在不同疾病中识别的一般微生物组信号(见补充说明)3.和补充图。9).
假定存在对疾病的普遍微生物反应后,我们接下来试图确定对健康和疾病有非特异性反应的个别属。我们认为一个属是非特异性的,共享的微生物反应的一部分,如果它显著富集或减少(问< 0.05)来自至少两种不同疾病的至少一个数据集(见补充说明)4和补充图。10而且11为了进一步讨论共享响应的替代定义和统计学意义)。我们在152个属中鉴定出24个健康相关属和20个疾病相关属,这些属在至少一个数据集中是显著的(图2)。3,补充图中的属标签。3.).我们还发现了7个与健康和疾病相关的属(即,它们在至少两种疾病的对照中富集,但在至少两种疾病的不同比较中,它们在对照中也被耗尽)(图2)。3、黑色)。也许这些属代表的细菌不成比例地受到混杂因素或技术人为因素的影响。另外,这些属中的不同物种或菌株可能在疾病或社区环境中发挥替代作用,在属水平上产生不同的反应。
我们发现了不同的微生物亚群拟杆菌门而且厚壁菌门对健康和疾病无特异性反应的门(图。3).订单梭菌属的(具体Lachnospiraceae而且Ruminococcacaea家庭)与多种疾病的健康相关,而顺序Lactobacillales和家人梭状芽孢杆菌都与疾病有关大多数非特定反应者都是按顺序排列的梭菌属的与健康相关,包括与健康患者非特异性相关的所有微生物的大多数(24个总健康相关属中的17个属)。按顺序排列的五个非特定应答者Lactobacillales在多种疾病的病例中得到了丰富。Lactobacillales属适应上消化道pH值较低的环境37.也许共享的疾病相关分类群是粪便转运时间较短和氧化还原状态和/或下肠pH值中断的指标,而不是特定的病原体。这些非特异性应答者与最近对六个宏基因组数据集的荟萃分析的结果一致,该分析还发现Lactobacillales而且梭菌属的在多项研究中,微生物是最具鉴别性的分类特征之一47.最后,我们发现了这个顺序细菌性的更混合:两个细菌性的属与健康无关,一个属与疾病有关,两个属与健康和疾病都有关。
个别研究中的大多数细菌关联与共同反应重叠。对于每个数据集至少有一个显著的(问< 0.05)的相关性,我们计算了相关属的百分比,这些属也是同一方向非特异性反应的一部分(图。3 b).引人注目的是,大多数微生物反应并不针对个别疾病;平均而言,数据集中51%的属级关联是与一种以上疾病相关的属。根据这一发现,重要的是,研究人员在进行未来病例对照研究时,要考虑已确定的微生物关联是否真的针对他们感兴趣的疾病,或者只是对一种常见症状(例如腹泻)作出反应,或者可能通常与健康或疾病有关。此外,他们可以利用许多微生物对疾病的非特异性反应这一知识,将假定的因果或诊断生物标记物缩小到不属于共同反应的微生物,因此更有可能对所研究的疾病具有特异性。研究人员可以从MicrobiomeHD数据库的分析中访问共享微生物反应者的更新列表16或者,他们也可以通过类似的跨疾病荟萃分析来整理自己的清单。
与健康无关的细菌在人群中普遍存在且数量丰富,而与疾病无关的细菌虽然大量存在,但并不普遍存在。我们计算了共享反应中每个属的平均相对丰度(即所有患者的总相对丰度除以非零丰度的患者数量)和普遍性(即非零丰度的患者数量除以患者总数)。我们发现与健康相关的属比与疾病相关的属更普遍,但不一定更丰富(图2)。3 c).因此,非特异性疾病相关属的存在/缺失似乎是疾病相关微生物转移的更好指标,而不是其相对丰度的变化。然而,一小部分非特异性疾病相关属在患者中相对普遍存在。其中最普遍的是大肠杆菌、志贺氏杆菌而且链球菌.埃希氏杆菌属包括常见的共生菌株和致病菌株48,它经常存在于健康人的肠道中,在病人体内也过多。属内肠杆菌科,乳杆菌科,链球菌科家族在上消化道占主导地位37,49并以低频率出现在许多人的粪便中。这些类群可能随着粪便转运时间的加快而变得丰富(即腹泻的特征)。37,50.
疾病内部和跨疾病元分析提高了可解释性
确定疾病特异性和非特异性微生物反应需要在多种疾病内部和跨多种疾病进行比较研究。需要对同一疾病进行多项研究,以确定与个别疾病一致相关的变化。在少于四个数据集的情况下,我们没有发现一致的细菌关联(图2)。1而且3).疾病内荟萃分析也提高了我们从任何一个数据集解释结果的能力。尽管没有显著差异,但其中一些研究(如Zhang et al。51,朱等。1)与对照组相比具有较高的可分类性(AUC > 0.7,图;1),表明可能存在单变量比较未检测到的疾病相关转移。然而,由于很少有其他关于同一疾病的研究可供比较,我们不能自信地解释超出报告的AUC的分类结果。对于其他具有高auc但很少有单变量相关性的研究(例如,Vincent等。34摩根等。27等。23),我们对高auc反映真实疾病相关差异的信心增加,因为高auc与来自同一疾病类型的其他分类器一致。
元分析确定了研究和条件中潜在的假阳性和假阴性。例如,我们发现在个别研究中报告的alpha多样性与疾病之间的关联在研究中往往失去了意义,除了腹泻和IBD(补充图)。6- - - - - -8).另一个潜在假阳性的例子是普氏菌和疾病。自闭症2类风湿性关节炎52,以及爱滋病病毒40,41每一种都被报道与普氏菌.对于每一种疾病,与普氏菌显著性较弱或因混杂因素而复杂化。在我们统计上保守的重新分析中,我们发现自闭症或关节炎之间没有联系普氏菌.如前所述,在艾滋病毒的情况下,与普氏菌是因为与疾病无关的人口因素吗39.不管是否平移普氏菌虽然与每一种被研究的疾病状态确实存在生物学上的联系,但很明显,这种变化并不特定于一种特定的疾病,不应该作为假定的疾病特异性生物标志物报告。我们还发现,通过元分析选出的某些信号并不总是在个别研究中成立。例如,小样本量的研究通常很少或没有显著的相关性(例如,Vincent等。34, Chen等。23和Willing等人。28).在这里,其他分析相同疾病的研究一致发现了相关性,这一事实加强了这一假设,即这些研究中缺乏微生物组相关信号是由于低功率而不是缺乏真正的信号。由于个体研究受到低统计能力、混杂变量和可能掩盖生物信号的批效应的困扰,随着更多的数据集和疾病被纳入未来的荟萃分析,对疾病特异性和非特异性微生物关联的识别将继续改善。
讨论
在这里,我们报告了人类肠道微生物组中与疾病相关的变化模式,其方向性(即,疾病富集属与疾病耗尽属的比例)和程度(即,病例和对照组之间不同的属的总数)有所不同。一些疾病的特征是致病或疾病相关细菌的入侵(如CRC),而其他疾病主要表现为健康相关微生物的耗尽(如IBD)。腹泻疾病会引起许多微生物群成员的大规模重新排列,而其他疾病则表现出较少的关联。我们还发现了一组与多种疾病非特异性相关的微生物,并表明在任何给定的研究中,这些微生物包括许多与疾病相关的属。
非特异性微生物反应的识别是未来病例对照微生物组研究中应该考虑的一个重要概念。它表明,应格外谨慎地解释这些研究,因为许多已确定的微生物关联可能表明对健康或疾病的共同反应,而不是特定疾病的生物学差异。与多种疾病非特异性相关的微生物将无法用于疾病特异性诊断或解决因果关系10.另一方面,与多种疾病的健康患者相关的细菌可以被开发成一种适用于许多不同疾病的通用益生菌。
此外,通过方向性和程度来表征“失调”是一个有用的框架,可以为未来研究与微生物组有关的复杂、异质性疾病提供假设。例如,对基于微生物组的诊断的搜索可能更适合于与疾病相关的微生物持续富集的疾病,如CRC。另一方面,患有以健康相关微生物消耗为特征的疾病的患者,如IBD,可能受益于旨在丰富这些分类群的益生元或益生菌干预措施。此外,以群落结构大规模变化为特征的条件可能非常适合用粪便微生物群移植进行治疗,如CDI18.虽然这些疾病中的许多不太可能通过抗生素、益生菌或粪便微生物群移植来完全治疗,但我们提出的框架可以通过产生可测试的假设来指导寻找新的治疗方法和病因,这些假设具有更高的成功可能性10.
这项分析首次比较了两种以上不同疾病的微生物组研究,并强调了公开原始数据和相关患者元数据的重要性,以实现未来更全面的分析。这一分析没有包括所有可能的研究,某些重要的胃肠道疾病(如肠易激综合征)没有纳入,这主要是由于数据和元数据的可用性。未来的研究应该扩大这项工作,包括来自相同疾病和更多疾病的更多队列。为了重新分析这些研究,我们应用了该领域常用的标准方法,并假设原始的研究设计和患者选择方法是足够的。我们很放心地发现,一种直接和标准化的方法能够恢复与以前在各种论文中报告的结果非常相似的结果。因此,我们没有正式调查队列间的异质性或技术上的研究批效应。然而,从我们的属级结果可以清楚地看出,即使在对同一疾病的研究中,也存在显著的差异。造成这种变化的原因有很多(实验和测序伪影、宿主相关协变量、随机疾病相关的群落变化等)。11,53,54),未来的分析应考虑纠正宿主混杂因素和技术批效应的方法。对批效应的关注促使我们在属水平上分析数据,这必然限制了我们对已识别关联的分辨率和生物学解释(例如,一个属内的不同物种或菌株可能与疾病有不同的关联,这在本分析中不会被捕捉到)。公开病例对照研究的原始数据,除了使未来的荟萃分析更全面外,还将允许研究人员开发纠正这些批量效应的方法。
尽管这项研究存在局限性,但我们的结果对生态失调提供了更细致的见解,揭示了更精确地描述疾病相关微生物群变化的不同类型的改变。随着病例对照队列数量的增加,类似的荟萃分析可用于比较相关疾病,并确定与一般宿主生理变化相关的微生物组改变。例如,可能有一组微生物对全身性炎症有反应或引起全身性炎症。我们能否通过比较多种炎症或自身免疫疾病来识别这些微生物,并对它们进行研究,以更好地了解微生物组和我们的免疫系统之间的相互作用?此外,一些微生物可能始终与神经系统疾病相关,并可能导致伴随或先于神经系统症状的胃肠道症状2,9.研究这些微生物可以通过识别这些细菌产生的常见神经活性分子来帮助我们理解“肠道-大脑轴”,这些分子也可以用作新治疗的靶标4,5,6.最后,荟萃分析可用于确定在IBD等异质性疾病中表现出明显微生物组转移的患者亚群,或在表现出随机微生物反应的情况下,允许进一步分层疾病亚型和微生物组破坏11,28,55.这项工作表明,在临床相关微生物组研究的更广泛的领域内,采用标准方法将新结果纳入背景是可行的,并为个别分析增加了价值。随着人们对这一领域的热情不断增长,研究人员应该利用越来越多的重复病例对照研究,迅速而有效地推进微生物组科学从假定的关联到变革性的临床影响。
方法
数据集收集
我们从PubMed的关键词搜索中,通过参考元分析和相关病例对照研究,确定了病例对照16S研究。我们纳入了公开可用的原始16S数据(fastq或fasta)的研究,以及每个样本的病例或对照状态的元数据。大多数数据是从在线存储库(例如SRA)或原始出版物中提供的链接下载的,但一些数据是在与作者个人沟通后获得的(补充表)3.).我们没有纳入任何需要额外伦理委员会批准或获取授权的研究(例如,对照dbGaP研究)。在对多个身体部位取样或对每个患者采集多个样本的研究中,我们还要求各自的元数据包含这些元数据。我们只分析了粪便16S样本,排除了少于15例病例的研究。在有多个对照组的CRC研究中(例如,健康和非CRC腺瘤),在我们的所有比较中,只有健康患者作为对照。在非健康对照(如非ibd患者)的研究中,这些患者被用作对照(如原始论文中所述)。在舒伯特等人的作品中。CDI研究33其中有CDI和非CDI腹泻患者,每组都作为独立的病例组与对照组进行比较。我们还分析了Zhu等人研究中的NASH和肥胖患者1作为独立的案例组。当肥胖研究报告身体质量指数而不是肥胖状况时,我们将BMI小于25的患者作为对照组,BMI大于30的患者作为病例组。
16 s处理
原始数据通过我们内部的16S处理管道下载和处理(https://github.com/thomasgurry/amplicon_sequencing_pipeline).数据和元数据的获取详见补充表3..当需要时,我们通过找到与所提供的条形码的精确匹配来对序列进行多路复用,并通过最多一个不匹配来修剪引物。一般情况下,在质量分数Q < 25的情况下,通过第一个碱基截断对序列进行质量过滤。然而,一些数据集没有通过这一严格的质量阈值(即,所得到的OTU表要么缺少许多原始样本,要么读取深度明显低于原始论文中报道的深度)。对于454个数据,我们将质量阈值放宽到20,而对于配对端的Illumina数据,我们删除了超过两个预期错误的读取。如果可能的话,所有的读数都被修剪到200 bp。在这种长度修剪丢弃了大部分序列的情况下,我们将阈值降低到150或101 bp。我们对每个数据集使用的具体处理参数可以在补充表中找到2.为了分配otu,我们使用USEARCH以100%相似度聚类otu56并使用RDP分类器为生成的otu分配分类17置信临界值为0.5。对于每个数据集,我们删除了读取次数少于100次的样本和读取次数少于10次的OTUs,以及在研究中存在于不到1%的样本中的OTUs。我们通过将每个OTU的值除以每个样本的总读数来计算每个OTU的相对丰度。然后,我们通过汇总OTUs各自的相对丰度来将其分解到属水平,丢弃任何在属水平上未注释的OTUs。所有的统计分析都是在该属水平的相对丰度数据上进行的。
统计分析
为了在每个数据集中执行案例和控制的监督分类,我们构建了具有五倍交叉验证的随机森林分类器。为了构建我们的训练和测试集,我们使用了python scikit-learn StratifiedKFold函数,并对数据进行了洗牌57.为了构建我们的分类器,我们使用了RandomForestClassifier函数和1000个估计器以及其他默认设置57.我们发现各种随机森林参数对auc没有显著影响(补充图。12而且13).我们根据交叉验证测试结果计算每个分类器的ROC曲线下的插值面积(AUC)。为了解释由于类别不平衡而产生的虚假高可分类性,我们还使用sklearn.metrics计算了每个分类器的Cohen’s kappa评分。cohen_kappa_score对测试集预测的影响(补充表4).kappa评分与auc (Pearsonρ= 0.9),这表明即使考虑到其底层数据分布,大多数分类器也表现良好。我们排除了young等人。18在所有分类器分析中,只有四个不同的对照患者。
我们使用scipy.stats.mstats.kruskalwallis函数对病例和对照中属的相对丰度进行了非参数Kruskal-Wallis检验的单变量分析58.我们使用statsmodels.sandbox.stats.multicomp.multipletests with method='fdr_bh'对每个数据集中的Benjamini-Hochberg错误发现率的多个假设检验进行了修正22.我们分别对每个数据集中的属级相对丰度进行了所有单变量分析,然后将这些结果在所有研究中进行了比较。
我们认为一个属始终与一种疾病相关(图。3,底部),是否与(问< 0.05)在至少两项关于该疾病的研究中,该疾病的方向相同。我们认为属是一种非特异性的微生物群落(图2)。3(上图),是否与(问< 0.05)在至少一个数据集的至少两种不同的疾病在同一方向。当我们定义这些非特异性属时,我们不包括使用非健康对照的数据集(Papa et al。19和Gevers等人。26)和Lozupone等人的数据集40在那里,微生物组信号反映的是行为,而不是疾病状态39.
为了构建我们的广义健康vs.疾病分类器(补充图9),我们首先连接了所有健康对照数据集的元数据和属级丰度数据(即除了Papa等人之外的所有数据集)。19和Gevers等人。26以非ibd患者作为对照,以及CDI young18只有四个不同的控制)。我们进行了保留一个数据集和保留一个疾病的交叉验证,并为每个交叉验证测试结果计算了AUC。
微生物群落分析
Alpha多样性基于未折叠的100% otu水平相对丰度计算,包括属水平上未标注的otu。我们用skbio.math.diversity.alpha计算了alpha多样性指标。Chao1、shannon和Simpson的实现。
我们计算了平均丰度和普遍性(图。3 c)的值,作为其在拥有16S数据的所有患者的每个数据集中的平均值的平均值,无论其疾病状态如何。为了计算每个属的丰度,我们首先计算每个属在每个数据集中的平均丰度。在此计算中,我们只计算了该属丰度非零的患者。然后,我们对所有数据集的平均丰度进行了计算。为了计算每个属的普遍性,我们计算了每个数据集中该属非零丰度患者的百分比。然后,我们对所有数据集上的这些普遍性取平均值。
代码的可用性
再现本文中所有分析的代码可在https://github.com/cduvallet/microbiomeHD.我们鼓励研究人员通过与我们联系,将他们现有和未来的病例对照研究纳入MicrobiomeHD数据库。
数据可用性
各研究的原始测序数据可在补充表中获取3..原始处理的OTU表可以在MicrobiomeHD数据库中访问,可在https://doi.org/10.5281/zenodo.84033316.补充文件,包括问每个数据集中所有属级比较的值,拥有三个以上数据集的疾病的疾病相关属,以及非特定属的列表可在https://github.com/cduvallet/microbiomeHD上获得。支持研究结果的所有其他相关数据均可在本文及其补充信息文件中获得,或可根据要求从通讯作者处获得。
参考文献
朱,L.等。非酒精性脂肪性肝炎(NASH)患者肠道微生物群的特征:内源性酒精与NASH之间的联系肝脏病学57, 601-609(2013)。
康,D. W.等。降低自闭症儿童肠道菌群普雷沃氏菌和其他发酵菌的发生率。《公共科学图书馆•综合》8, e68322(2013)。
特恩鲍,P. J.等人。与肥胖相关的肠道微生物组具有增加的能量收集能力。自然444, 1027-131(2006)。
萧,E. Y.等。微生物群调节与神经发育障碍相关的行为和生理异常。细胞155, 1451-1463(2013)。
Cryan, J. & Dinan, T.改变思维的微生物:肠道微生物群对大脑和行为的影响。神经科学。13, 701-712(2012)。
Poutahidis, T.等人。微生物共生体通过神经肽荷尔蒙催产素加速伤口愈合。《公共科学图书馆•综合》8, e78898(2013)。
儿子,J.等人。自闭症谱系障碍儿童与simons simplex收集的典型兄弟姐妹粪便微生物群的比较。《公共科学图书馆•综合》10, e0137725(2015)。
王,T.等。结直肠癌患者与健康志愿者肠道菌群的结构分离ISME J。6, 320-329(2011)。
肠道菌群与帕金森病及其临床表型相关。Mov。Disord。30., 350-358(2014)。
奥尔森,S. W.和Alm, E. J.生态失调不是答案。Microbiol Nat。1, 16228(2016)。
Zaneveld, J. R., McMinds, R. & Thurber, R. V.压力和稳定性:将安娜·卡列尼娜原理应用于动物微生物群落。Microbiol Nat。2, 17121(2017)。8月。
Walters, W. A., Xu, Z. & Knight, R.与肥胖和ibd相关的人类肠道微生物的元分析。2月。588, 4223-4233(2014)。
Sze, m.a. & Schloss, p.d.在噪音中寻找信号:重新审视肥胖和微生物组。mBio7, e01018-e01020(2016)。
斐努凯恩,M. M.,夏普顿,T. J.,劳伦特,T. J.和波拉德,K. S.微生物组中肥胖的分类特征?切入问题的核心。《公共科学图书馆•综合》9, e84689(2014)。
Ridaura, v.k.等。肥胖双胞胎肠道菌群不协调调节小鼠代谢。科学341, 1241214(2013)。
Duvallet, C., Gibbons, S., Gurry, T., Irizarry, R. & Alm, E.微生物omehd:健康和疾病中的人类肠道微生物组。Zenodo.https://doi.org/10.5281/zenodo.840333(2017)。
王琪,王志强,王志强,王志强,王志强。基于朴素贝叶斯分类器的rrna序列快速分类。达成。环绕。Microbiol。73, 5261-5267(2007)。
年轻人,我,等。粪便菌群移植治疗复发性艰难梭菌感染使用来自不相关捐赠者的冷冻接种物:一项随机、开放标签、对照试点研究中国。感染。说。58, 1515-1522(2014)。
爸爸,e等人。胃肠道微生物群的非侵入性测绘确定炎症性肠病儿童。《公共科学图书馆•综合》7, e39242(2012)。
Baxter, N. T., Ruffin, M. T., Rogers, M. A. & Schloss, P. D.基于微生物群的模型提高了粪便免疫化学检测结肠病变的敏感性。基因组医学。8, 37(2016)。
泽勒,G.等。粪便微生物群在结直肠癌早期检测中的潜力。摩尔。系统。医学杂志。10, 766-766(2014)。
Yoav B. & Yosef H.控制错误发现率:一种实用而强大的多重测试方法。J. R.统计社57, 289-300(1995)。
陈伟,刘芳,凌志,佟晓霞,向昌。结直肠癌患者肠管及黏膜相关菌群的研究。《公共科学图书馆•综合》7, e39743(2012)。
韩,Y. W.有核梭杆菌:共生转化的病原体。咕咕叫。当今。Microbiol。23, 141-147(2015)。
Flynn, K. J. Baxter, N. T. & Schloss, P. D.口腔细菌在结直肠癌中的代谢和群落协同作用。mSphere1, e00102-16(2016)。
Gevers, D.等人。新发克罗恩病中未治疗的微生物群。细胞宿主微生物15, 382-392(2014)。
摩根,X. C.等。炎症性肠病肠道微生物功能障碍及其治疗基因组医学杂志。13, r79(2012)。
一项对双胞胎的焦磷酸测序研究表明,胃肠道微生物谱随着炎症性肠病表型的变化而变化。胃肠病学139, 1844-1854 (2010)
杨晓明,杨晓明,杨晓明,丁基辅酶a:乙酸辅酶转移酶基因的克隆及其在人类结肠产生丁酸盐细菌中的应用。环绕。Microbiol。12, 304-314(2010)。
弗林特,H. J.,斯科特,K. P.,路易斯,P.和邓肯,S. H.肠道菌群在营养和健康中的作用。胃肠醇。乙醇。9, 577-589(2012)。
Miquel, S.等。普氏粪杆菌与人体肠道健康。咕咕叫。当今。Microbiol。16, 255-261(2013)。
Reeves, a . E., Koenigsknecht, M. J., Bergin, I. L. & Young, V. B.接种lachnospiraceae小鼠分离物后无菌小鼠胃肠道艰难梭菌的抑制。感染。Immun。80, 3786-3794(2012)。
舒伯特等人。微生物组数据区分艰难梭菌感染和非艰难梭菌感染。来自健康对照组的难辨性腹泻。mBio5, e01021-14-e01021-14(2014)。
文森特,C.等。肠道梭状芽胞杆菌的减少先于医院内艰难梭状芽胞杆菌感染的发展。微生物组1, 18(2013)。
辛格等人。与急性肠道感染和疾病恢复相关的肠道微生物群落微生物组3., 45(2015)。
黄,J. M. W., Souza, R. De, Kendall, C. W. C., Emam, A. & Jenkins, D. J. A.结肠健康:发酵和短链脂肪酸。j .中国。杂志。40, 235-243(2006)。
唐纳森,g.p.,李,S. M. &马兹马尼安,S. K.细菌菌群的肠道生物地理学。Nat. Rev.微生物。14, 20-32(2016)。
柯尔斯纳,J. B. &肖特,R. G.非特异性炎症性肠病的最新进展。新英格兰。j .地中海。306, 775-785(1982)。
诺格拉-朱利安,等人。肠道菌群与性取向和hiv感染有关。EBioMedicine5, 135-146(2016)。
Lozupone, c.a.等。肠道菌群的改变与hiv-1感染有关。细胞宿主微生物14, 329-339(2013)。
Dinh, D. M.等。慢性HIV感染的肠道菌群、微生物易位和全身炎症。j .感染。说。211, 19-27(2014)。
特恩鲍,P. J.等人。肥胖和瘦弱双胞胎的核心肠道微生物群。自然457, 480-484(2008)。
古德里奇,J. K.等。人类基因塑造了肠道微生物群。细胞159, 789-799(2014)。11月。
Zupancic, M. L.等。旧阶阿米什人肠道菌群分析及其与代谢综合征的关系。《公共科学图书馆•综合》7, e43052(2012)。
Ross, m.c.等人16s gut社区的卡梅伦县西班牙裔队列。微生物组3., 7(2015)。
莫斯卡,A.勒克莱尔,M. & Hugot, J. P.肠道微生物群多样性和人类疾病:我们应该在我们的生态系统中重新引入关键的捕食者吗?前面。Microbiol。7, 455(2016)。
Pasolli, E., Truong, D. T., Malik, F., Waldron, L. & Segata, N.大型宏基因组数据集的机器学习元分析:工具和生物学见解。公共科学图书馆第一版。医学杂志。12, e1004977(2016)。
拉斯科,d.a.等人。大肠埃希氏菌的泛基因组结构:大肠埃希氏菌共生和致病分离株的比较基因组分析。j . Bacteriol。190, 6881-6893(2008)。
王,Z.-K。&杨,y - s。上消化道菌群与消化系统疾病。世界J.胃肠醇。19, 1541-1550(2013)。
胃肠道的微生物生态学。为基础。启Microbiol。31, 107-133(1977)。
通过基于16s rrna的焦磷酸测序对与MHE相关的肠道菌群进行大规模调查。点。j .杂志。108, 1601-1611(2013)。
Scher, J. U.等。肠道copri普氏菌扩张与增强关节炎易感性相关。eLife2, e01202(2013)。
法洛尼,G.等。肠道微生物组变异的人群水平分析。科学352, 560-564(2016)。
David, l.a.等人。宿主的生活方式在日常时间尺度上影响人体微生物群。基因组医学杂志。15, r89(2014)。
帕斯卡,V.等人。克罗恩病的微生物特征肠道66, 813-822(2017)。2月。
搜索和聚类的速度比爆炸快几个数量级。生物信息学26, 2460-2461(2010)。
Pedregosa, F.等人。Scikit-learn: Python中的机器学习。机器学习。Res。12, 2825-2830(2011)。
琼斯,E.等。SciPy: Python的开源科学工具。http://www.scipy.org/(2001)。
Alkanani, A. K.等。肠道菌群的改变与1型糖尿病的易感性相关。糖尿病64, 3510-3520(2015)。
Meja-León, M. E., Petrosino, J. F., Ajami, N. J., Domnguez-Bello, M. G. & de la Barca, A. M. C.墨西哥1型糖尿病儿童粪便菌群失衡。Sci.代表.4, 3814(2014)。
王文伟等。非酒精性脂肪性肝炎患者粪便微生物群的分子特征——一项纵向研究《公共科学图书馆•综合》8, e62885(2013)。
确认
我们感谢公开提供数据的作者,以及在个人沟通后提供数据的作者。该项目部分得到了麻省理工学院微生物信息学和治疗学中心的支持,C.D.通过国防科学与工程研究生奖学金(NDSEG)计划感谢国防部的支持。我们感谢Shijie Zhao和Xiaoqian Yu对文献综述的早期贡献,以及Nathaniel Chu和Alm和Irizarry小组的其他成员的有益讨论。
作者信息
作者及隶属关系
贡献
c.d., t.g.和E.A.构想了这项研究。c.d., s.g.和T.G.识别并下载了数据集。C.D.和T.G.编写了处理数据的代码。cd处理了数据并进行了所有分析。c.d., s.g., r.i.和E.A.解释了结果并准备了手稿。
相应的作者
道德声明
相互竞争的利益
E.A.是OpenBiome的董事会成员,OpenBiome是一家非盈利粪便银行。E.A.是Finch Therapeutics的联合创始人之一,该公司旨在开发基于微生物群的治疗方法。其余作者声明没有相互竞争的经济利益。
额外的信息
出版商的注意:施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
杜瓦莱,C.,吉本斯,s.m.,高锐,T.。et al。肠道微生物组研究的元分析确定了疾病特异性和共同反应。Nat Commun8, 1784(2017)。https://doi.org/10.1038/s41467-017-01973-8
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41467-017-01973-8
进一步的阅读
用SameStr进行宏基因组菌株检测:鉴定可通过粪便移植转移的持久核心肠道菌群
微生物组(2022)
荟萃分析定义了各种疾病中主要的共享微生物反应和特定的炎症性肠病信号
基因组生物学(2022)
槐脂通过改善早期断奶仔猪的肠道微环境来预防早期断奶综合征
BMC兽医研究(2022)
人类微生物群中的超级分类单元被确定与结直肠癌有关
BMC生物信息学(2022)
基于互信息的微生物组网络分析筛选asv /OTUs
BMC生物信息学(2022)