文摘
英国生物库的项目是一个前瞻性群组研究遗传和表型数据收集约500000人来自英国,在招聘年龄在40至69。开放资源的独特之处在于它的规模和范围。丰富的各种表型和与健康有关的信息可以在每个参与者,包括生物测量,生活方式指标,在血液和尿液生物标志物,身体和大脑的成像。后续提供的信息是连接健康和医疗记录。全基因组基因型数据已经收集了所有的参与者,提供许多机会的发现新的基因协会和复杂性状的遗传基础。这里我们描述基因数据的集中分析,包括基因型质量、属性的人口结构和基因数据的关联性,高效逐步和基因型参数,增加测试的变异的数量约为9600万。古典晚上11人类白细胞抗原基因等位变异是估算,导致经济复苏的信号与已知的人类白细胞抗原等位基因之间的关联和许多疾病。
主要
理解基因的作用在表型和疾病的变化,及其潜在的与其他因素的相互作用,对更好地理解人类生物学是至关重要的。希望这将导致更多的成功的药物开发1,可能更有效和个性化的治疗。因此,英国生物库资源的重要组成部分已经全基因组遗传数据的收集在每个参与者使用专型数组2。一个临时版本的基因型数据约150000英国生物库参与者在2015年5月3已经帮助无数的研究4,5,6。
在这篇文章中,我们总结了现有的和表型的计划内容资源和描述基因数据集完整的500000名参与者。促进它的广泛使用,我们应用一系列的质量控制程序,进行了一系列的分析,揭示基因数据(属性的人口结构和relatedness-that下游分析是很重要的。此外,我们估计单和估算基因型数据集增加测试的变异的数量超过100倍,约9600万个变异。我们也估算古典等位变异人类白细胞抗原(HLA)基因在11,和复制信号已知的HLA等位基因之间的关联和许多常见疾病。我们描述的工具,允许有效的全基因组关联研究(GWAS)的多个特征和快速phenome-wide协会研究,这与一个新的压缩文件格式,用于分发数据集。基因分型的进一步检查和估算数据集,我们执行一个测试用例全基因组关联研究人类特征扫描,站的高度。
英国生物库
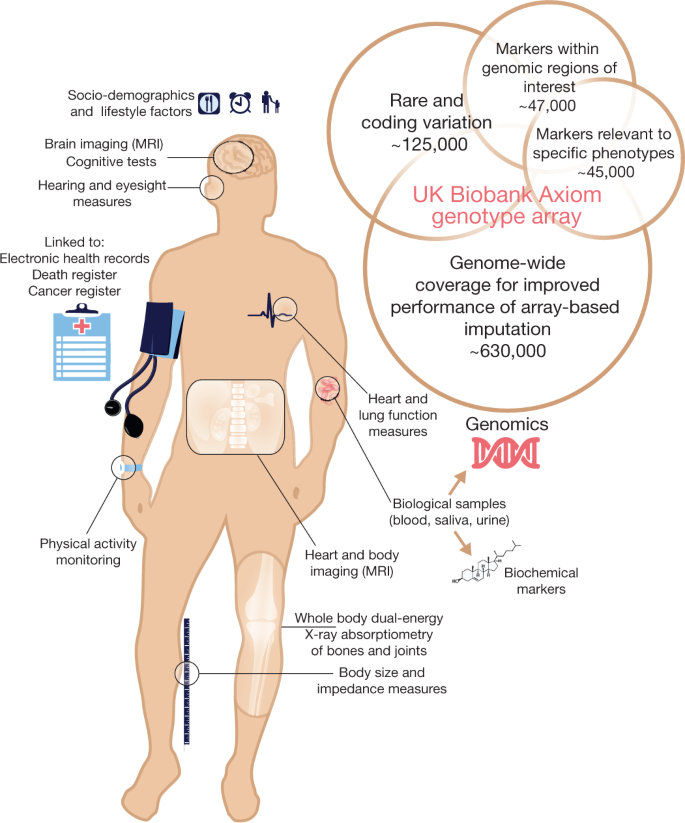
各种各样的表型信息以及生物样品已经收集了大约500000英国生物库的每个参与者(图。1)。在招聘中,参与者提供电子签名同意,socio-demographic回答问题,生活方式和健康相关因素,完成了一系列的物理措施(见扩展数据表1)。他们还提供血液,尿液和唾液样本,存储在这样一种方式,允许执行许多不同类型的分析(例如,基因,蛋白质组学和metabonomic分析)7。一旦招聘全面展开,进一步增强了评估访问,包括眼睛的一系列措施,一个心电图仪测试,动脉硬化和听力测试。
基线信息已经并将继续是,在几个方面扩展。例如,重复评估计划在队列的子集进行每隔几年,使测量的校准,调整回归稀释,纵向变化的估计。身体活动的客观措施也被收集(使用三轴加速度计)在2013 - 2014年的100000名参与者8重复措施被收集在一段(季节性的基础上)从2500年的参与者。多模式成像评估正在进行中,包括大脑的磁共振成像(MRI)9、心脏10和身体,颈动脉超声检查11和一个全身骨骼和关节的双能x线吸收仪12。数据收集从2014年开始,预计7 - 8年的100000名参与者实现成像专用成像评估中心在英国,和重复成像措施计划参与者的一个子集。
所有参与者提供同意后续通过链接到他们的健康记录。截至2018年5月,有超过14000人死亡,79000名参与者与癌症诊断,并与至少一个住院400000人。现在正在进行相当大的努力,把数据从一个范围的其他国家的数据集,包括初级保健,筛查项目,针对疾病的注册,以及直接要求参与者对健康相关结果通过在线问卷调查(见扩展数据表1)。也在努力开发可扩展的方法,可以详细描述不同健康状况通过交叉引用多个编码的临床信息的来源13。
测量范围广泛的生化标记主要感兴趣的研究社区也一直在进行,包括那些已经知道对疾病(例如,脂质在血管疾病和性激素对癌症),(例如,HbA的诊断价值1 c糖尿病和关节炎类风湿因子),或表型特征的能力,否则不好评估(例如,生物标志物肾和肝功能)。
英国生物库是开放获取资源,鼓励研究人员来自世界各地,包括学术,慈善机构,公共和商业部门,访问数据的任何健康相关研究公共利益。
全基因组基因分型
英国生物库的遗传数据包含488377名参与者的基因型。这是化验使用两个非常相似的基因序列。49950人参与了英国生物库的子集肺外显子组变异评价(英国BiLEVE)研究基因分型在807411标记使用由Affymetrix英国BiLEVE公理数组应用生物系统公司(现在的热费希尔科学的一部分),这是其他地方描述6。438427年,参与者使用密切相关的基因分型应用生物系统公司英国生物库Axiom数组(825927标记),股价95%的标记内容与英国BiLEVE Axiom数组。英国生物库的标记内容选择公理数组来捕获全基因组遗传变异(单核苷酸多态性(snp)和短的插入和删除(indels)),并总结了无花果。1。许多标记包括因为已知的联想,或可能的角色,疾病。数组还包括编码变异在一系列微小等位基因频率(加),包括罕见的标记(< 1%加);和标记,提供良好的全基因组范围的归责欧洲人共同(> 5%)和低频率(1 - 5%)加范围。数组的进一步的细节设计在英国生物库公理数组内容摘要2。
DNA从存储中提取血液样本被收集到的参与者在他们访问英国生物库评估中心。基因分型是由Affymetrix研究服务实验室在106年连续批约4700(见样品方法补充表12)。Affymetrix应用自定义基因型筛选优化管道和质量呼吁biobank-scale pcr实验和小说基因序列,含有标记没有以前输入(见使用Affymetrix技术方法)。这导致一组基因型呼吁489212个样本812428独特的标记(biallelic SNPs和indels)从两个数组,我们进一步进行质量控制和分析(扩展数据表2)。
我们的质量控制管道是专门适应大规模数据集的多元民族的参与者,在很多批次基因分型,使用两个不同的数组,将被许多研究人员用来解决各种研究问题。参与者报告了他们的种族背景通过选择一组固定的类别14。尽管大多数(94%)的人对广泛级别组内报告他们的种族背景“白色”,仍有大约22000个人自我报告的原始欧洲以外的种族背景(扩展的数据表3)。我们使用方法基于主成分分析(PCA)占人口结构标记和纸浆包质量控制(见方法)。
识别标记质量太差,我们使用统计测试主要是检查一致性实验设计的因素,比如数组或批处理(见方法;扩展的数据表4)。由于这些测试,我们将错过0.97%的基因型由Affymetrix的电话。我们确定了质量差的样品使用的指标缺失率和杂合性调整人口结构(扩展数据图。1),在一个或两个极端值的度量指标的样品质量差,例如,DNA污染15。我们确定了968个这样的样品(0.2%),为研究人员提供这个清单。
每个个人自述性之间的不匹配,性推断从标记的相对强度X和Y染色体16,可以用来检测可能的样本处理不当或其他类型的笔误。在这种规模的数据集,一些这样的不匹配将会由于变性或双性人,或罕见的遗传变异的实例,如性染色体非整倍性17。使用信息测量强度的X和Y染色体(见方法),我们确定了一组652例(0.134%)患者的性染色体核型形成推定地不同于XY或XX(无花果。二维补充表2)。
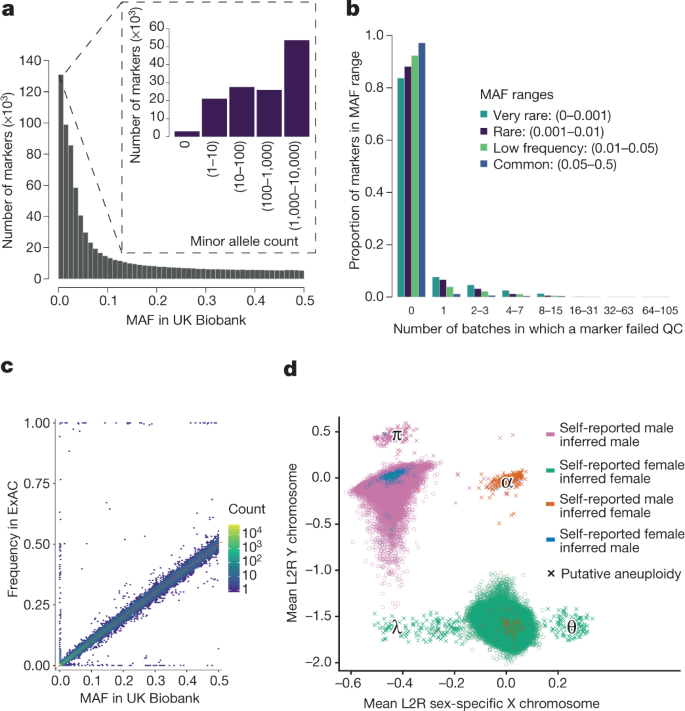
英国生物库的所有情节都显示属性基因型数据在应用质量控制。一个加器分布基于所有样本(805426标记)。插图显示只罕见的标记(加< 0.01)。b分布的数量批量质量控制(QC)测试失败(见一个标志方法)。每一个加范围,我们显示标记的部分失败指定批次的数量。c加的,比较英国生物库ExAC相同的等位基因的频率,在欧洲血统的参与者在每个研究(补充信息)。这个分析使用91298重叠的标记。显示每个六角形本彩色标记的数量在下降,本(日志10规模)。虚线红线所示x=y。截然不同的标记等位基因频率出现在顶部,底部和左手的情节包含大约300标记。这是0.3%的比较(见标记补充信息讨论)。d,意思是日志2比率(左2R) X和Y染色体上为每个示例,说明可能的性染色体非整倍性(见方法)。有652个样本可能的性染色体非整倍性(十字架)。集群的位置的不同假定的分析是由希腊符号表示:λ= X0(或马赛克XX / X0),θ= XXX,α= XXY,和π= XYY。项个人在这些地区补充表中给出2。自述性的颜色表示不同的组合,和性推断,Affymetrix(从基因数据)。几乎所有样品(99.9%)、自我报告和推断性是相同的,但对于小数量的样品(378)他们不匹配补充信息讨论)。
我们的质量控制管道的应用导致了488377年公布的数据集样本和805426标记与图中所示的属性数组。2 a - c。一组588对实验重复显示非常高的基因型一致,意思是99.87%,最低99.39%的基因型完全相同的(补充图。13)。我们比较等位基因频率与欧洲血统的英国生物库参与者从一个独立源估计,外显子组聚合财团(ExAC)数据库18在91298年的一组重叠的标记。我们不希望等位基因频率在两个研究完全匹配由于细微的差别在每个个体的遗传背景研究,以及两种技术的敏感性和特异性的差异(外显子组测序和基因序列)。少量的标记(大约300年)有非常不同的等位基因频率(见补充信息部分2.4)。这可能是由于在英国生物库数组或非工作probesets注释错误在英国生物库数组或ExAC, or映射序列数据中的错误的地区更复杂的变化。尽管如此,总体的等位基因频率是令人鼓舞的是相似的(r2= 0.93)(图2摄氏度;补充图。4)。
110000多罕见的标记(在英国生物库加< 0.01)包括在两个数组用于英国生物库队列2。变异发生在非常低的频率存在一个特定的基因型调用使用数组技术的挑战。可以挑战区分样本,真正有轻微的等位基因,从一个的强度在尾分布的主要纯合子集群(扩展数据图。2)。难得的一个更大的部分标记失败比低频率和质量控制测试常见的标记,但是84%的人仍然通过所有批次(无花果。2 b)。我们建议研究人员视觉检查集群的阴谋,类似于补充图。2等标记的使用效用,召唤师(https://github.com/wtsi-medical-genomics/evoker),尤其是罕见的标记。
祖先的多样性和神秘的亲缘
基因型数据提供了一个独特的机会来研究不同祖先起源(扩展数据表3英国生物库的参与者。占祖先背景是必不可少的流行病学研究和遗传分析,如GWAS19。我们使用主成分分析来衡量人口结构在英国生物库队列(见方法)。图3显示了前四个主成分结果绘制在连续双(参见扩展数据图。3和补充无花果。6,7)。正如所料,个人有类似的主成分得分有类似的自我报告的种族背景。例如,前两个主成分分离出个人与撒哈拉以南非洲血统,欧洲血统和东亚血统。自我报告作为混合种族的人往往会落在其组成团体之间的连续体。进一步的主成分捕获种群结构在陆运地理尺度(扩展数据图。3)。我们的主成分分析显示人口结构中最常见的种族背景类别(88.26%)、“英国”在更高的级别组“白”(补充图。8)。我们使用自我报告的种族背景和主成分分析结果为研究人员提供一个列表,409728人(84%)有非常相似的遗传背景相对于整个队列(见方法)。
一个参与者,每个点代表一个英国生物库(n= 488377个样本)和被放置在每个主成分得分(PC)的前4个主成分。颜色和形状表示每个个体的自我报告的种族背景。看到扩展数据表3在每个类别的比例。b亲戚的数量分布,参与者在英国生物库队列。每个酒吧的高度显示计数的参与者(日志10规模)表示数量的亲戚。颜色显示的比例每个亲缘类在一个酒吧。c,家庭组织在英国生物库队列的例子。点代表参与者,颜色的点之间显示出他们的推断关系(例如,蓝线加入完整的兄弟姐妹)。家庭网络的整数显示总数的队列(如果多于一个)相同的配置,忽略三度对。
密切的关系(例如,兄弟姐妹)在英国生物库的参与者并没有记录在其他表型信息的集合。此信息可以重要的流行病学分析20.,以及在GWAS21。我们使用了基因数据来识别相关的个人通过为所有成对的样品估计亲属关系系数,并报告系数对亲戚我们推断逼供的亲戚或接近(见方法)。共有147731名英国生物库参与者(30.3%)推断是相关(第三个学位或接近)至少一人队列,并形成107162相关双(扩展数据表5)。这是一个非常巨大的数字,它不是仅仅由过度的逼供的亲戚。例如,兄弟姐妹的数量对(22666)大约是两倍理论上预期在一个随机样本(大小)的合格的英国人,后考虑到典型的家庭规模(补充表4)。数量大于预期的相关双取样偏差可以解释,因为,例如,一个人更有可能同意参与因为家庭成员也参与其中。似乎是可信的,而且,如果集群相关个人地理位置而不是随机坐落在英国,英国生物库的招聘策略评估中心22自然会倾向于oversample相关的个人。
对个人在英国生物库相关的群体形式网络相关的个人。在大多数情况下,这些都是大小两个,但是也有很多组大小队列(图三个或更大。3 b),即使限制二级亲属或近亲属对。通过考虑关系类型和年龄和性别的个体在每个家庭群体中,我们确定了1066套三人小组(两个父母和一个子女),包括1029独特的父母和37集四重奏(两个父母和两个孩子)。
5或更多的人有172个家庭组织二级亲属或接近(无花果。3 c)。其中一个组有11人都是彼此的二级亲属(父异母或异父同母兄弟姐妹、祖父母/孙子或慈祥的)。因为所有的55对二级亲属,至少10人必须寻找相同的共享父(见补充材料)。我们确认共享家长必须他们的父亲因为他们不携带相同的线粒体的等位基因,和男性有相同的Y染色体等位基因(数据没有显示)。
单体型估计和基因型污名
我们估计单整个队列(预研阶段),其次是单倍体归责23。预研阶段的步骤中,我们仅仅使用标记出现在英国BiLEVE和英国生物库Axiom数组。我们删除标记,没有质量控制多个批处理,整体缺失率大于5%,小于0.0001的加。我们移除样品确认为异常值的杂合性缺失率。这些过滤器导致了487442年与670739年常染色体标记数据集样本。使用SHAPEIT3常染色体上逐步进行24(见方法和https://jmarchini.org/software/)。第三阶段1000基因组数据集25被用作参考面板,主要帮助逐步样本与非欧洲血统。在一个单独的实验,利用阶段推断从mother-father-child三人小组,我们估计平均逐步切换错误率为0.229%(见方法)。
我们使用了单体型参考联盟(HRC)26数据作为主要归责参考面板,因为它是最大的广泛可用的设置(64976)欧洲单39235157个snp。补充图。15显示了一个单独的结果归责实验表明HRC比UK10K面板面板产生更好的归责性能,特别是在较低的等位基因频率,英国生物库Axiom数组和其他商用数组执行媲美。
我们也估算使用合并的英国生物库UK10K和1000基因组三期参考面板2787696888年,bi-allelic标记。我们结合这个估算数据从HRC面板,使用HRC归罪SNP在场时在两个面板。进行归责与IMPUTE4计划(https://jmarchini.org/software/),这是一个重新编码版本的单倍体归罪在IMPUTE2功能实现23(见方法)。归责过程是一个数据集的结果有93095623个常染色体snp,短indels和大型结构变异在487442人。我们估算一个额外的3963705 X染色体上的标记(方法)。SNP数据库(dbSNP)参考SNP (rs) ID分配给尽可能多的标记使用参考SNP ID列表可以从UCSC基因组注释GRCh37组装的人类基因组数据库(http://hgdownload.cse.ucsc.edu/goldenpath/hg19/database/)。
扩展的数据图。4显示所有标记信息的分布分数估算数据集。一个信息的α在一个样本米个人在估算标志表明的数据量大约相当于一组完全观察到的基因型数据的样本大小αM。图说明大多数标记0.1%以上分数高频率信息。先前的GWAS倾向于使用一个过滤器信息约0.3大致对应于一个有效的样本容量约150000。因此,它可能会减少分数阈值的信息,仍然获得良好的检测协会。
我们开发了一个新的BGEN文件格式(v1.2;http://www.well.ox.ac.uk/ gav / bgen_format / bgen_format.html图书馆)和软件(BGEN;https://bitbucket.org/gavinband/bgen)提供改进的数据压缩,分阶段单体型数据存储和随机存取的数据通过使用一个单独的索引文件。使用这种新的格式,完整的估算文件需要2.1 Tb的文件空间。一个新项目(BGENIE;https://jmarchini.org/software)建成使用BGEN库进行快速多性状GWAS phenome-wide协会研究28(见补充信息)。
归责的经典HLA等位基因
主要组织相容性复合体(MHC) 6号染色体上是最人类基因组多态区域,包含最多的常见疾病遗传关联29日。我们推算HLA类型在两个区域(也称为四位数)分辨率11经典HLA基因(HLA- - - - - -一个,HLA- - - - - -B,HLA- - - - - -C,HLA- - - - - -DRB1,HLA- - - - - -DRB3,HLA- - - - - -DRB4,HLA- - - - - -DRB5,HLA-DQA1,HLA- - - - - -DQB1,HLA- - - - - -DPA1和HLA- - - - - -DPB1)利用HLA *小鬼:02 multi-population参考面板(补充表算法5和6)30.和验证使用交叉验证实验的准确性。在一个典型的使用,情况估计精度优于96%所有位点(见方法和补充表7,8)。
为了演示HLA归责的效用,我们协会执行测试已知的疾病HLA关联。我们分析了409724人(见英国白人血统的子集方法)和集中在11自我免疫介导性疾病与已知的HLA关联。对于每个疾病在我们的分析,我们确定了HLA等位基因关联的最有力的证据。在所有情况下这是一致的(见以前的报告方法和补充表9)。我们进一步复制独立HLA关联在一个单一的疾病研究多发性硬化(MS)国际多发性硬化遗传易感性的财团(IMSGC)31日。我们观察到的证据协会和效果预估HLA等位基因整合的方向和相对大小与IMSGC研究中发现,尽管在11个14例这是接近1,符合回归稀释偏见带来的低利率的表型(表错误1)。
GWAS的站的高度
直接评估的潜在基因分型和估算数据,我们进行了几站无关的高度使用343321,欧洲血统参与者(见英国生物库方法)。我们比较我们的结果的重叠荟萃分析欧洲血统的253288人进行的人体测量特征的遗传调查(巨人)财团32。
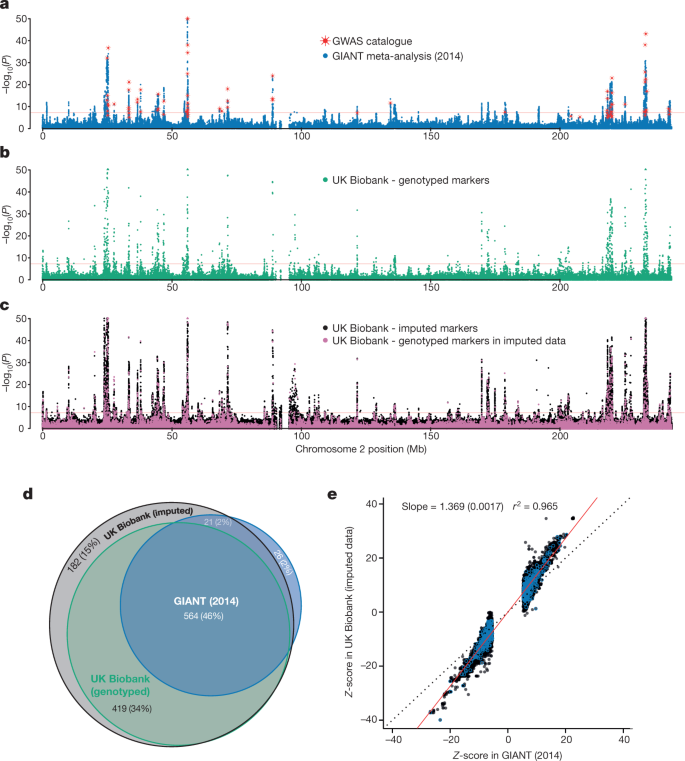
令人放心的是,协会信号的模式在英国生物库和巨大的结果都很相似(图4得了),Z分数相关的标记高度相关(r2= 0.965;无花果。4 e)。获得执政的英国生物库队列是明确的,与许多位点达到全基因组意义(P< 5×10−8在英国生物库而不是巨大的研究(图。4 d补充图。16);和Z成绩相关联的标记系统更高的英国生物库(回归斜率= 1.369,无花果。4 e)。地区协会的英国生物库显示模式信号预期的连锁不平衡结构和重组率在该地区(见扩展数据图。5一个例子)。
结果(P人类高度值)之间的关联测试并使用三个不同的基因型为2号染色体组数据。在一个- - - - - -c,P值显示在−日志10规模,限制在50视觉清晰度和未调整的多重比较。标记与−日志10(P)> 50 50的绘制y轴和显示为三角形,而不是点。水平红线表示P= 5×10−8。一个公布的结果,由巨大的荟萃分析32(n= 253288),与NCBI GWAS目录用红色标记重叠(策划报告P值)。b协会统计,(从线性混合模型方法英国生物库标记基因型数据)(n= 343321)。c协会统计,(从线性混合模型方法英国生物库标记)的估算数据(n= 343321)。点颜色粉色标记的基因表明用于预研阶段和非难。这意味着大多数的数据在每一个这些标记的来自于基因型分析。黑色点(绝大多数,~ 800万)表明充分估算标记。d,维恩图的计数结果的数量1 mb windows与至少一个轨迹P< 5×10−8巨头,英国生物库基因分型和英国生物库估算数据集(见方法)。括号里的百分比是工会的windows的比例在所有三个数据源(1215)。英国生物库中包含的只有三个窗口基因分型数据而不是估算数据。e的,比较Z再保险公司e英国生物库的年代(y轴)和巨人(x轴)。Z分数计算效应值除以标准误差,但只是为标记P< 5×10−8在巨大的一组575个相关地区,我们还用于可靠组分析(见方法)。用最小的标记P值(巨人)在每个地区突出显示蓝色的圆圈。黑色虚线所示x=y,红色实线显示了线性回归直线对这些数据估计。回归系数的标准错误显示在括号中。皮尔森的相关性是用于计算r2价值。
英国生物库的有效性评估基因精细定位数据在相关的位点,我们计算95%可信集33575地区(包含至少一个全基因组的重要标志P< 5×10−8)在巨人和英国生物库(参见估算数据方法)。标记的数量我们分析英国生物库(768502)是大大超过在巨大(106263),这影响分辨率相关联的任何给定的区域(扩展数据图。6)。在考虑所有标记时,可靠的设置在英国生物库的大小通常是更大的(中等大小= 8)比巨人(中等大小= 6),但在可信的snp的比例每个区域(扩展数据图。6 b)通常是较小的英国生物库(中位数比例= 0.010)比巨人(中位数比例= 0.047)。通过限制标记的两项研究(105421)我们发现95%可信集的规模一般较小的英国生物库(中等大小= 4)比巨人(中等大小= 6)。95%可信集只包含1的数量标志是123年在英国生物库和76年的巨人。
结论
基因数据的临时释放大约150000名参与者在英国生物库已经促进了许多论文探索人类遗传变异和疾病之间的联系,以及他们与广泛的环境和生活方式的因素。英国生物库的进一步持续增长的表型信息,因为研究人员返回结果的分析英国生物库共享。在线资源正在开发共享使用英国生物库数据分析的结果,包括GWAS的释放结果成千上万的表型(http://www.nealelab.is/uk-biobank)和牛津大学脑成像遗传学服务器28(http://big.stats.ox.ac.uk/)。我们预计,英国生物库的完整基因数据的可用性将导致进一步改变生产力研究周期。英国生物库是一个功能强大的极有价值的例子,可以实现从规模庞大的人口研究,结合遗传学和广泛而深刻的表现型和连锁健康记录加上一个强大的数据共享政策。很可能预示着一个新时代,这些和相关资源驱动,提高对人类生物学和疾病的理解。
方法
样本数据收集、检索、DNA提取和基因型
伦理批准英国生物库研究从西北研究中心获得伦理委员会(11 /西北/ 0382)。收集血液样本从参与者在他们访问英国生物库评估中心和样本存储在斯托克波特的英国生物库设施,英国7。在一段时间内的18个月样本检索,DNA提取,和96 - 94孔板×50-μl整除被运到Affymetrix pcr实验室研究服务。是特别注意在英国生物库的自动样本检索过程,确保实验单位如盘子或提取的时机没有关联与基线系统的表型,如年龄、性别、种族背景,或者样本集合的时间和地点。英国生物库的详细示例检索和DNA提取过程是前面描述的34。
收据的DNA样本,Affymetrix GeneTitan多渠道(MC)的样品处理仪器在96 - 94孔板包含英国生物库样本和两个控制样本1000人基因工程25。基因型被称为从数组中强度数据,单位叫做“批次”由多个板块。在整个群体,有106批4700英国生物库样本(补充信息补充表12)。早些时候临时数据发布后,Affymetrix开发一个定制的基因型管道优化呼吁biobank-scale pcr实验,利用multiple-batch的设计35。这个管道是适用于所有样本,包括150000个样本,临时数据发布的一部分。因此,一些基因型呼吁这些样本可能在不同的临时数据发布和最后的数据发布(见下文)。
例行质量检查进行了样本检索过程中DNA提取36,基因型打电话37。任何样品没有通过这些检查是排除产生的基因型。专门设计的数组包含一个标记的数量没有以前使用Affymetrix基因型阵列技术类型。也因此,Affymetrix应用一系列的检查,以确定是否为给定的标记基因型鉴定是成功的,在一个批处理,或在所有样本。这些新尝试化验没有成功,Affymetrix排除的标记数据交付(看到了吗补充信息详情)。
概略介绍质量控制
我们确定质量较差标记使用统计测试主要是检查设计的一致性的基因型实验因素。专门为批处理的影响,我们测试板的影响,偏离哈迪温伯格平衡,性影响,阵列的影响,和不整合控制复制。看到补充信息为每个测试的细节,和补充图。3对影响标记的例子。标记失败了至少一个测试在一个给定的批处理,我们设置了基因型电话,批失踪。我们还提供一个标志数据的发布表明是否要求标记被设置为给定批失踪。如果有证据表明一个标志并不是可靠的所有批次,我们排除了数据的标记。减弱人口结构的影响,我们都应用使用的一个子集概略介绍质量控制测试463844估计欧洲血统的人。之前我们确认这些个体的基因型数据进行任何质量控制时,将所有的英国生物库样本的两个主要的四个主成分1000基因组数量(CEU、套、慢性乙肝和被25。然后我们选择样本主成分得分下降的邻居CEU集群(补充信息)。
纸浆包质量控制
我们确定了质量差的样品使用的指标缺失率和杂合性计算使用一组605876个高质量的常染色体标记类型数组(见补充信息为标准)。极端值的一个或两个这些度量指标的样品质量差,例如,DNA污染15。取样的杂合性的一部分non-missing标记被称为heterozygous-can也是敏感的自然现象,包括人口结构、最近的掺合料和父母的血缘关系。我们采取了额外的措施来避免分类质量好的样品,因为这些影响。例如,我们调整人口结构的杂合性拟合的线性回归模型前六个主成分在PCA预测(扩展数据图。1)。使用这种调整我们确定了968个样本与异常高的杂合性或缺失率(> 5%补充信息)。这些样品的列表是作为数据发布的一部分提供的。
我们还进行了质量控制的性染色体使用一组特定于15766高质量标记X和Y染色体。Affymetrix推断性别的每个单独的基于标记的相对强度X和Y染色体16。性也是报告的参与者,这些来源之间的不匹配可以用来检测样本处理不当或其他类型的笔误。然而,在这种规模的数据集,一些这样的不匹配将会由于变性人,或真正的实例(但罕见)的遗传变异,如性染色体非整倍性17。Affymetrix基因型呼吁X和Y染色体只允许单倍体和二倍体基因型电话,根据推断性16。因此,病例全部或镶嵌性染色体非整倍性可能会导致基因型呼吁所有妥协,或部分,性染色体(但不影响常染色体)。例如,患者核型XXY可能质量较次的基因型呼吁pseudo-autosomal地区(PAR)的X染色体,因为他们实际上是三倍体在这个地区。使用信息测量强度的X和Y染色体,我们确定了一组652例(0.134%)患者性染色体核型形成推定地不同于XY或XX(无花果。二维补充表2)。样品的列表提供了数据发布的一部分。研究人员想要识别性不匹配应该比较自述性和推断性数据字段。
我们没有删除样本数据由于任何上述分析,而是提供信息数据发布的一部分。然而,我们排除了少量的样本(835),我们确认为样本复制(而不是同卵双胞胎,看到补充信息),或者可能是参与在实验室样品处理不当(~ 10),以及参与者要求退出这个项目之前发布的数据。
比较临时和最终发布数据
后续的临时释放基因型(2015年5月)大约150000英国生物库参与者对基因型进行了调用算法的改进35和质量控制程序。因此我们能看到一些改变样本的基因型电话和丢失的数据文件包含在临时数据发布和最后的数据发布。不调和non-missing标记很低(平均6.7×10−5;补充图。1);为每个样本有24500个基因型和调用(平均)失踪的临时数据,但在这个版本non-missing调用。这是小得多的相反方向,500个电话,平均而言,失踪在这个版本而不是丢失的临时数据,所以平均净增24000个基因型电话/样品。
主成分分析
我们使用一种算法(fastPCA计算主成分38)执行与成千上万的样本数据集的近似只有顶部n主成分解释最变化,n事先指定。我们计算前40名校长组件使用一组407219无关的,高质量的样品和147604高质量标记修剪减少连锁不平衡39。然后我们计算相应的主要component-loadings和预计所有样本主成分,形成一组队列中的所有样本主成分得分(补充信息)。
英国白人祖先子集
研究人员可能希望只分析一组的相对同质的祖先减少混淆的风险由于祖先背景的差异。尽管英国生物库队列包括大量的参与者从一个广泛的种族背景,这种分析是可行的在样本大小不影响太多,因为大多数参与者的英国生物库队列报告他们的种族背景“英国”,在更高的级别组“白”(88.26%)。我们的主成分分析显示人口结构甚至在这个类别(补充图。8),所以我们使用自我报告的种族背景和遗传信息来识别的一个子集,409728人(84%)自我报告为“英国”,谁有非常相似的祖先背景基于主成分分析的结果(补充信息)。精细的人口结构已知存在于英国但方法检测这样的细微结构40可用的分析不可行的在英国生物库的规模应用。英国白人祖先子集可能因此还含有细微结构出席国家等级。
亲属关系系数估计
我们使用了一个估计量在软件实现的,王41,因为它是健壮的人口结构(也就是说,不依赖于精确的估计人口等位基因频率)是实现一个算法高效足以考虑所有对(~ 1.2×1011)在一个可行的时间。王的作者所指出的,最近我们发现外加剂(例如,“混合”祖先背景)倾向于夸大亲属关系的估计系数,与标记之间的估计假设哈迪温伯格平衡相同的底层等位基因频率在一个独立的个体。我们使用的一个子集来缓解这种效应只有标记只是祖先的弱信息背景(补充信息补充图。12)。我们也排除个人的一小部分(977)的亲属关系估计,作为他们属性(例如,缺失率高),将导致不可靠的亲属关系估计(补充信息)。我们使用亲属关系类呼吁每一对相关系数和分数的标记他们分享没有等位基因(IBS0)。看到部分补充信息3.7获取详细信息。
以确保我们没有高估相关的数量对,我们推断相关对(在数据的一个子集)使用不同的推理方法实现的叮铃声(“基因组”命令;https://www.cog-genomics.org/plink2),确认100%的双胞胎,parent-offspring和兄弟姐妹对,对总体的99.9% (补充信息)。
单体型估计
单体型估计(逐步)进行了使用SHAPEIT3 15000年块标记,重叠250块之间的标记。每个块4核/工作和使用年代= 200复制状态。块被结扎使用修改后的版本的hapfuse计划(https://bitbucket.org/wkretzsch/hapfuse/src)。
我们评估的准确性逐步在一个单独的实验利用mother-father-child三人小组在英国生物库队列被识别。这个家庭信息可以用来推断的大量标记阶段三的父母。这些被用作一组事实family-inferred单体型,是常见的分阶段文学。每个三的父母从数据集中删除,然后单估计在20号染色体SHAPEIT3在单个运行。16175年的这个数据集由常染色体标记。然后推断单体型比真相设置使用开关误差指标。使用一组696个三人小组自我种族背景“英国”(在更高的级别组“白色”),没有其他双胞胎或第一或二级亲属在英国生物库的数据集,我们估计平均切换错误率为0.229%。我们也使用397个三人小组的一个子集,也没有逼供的亲戚和获得的平均切换错误率为0.234%。这些错误率是类似于那些由其他在这个规模逐步可以处理数据的方法42,43。调查样本量的影响上进行性能和下游归罪性能表明,不同方法在基因型归责和GWAS的影响可以忽略42。
归责
促进快速归责的500000个样本,我们重新编码IMPUTE223把注意力集中在已预研阶段当样本所需的单倍体非难。这个新版本的程序被称为IMPUTE4(见https://jmarchini.org/software/),但在IMPUTE2使用完全相同的隐马尔科夫模型,并产生相同的结果IMPUTE2运行时使用所有参考单隐状态(数据没有显示)。减少内存使用,增加速度我们使用紧凑的数据结构存储携带非引用指标的单体型等位基因的变体网站参考面板。不仅是这个数据结构紧凑,而且在每个阶段的它还允许forward-backward算法计算涉及的发射部分只隐马尔可夫模型和单体型的子集,携带非引用等位基因的一种有效的方式。进一步增加速度是通过只计算边际概率复制这些网站共同目标和参考数据集,然后线性插值这些snp中间这些网站,需要估算。归罪在大块的大约50000估算进行标记与一个250 kb的缓冲地区和5000样本/计算工作。合并后的每个样品处理时间对整个基因组大约10分钟。
单体型估计和基因型归罪在X染色体上
单体型估计的X染色体基因型数据应用相同的过滤步骤常染色体基因型数据,与一些额外的过滤器。性别地区和pseudo-autosomal地区(PAR),样本排除被认定为有可能性染色体非整倍性(见上图)。PAR,我们另外排除样品缺失率> 5%的标记标准。性别染色体X的区域,这导致了一个16601标记和486790个样本的数据集。1239年票面这导致一个数据集的标记和486476个样本。单体型估计和基因型进行归责两个pseudo-autosomal区域和non-pseudo常染色体区域分开,并使用相同的方法和参考数据集用于常染色体。
HLA污名和验证
为每个单独的我们定义每个位点的HLA基因型与最大后验概率的一对等位基因HLA报道*小鬼:02。我们协会执行分析(见,例如,裁判。31日)为HLA等位基因和每个疾病使用逻辑回归。风险模型(添加剂、显性、隐性或一般),如前所述31日,被用来使比较效应大小的估计。验证和进一步的细节,请参阅附加信息部分5。我们重复分析,基因型与< 0.7的最大后验概率失踪。没有观察到显著差异比较完整的分析(数据未显示)。作为一个消极的控制,我们跑协会分析与推算HLA等位基因HLA地区2型糖尿病(2849例)和心肌梗死(9725例),总共有409724个人和我们发现无显著关联(所有P> 2.40×10−4协会的Bonferroni纠正水平)与HLA等位基因,这是符合HLA地区缺乏关联最近每个表型的分析44,45
我们估计的准确性归责过程参考面板中使用5倍交叉验证样本。对于欧洲血统的样本,估计四位数精度最大后验概率的基因型是93.9%以上的所有11个位点(补充表7)。这个精度提高到96.1%以上,所有11个位点限制后HLA等位基因变异后验概率大于0.70。这导致调用所有位点率高于95.1%(补充表8)。
GWAS的站的高度
我们进行了几站的高度使用直接基因分型和估算数据形式向研究人员提供,但与样品的一个子集。具体地说,我们只包括样品的以下属性:(i)进行非难;(2)在英国白人血统子集(见上图);和(3)推断性匹配的自述性。从这一组我们选择一组344397无关的个人(补充信息)。站的高度,还有1076个人被排除在外由于失踪的表型值,留下343321协会测试。
我们使用软件BOLT-LMM (v2.2)46寻找证据,统计每一个标记和站高度之间的联系。我们报告协会统计数据基于线性混合模型(BOLT-LMM-inf)协变量如下:(i)数组(英国BiLEVE Axiom数组或英国生物库Axiom数组);(2)性别(推断);(3)时代参加了英国生物库评估中心;和(iv)主成分1 - 20。
计算主成分得分都只使用个人在英国白人祖先子集,否则用同样的方法如上所述。我们进行了测试使用单独的基因型和估算数据文件。
关联地区站高度GWAS的例子
扩展的数据图。5显示了一个示例2号染色体上的一个相关的区域。相关性(r2)之间的标记在这个区域显示一个模式,预期在连锁不平衡的背景下,和当地的重组率。stripe-like模式协会的统计数据表明多个突变发生在类似的分支系统树的底层数据,这可能是导致不同程度的因果标志(s)。之间的相关性最相关的标记和该地区的其他标记在小峰复合急剧下降47向右相关的最重要的标志。值得注意的是,这一标记基因型的估算,它指向的成功归责在这项研究中,一般来说,价值将数以百万计的标记。人类身高是一个高度多基因性状,所以提供了一个机会来检查协会,许多这样的地区和其他地区,我们视觉检查显示出类似的模式。
比较巨大的和英国生物库GWAS的结果
无花果。4 d, e和可信集分析我们过去常染色体标记,在每个数据源和筛选标记,这样加> 0.001 (GWAS人口中定义),和信息得分> 0.3英国生物库估算数据。英国生物库有16443622个这样的标记估算数据,703946年英国生物库基因分型数据,和2546872年的巨人。
对于给定的表型,95%可信的设定在一个地区协会是最小的一组标记,在一起有95%的后验概率包含标记与表型有关。我们发现可信集站的高度使用前面描述的方法33和总结的结果扩展数据图。6。重要的是要注意,这种方法是基于一个模型中,有一个因果标志的地区和基因型标记的数据是可用的。因此我们的结果应该被认为是表明一个更详细的分析,例如,区域首先分析区分独立协会的信号。
在我们的分析中,我们首先定义一组575个非重叠区域与站的高度根据以前使用过程15(见补充信息)。对于每一个研究中,我们进行了两套独立的分析找到可信的在这些区域:(A)中使用的所有标记每个研究(768502年英国生物库估算数据;106263年巨人);和(B)只使用这些标记在这两项研究(105421)。
对于每一个标记在每一个研究中,我们计算一个贝叶斯因子站高度支持与使用效果大小和标准错误,和0.22作为前33方差的影响大小。确保尺度效应是相同规模的研究我们英国生物库扩展效应大小和标准错误的残差的标准差衡量表型(站高度)后回归用于GWAS反是。然后我们确认标记重叠的效果估计两者之间的比较研究。
如果有一个因果标记和基因型的标记数据是可用的,然后一个标志的后验概率我驱动信号的关系r是由:
男朋友在哪里基米-雷克南贝叶斯因子标记吗我在r地区33。找到可靠的区域设置的95%向下的列表标记从最高到最低订购后验概率和停止当累积后达到0.95。
我们评估我们的结果的敏感性的选择之前进行同样的分析(0.02之前使用一个小得多的2(20之前)和大得多2)。之前我们发现总体的选择对结果没有影响。专门为我们报告的主要文本值,中值可信集大小未受影响的分析。大之前,single-marker可信集被数的影响除了分析B在英国生物库(从123年到122年),和可靠的标记集平均比例在所有分析的影响。小前,single-marker可信集的数量只有改变进行分析,从78年到75年的巨人,和85年到86年在英国生物库,标记在可信集的平均比例略有增加在所有分析(最大值增加从0.047到0.051)。
代码的可用性
使用IMPUTE4.0基因型进行归责。预编译的二进制文件的最新版本IMPUTE4可用https://jmarchini.org/software/。这个软件许可是免费供学术机构的研究人员使用。可在BGEN库源代码https://bitbucket.org/gavinband/bgen。使用这个库BGENIE构建。预编译的二进制文件的最新版本BGENIE可用https://jmarchini.org/software/。这个软件目前许可免费供学术机构的研究人员使用。商业组织希望使用IMPUTE4或BGENIE必须询问从牛津大学的执照。
报告总结
进一步研究信息设计是可用的自然研究报告摘要与本文有关。
数据可用性
英国生物库生成的遗传和表型数据集分析在当前的研究中可以通过英国生物库(参见数据访问过程http://www.ukbiobank.ac.uk/register-apply/)。英国生物库的遗传数据的详细信息是可用的http://www.ukbiobank.ac.uk/scientists-3/genetic-data/和http://biobank.ctsu.ox.ac.uk/crystal/label.cgi?id=100314。样本和基因数据的具体数量目前在英国生物库可能略有不同的描述。
引用
Plenge, r . M。,年代colnick, E. M. & Altshuler, D. Validating therapeutic targets through human genetics.Nat。启药物。12,581 - 594 (2013)。
英国生物库。英国生物库Axiom数组内容摘要http://www.ukbiobank.ac.uk/wp content/uploads/2014/04/uk -生物- axiom数组-内容-总结- 2014. - pdf(2014)。
英国生物库。英国生物库的基因分型和质量控制,大规模、广泛表型潜在资源http://biobank.ctsu.ox.ac.uk/crystal/docs/genotyping_qc.pdf(2015)。
年轻,我。,Wauthier, F. & Donnelly, P. Multiple novel gene-by-environment interactions modify the effect of FTO variants on body mass index.Nat。Commun。712724 (2016)。
Astle w·j . et al。人类血液细胞性状变异的等位基因的景观和链接常见的复杂疾病。细胞167年,1415 - 1429。e19 (2016)。
北斗七星,l . v . et al。小说对吸烟行为的遗传学,肺功能,和慢性阻塞性肺疾病(英国BiLEVE):英国生物库的遗传关联研究。和出版的《柳叶刀》杂志上。地中海。3,769 - 781 (2015)。
艾略特,p & Peakman t . c .英国生物样品处理和存储协议收集、处理和归档的人类血液和尿液。Int。j .论文。37,234 - 244 (2008)。
多尔蒂,a . et al .大规模人口身体活动评估使用手腕佩戴加速度计:英国生物库的研究。《公共科学图书馆•综合》12e0169649 (2017)。
米勒,k . l . et al .人口多通道脑成像在英国生物库前瞻性流行病学研究。Nat。>。19,1523 - 1536 (2016)。
彼得森,s e . et al .成像人口科学:心血管磁共振在100000年英国生物库的参与者——基本原理、挑战和方法。j . Cardiovasc。粉剂。的原因。15,46 (2013)。
科菲,et al。协议在100000年为颈动脉成像和质量保证参与者的英国生物库:开发和评估。欧元。j .上一页。心功能杂志。24,1799 - 1806 (2017)。
哈维:C。,米一个tthews, P., Collins, R., Cooper, C. & Group, U. B. M. A. Osteoporosis epidemiology in UK Biobank: a unique opportunity for international researchers.骨质疏松症Int。24,2903 - 2905 (2013)。
Sudlow, c . et al .英国生物库:一个开放存取资源识别多种复杂疾病的原因中、老年。科学硕士。12e1001779 (2015)。
英国生物库。触摸屏问卷排序,验证和依赖关系https://biobank.ctsu.ox.ac.uk/crystal/docs/TouchscreenQuestionsMainFinal.pdf(2018)。
国际遗传多发性硬化症协会& Wellcome Trust病例控制协会2。遗传风险和细胞介导免疫的主要作用机制在多发性硬化症。自然476年,214 - 219 (2011)。
Affymetrix。Axiom基因分型方案数据分析指导http://tools.thermofisher.com/content/sfs/manuals/axiom_genotyping_solution_analysis_guide.pdf(2017)。
尼尔森,j . & Wohlert m .染色体异常发现在34910年刚出生的孩子们:从13年发病率研究结果在奥尔胡斯,丹麦。嗡嗡声。麝猫。87年,81 - 83 (1991)。
列克,m . et al . 60706年分析蛋白质编码基因变异人类。自然536年,285 - 291 (2016)。
出面协调,J。,C一个rdon, L. R., Phillips, M. S. & Donnelly, P. The effects of human population structure on large genetic association studies.Nat麝猫。。36,512 - 517 (2004)。
柴田则k . et al。神秘的亲缘的混杂效应的环境风险收缩压群组研究。摩尔,麝猫。基因组医学。145-53 (2013)。
沃伊特b.f. & Pritchard j . k .混杂,在病例对照关联研究神秘的亲缘。公共科学图书馆麝猫。1e32 (2005)。
英国生物库。英国生物库:大规模的前瞻性流行病学资源的协议http://www.ukbiobank.ac.uk/wp-content/uploads/2011/11/UK-Biobank-Protocol.pdf(2007)。
豪伊,B。,Fuchsberger, C., Stephens, M., Marchini, J. & Abecasis, G. R. Fast and accurate genotype imputation in genome-wide association studies through pre-phasing.Nat麝猫。。44,955 - 959 (2012)。
奥康奈尔,j . et al .单体型估计biobank-scale数据集。Nat麝猫。。48,817 - 820 (2016)。
1000人基因组计划财团。一个全球参考人类遗传变异。自然526年,68 - 74 (2015)。
麦卡锡,s . et al .参考小组64976个单基因型污名。Nat麝猫。。48,1279 - 1283 (2016)。
黄,j . et al。改进的归责的低频和罕见变异使用UK10K单体型参考面板。Nat。Commun。68111 (2015)。
艾略特,l . et al .全基因组关联研究英国生物库的脑成像表型。Nat。Commun。91470 (2018)。
翻滚,d . et al . NHGRI GWAS目录,策划资源SNP-trait协会。核酸Res。42D1001-D1006 (2014)。
狄尔泰,a . et al . Multi-population经典HLA类型的污名。公共科学图书馆第一版。医学杂志。9e1002877 (2013)。
国际遗传多发性硬化症协会。二类HLA交互调节遗传多发性硬化的风险。Nat麝猫。。47,1107 - 1113 (2015)。
木头,a . r . et al .定义角色的常见变异基因和生物结构的成人身高。Nat麝猫。。46,1173 - 1186 (2014)。
威康信托基金会病例控制协会等。贝叶斯细化协会14位点3常见疾病的信号。Nat麝猫。。44,1294 - 1301 (2012)。
威尔士,S。,Pe一个kman, T., Sheard, S. & Almond, R. Comparison of DNA quantification methodology used in the DNA extraction protocol for the UK Biobank cohort.BMC基因组学1826 (2017)。
Affymetrix。UKB_WCSGAX:英国生物库500 k型样本数据生成Affymetrix研究服务的实验室http://biobank.ndph.ox.ac.uk/showcase/docs/affy_data_generation2017.pdf(2017)。
英国生物库。基因分型500000英国生物库的参与者:描述样本基因分型结果处理工作流和DNA的准备https://biobank.ctsu.ox.ac.uk/crystal/docs/genotyping_sample_workflow.pdf(2015)。
Affymetrix。UKB_WCSGAX:英国生物库500 k Affymetrix研究服务实验室样品处理http://biobank.ndph.ox.ac.uk/showcase/docs/affy_lab_process2017.pdf(2017)。
加林斯基,k . j . et al .快速的主成分分析显示ADH1B在欧洲和东亚的趋同进化。点。j .的嗡嗡声。麝猫。98年,456 - 472 (2016)。
价格,a . l . et al。远程LD在混血人口可以混淆基因组扫描。点。j .的嗡嗡声。麝猫。83年,132 - 135,作者回复135 - 139 (2008)。
劳森,d J。,Hellenthal, G., Myers, S. & Falush, D. Inference of population structure using dense haplotype data.公共科学图书馆麝猫。8e1002453 (2012)。
Manichaikul, a . et al .健壮的关系推理在全基因组关联研究。生物信息学26,2867 - 2873 (2010)。
Loh, P.-R。,P一个lamara, P. F. & Price, A. L. Fast and accurate long-range phasing in a UK Biobank cohort.Nat麝猫。。48,811 - 816 (2016)。
Loh, P.-R。et al . Reference-based逐步使用单体型引用财团面板。Nat麝猫。。48,1443 - 1448 (2016)。
韦伯,t·r . et al .系统评价基因多效性标识6进一步与冠状动脉疾病相关基因座。j。科尔。心功能杂志。69年,823 - 836 (2017)。
Fuchsberger,等。2型糖尿病的遗传结构。自然536年41-47 (2016)。
Loh, P.-R。et al。高效的贝叶斯混合模型分析增加与大群体力量的联系。Nat麝猫。。47,284 - 290 (2015)。
国际人类基因组单体型图财团。人类基因组单体型图。自然437年,1299 - 1320 (2005)。
兰,j . et al。重复的可接受性网络混合饮食评估前24小时膳食摄入量:牛津WebQ在英国生物库的管理。Br。j .减轻。115年,681 - 686 (2016)。
确认
我们承认威康信托核心奖090532 / Z / 09 / Z - 203141 / Z / 16 / Z和赠款095552 / Z / 11 / Z(下)100956 / Z / 13 / Z(通用)和100308 / Z / 12 / Z(交流)。J.M.是617306年由欧洲研究委员会资助。S.L.支持澳大利亚NHMRC职业发展奖学金1053756。样品处理和基因分型是国立卫生研究所的支持下,医学研究委员会和英国心脏基金会。我们感谢人类遗传学研究计算核心康中心寻求帮助的计算工作量。我们感谢Affymetrix讨论有关质量控制。我们感谢a .年轻,a狄尔泰和l . Moutsianas援助方面的数据分析。我们承认英国生物库协调中心人员为他们的角色在提取DNA来做这个项目。我们感谢m . Kuzma-Kuzniarska (http://mybioscience.org/图1)。
审核人信息
自然由于大肠银行,m . Boehnke b . Pasaniuc d·麦克阿瑟,另一个匿名的评论家(s)为他们的贡献的同行评审工作。
作者信息
作者和联系
贡献
A.Y.,N.A. and M.E. coordinated data availability. Data analysis was performed as follows: quality control analysis (C.B., C.F., D.P. and S.W.), HLA imputation and association testing (A.C., A.M. and D.V), phasing, imputation, file formats and multiple trait analysis (O.D., J.O., G.B., K.S., L.T.E. and J.M.) and GWAS testing (C.B., C.F. and J.M.). Supervision of these activities was provided by G.M., P.D. and J.M. C.B., C.F., A.C., S.L., N.A., G.M., P.D. and J.M. wrote the paper.
相应的作者
道德声明
相互竞争的利益
J.M.是创始人和主任Gensci警察局,通用和年代。L. are partners in Peptide Groove LLP. G.M. and P.D. are founders and directors of Genomics Plc. The remaining authors declare no competing financial interests.
额外的信息
出版商的注意施普林格自然保持中立在发表关于司法主权地图和所属机构。
扩展数据数据和表
扩展数据图1总结纸浆包质量控制。
一个- - - - - -c三块显示,杂合性缺失率,我们用来标记质量差的样品(n= 488377个样本)。面板一个和b显示为每个样本之前和之后的杂合性,分别纠正祖先背景使用主成分。符号(形状和颜色)表示每个参与者的自我报告的种族背景。面板c显示的968个样本标记为异常值(红色),和所有其他的样品(黑色),与其他两个形状相同的情节。垂直线显示阈值我们用来调用样品作为离群值缺失率。在所有情节缺失率数据转换为分对数的规模,但与轴带注释的原始值。
扩展数据图2的例子强度数据和基因型要求标记不同的等位基因频率。
每个sub-figure显示强度数据为一个标记在六个不同的批次。批次标签前缀“UKBiLEVEAX”只包含样本类型使用英国BiLEVE Axiom数组,和那些前缀“批处理”只包含样本类型使用英国生物库Axiom数组。每个点代表一个样本,根据的推断基因型彩色标记。的x和y轴强度的转换探头组针对每个等位基因A和B(见补充信息对探针集)的定义。椭圆表示后验概率分布的位置和形状(二维多元正态)改变了三种基因型的强度的批处理。即,每个椭圆画,它包含85%的概率密度。看到AffymetrixAxiom基因分型方案数据分析指导16Affymetrix基因型调用的更多细节。计算每个标记的,我们使用发布英国生物库中的所有样本基因型数据。一个标记加器为0.077,布置得井然有序基因型集群。b,强度标记与加0.00092布置得井然有序基因型集群。像预计的那样在哈迪温伯格平衡,没有和样品小纯合子基因型的实例。c,强度标记加器为0.00066,杂合子的集群并不是分开的大结合体主要集中在一些批次,使其更难以自信地称之为杂合的基因型。
扩展数据图3指的是主成分得分为每个自我出生的国家。
每一列显示每个元素一个主成分和主成分得分均值为个人出生在标签的国家,按比例缩小的标准差的主成分得分。在每一列元素只有彩色如果这个国家有一个非零系数(P< 10−5;双面的t以及)与出生地线性模型预测和主成分得分结果(n= 487848个样本)。国家(行)已被命令使用层次聚类(“hclust”函数R)。每个国家的标签旁边的符号显示最常见的种族背景类别之间的参与者在这个国家出生的。举例来说,最常见的自我报告的种族背景的参与者出生在斯里兰卡是“任何其他亚洲背景”。不到20个人在那里出生的国家被排除在分析之外。
扩展数据图4分配分数在常染色体标记估算数据集的信息。
左上角图显示了完整的分数的分布信息。剩下的面板显示部分加分布;加器> 5%、1% < 5%、0.1%≤≤乘加加< 1%、0.01%≤乘加<≤0.1%和0.001%加< 0.01%。
扩展数据图5例区域站高度GWAS的协会。
GWAS协会统计数据(P值)站高度关注的染色体2 ~ 3 mb地区没有达到全基因组意义巨人(2014)分析,但在英国生物库(线性混合模型;看到方法)。的P多个测试值显示不调整。英国生物库的标记基因分型表现为钻石,和估算标记圆圈。最小的两个标记P值为每个基因分型数据和估算数据放大并强调了黑色的轮廓,和其他英国生物库标记颜色根据其相关性(r2),这两个之一。即基因标记与领先的基因标记(rs17713396)和估算指标主要估算标记(rs12714401)。标记与r2小于0.1的值显示为黑色或绿色的。
扩展数据图6比较精细定位的巨人(2014)和英国生物库估算数据。
这里我们总结我们的可靠的分析结果在巨大(2014)和575年英国生物库基因组区域与站在这两项研究(见高度相关方法)。情节上的红色实线表示x=y。一个,情节比较标记的数量的95%可信集大小小于18标记在这两项研究(363个地区在左边的情节;445在右边的情节)。b,c,从分析考虑所有情节都标记在每一个研究。在b我们展示,每个地区的比例分析对于一个给定的研究中使用的标记的95%可信的研究。情节包含相同的363个地区如左边所示图一个。在c我们总结一下,575个地区,多少重量我们英国生物库的分析放在标记,分析巨人(2014)表示很重要。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
Bycroft C。,Freeman, C., Petkova, D.et al。英国生物库资源深表现型和基因数据。自然562年,203 - 209 (2018)。https://doi.org/10.1038/s41586 - 018 - 0579 - z
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586 - 018 - 0579 - z
关键字
- 深的表现型
- 基因型归责
- 人体测量特征的遗传调查(巨大的)
- Pseudo-autosomal区域(PAR)
- 接受设置
进一步的阅读
儿童哮喘的影响在成人身高:证据来自英国生物库
BMC医学(2022)
框架研究大陆血统的英国生物库
人类基因组学(2022)
识别基因与大脑体积差异通过组织特定的转录组推理从GWAS汇总数据
BMC生物信息学(2022)
精神表现的罕见变异在医学上可操作的基因:PheWAS方法
BMC基因组学(2022)
遗传易感性、基于屏幕的久坐不动的活动和冠心病的发病率
BMC医学(2022)