摘要
一种新型高致病性冠状病毒(严重急性呼吸综合征冠状病毒-2,SARS-CoV-2)从2019年12月开始在中国湖北省武汉市爆发,并迅速蔓延到全国和世界其他国家1,2,3..为了在原子水平上更好地理解感染的最初步骤,我们确定了SARS-CoV-2与细胞受体ACE2结合的刺突蛋白的受体结合域(RBD)的晶体结构。SARS-CoV-2 RBD的整体ACE2结合模式与SARS-CoV RBD几乎相同,后者也使用ACE2作为细胞受体4.结构分析发现了SARS-CoV-2 RBD中对ACE2结合至关重要的残基,其中大多数要么高度保守,要么与SARS-CoV RBD中的残基具有相似的侧链性质。这种结构和序列的相似性强烈表明SARS- cov -2和SARS- cov rbd之间存在趋同进化,以改善与ACE2的结合,尽管SARS- cov -2并不在SARS和SARS相关冠状病毒中聚集1,2,3.,5.我们还分析了两种以RBD为靶点的SARS-CoV抗体的表位,以便与SARS-CoV-2 RBD结合,为未来交叉反应抗体的鉴定提供了见解。
主要
高致病性冠状病毒SARS-CoV-2在武汉出现并迅速在国际上传播,构成了严重的全球公共卫生紧急事件1,2,3..与2003年感染致病性SARS-CoV和2012年感染中东呼吸综合征冠状病毒(MERS-CoV)的患者相似,感染SARS-CoV的患者表现出一系列症状,包括干咳、发烧、头痛、呼吸困难和肺炎,估计死亡率在3%至5%之间6,7,8.自2019年12月疫情发生以来,新冠病毒已在中国蔓延,并扩散到全球80多个国家和地区。截至2020年3月5日,中国已确诊80565例感染,3015例感染患者已死亡(https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports/).由于SARS-CoV-2在全球迅速传播,世界卫生组织宣布进入国际关注的突发公共卫生事件。
对冠状病毒基因组的系统发育分析显示,SARS-CoV-2是该病毒的一种Betacoronavirus属,包括SARS-CoV、MERS-CoV、蝙蝠sars相关冠状病毒(SARSr-CoV),以及在人类和不同动物物种中发现的其他病毒1,2,3.,5.蝙蝠冠状病毒RaTG13似乎是SARS-CoV-2最近的亲戚,在刺突中有超过93.1%的序列相同(年代)基因。然而,SARS-CoV和其他SARS-CoV与SARS-CoV-2不同,它们的序列相同度不到80%1.
冠状病毒利用胞膜上的同源三聚体穗状糖蛋白(包括每个穗状单体中的S1亚基和S2亚基)与细胞受体结合。这种结合触发了一系列的事件,导致细胞和病毒膜之间的融合,从而进入细胞。先前对SARS-CoV刺突蛋白及其与细胞受体ACE2相互作用的冷冻电子显微镜研究表明,受体结合诱导S1与ACE2分离,促使S2从亚稳态的预融合过渡到更稳定的融合后状态,这对膜融合至关重要9,10,11,12.因此,与ACE2受体结合是SARS-CoV进入靶细胞的关键的初始步骤。最近的研究也强调了ACE2在介导SARS-CoV-2进入中的重要作用1,13,14,15.表达ACE2的HeLa细胞容易感染SARS-CoV-2,而不表达ACE2的HeLa细胞则不容易感染1.体外结合测量还显示,SARS-CoV-2 RBD与ACE2结合具有低纳摩尔的亲和力,表明RBD是S1亚基中负责SARS-CoV-2与ACE2结合的关键功能成分13,16.
最近在两项独立的研究中报道了SARS-CoV-2刺突三聚体的低温电子显微镜结构13,17.然而,对一个可用的穗状结构的检查显示,RBD的建模不完整,特别是对直接与ACE2相互作用的受体结合基motif (RBM)17.对SARS-CoV-2 RBD和ACE2之间相互作用的计算机建模已经确定了一些可能参与相互作用的残留物;然而,介导相互作用的实际残基尚不清楚18.此外,尽管从SARS-CoV感染康复患者的血清或血浆中检测到交叉反应性的SARS-CoV-2中和活性15在美国,没有分离的SARS-CoV单克隆抗体能够中和SARS-CoV-216,17.这些发现突出了SARS-CoV和SARS-CoV-2 rbd之间的一些内在序列和结构差异。
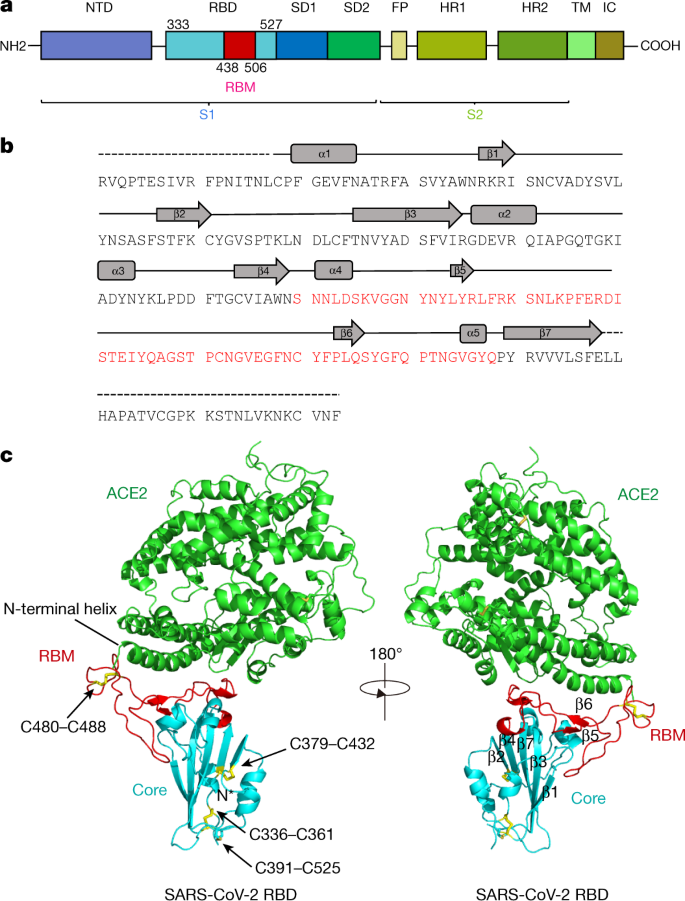
为了以更高的分辨率阐明SARS-CoV-2 RBD和ACE2之间的相互作用,我们使用x射线晶体学确定了SARS-CoV-2 RBD - ACE2复合体的结构。这些原子层面的结构信息极大地提高了我们对SARS-CoV-2与敏感细胞之间相互作用的理解,为中和抗体提供了精确的靶点,并有助于基于结构的疫苗设计,这是目前对抗SARS-CoV-2急需的。具体来说,我们表达了SARS-CoV-2的RBD(残基Arg319-Phe541)(图。1 a, b)和Hi5昆虫细胞中ACE2的n端肽酶结构域(残基Ser19-Asp615),并通过Ni-NTA亲和纯化和凝胶过滤对其进行纯化。1).利用SARS-CoV的RBD和ACE2结构作为搜索模型,通过分子置换确定复合物的结构4,并细化到分辨率2.45 Å与最终R工作而且R免费的系数分别为19.6%和23.7%。2和扩展数据表1).最终模型包含SARS-CoV-2 RBD的Thr333-Gly526残基,ACE2 n端肽酶结构域的Ser19-Asp615残基,一个锌离子,四个N-乙酰-β-氨基葡萄糖(NAG)聚糖与ACE2 Asn90、Asn322、Asn546和RBD Asn343以及80个水分子连接。
SARS-CoV-2 RBD有一个扭曲的五股反平行β片(β1、β2、β3、β4和β7),其核心是短的螺旋和环。1 b, c).在核心的β4和β7链之间,有一个扩展的插入点,包含短的β5和β6链,α4和α5螺旋和环(图1)。1 b, c).这种扩展插入就是RBM,它包含了SARS-CoV-2与ACE2结合的大部分接触残基。在RBD中总共发现了9个半胱氨酸残基,其中8个形成了4对二硫键,在最终模型中被分解。在这四对结构中,有三对位于核心(Cys336-Cys361, Cys379-Cys432和Cys391-Cys525),这有助于稳定β片结构(图1)。1 c);剩下的一对(Cys480-Cys488)连接RBM远端环(图4)。1 c).ACE2的n端肽酶结构域有两个裂片,在它们之间形成肽底物结合位点。SARS-CoV-2 RBD中的扩展RBM与ACE2小瓣的底部接触,RBM的外表面为凹形,容纳ACE2的n端螺旋(图1)。1 c).SARS-CoV-2 RBD的整体结构与SARS-CoV RBD相似(扩展数据图)。3),对于174个对齐的Cα原子。即使在序列变化较多的RBM中,其整体结构也与SARS-CoV RBD高度相似(rm.s.d.为1.3 Å),仅在远端有一个明显的构像变化(扩展数据图)。3).SARS-CoV-2 RBD与ACE2的整体结合模式也与之前确定的SARS-CoV RBD - ACE2复合体结构中观察到的模式几乎相同4(扩展数据图。3 b).
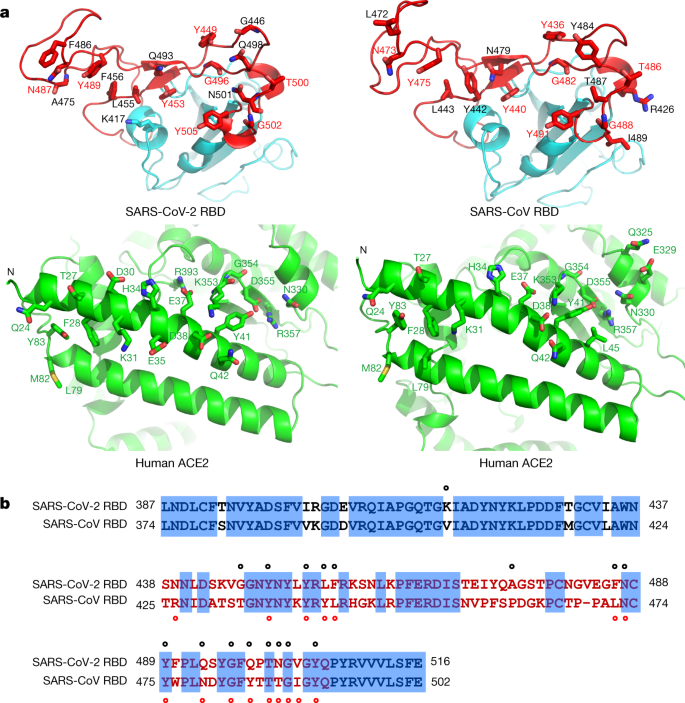
ACE2的n端螺旋被RBM的外表面承载后,形成了1687 Å的大埋面2(8642在RBD和823 Å2在SARS-CoV-2 RBD-ACE2接口上。高度相似的埋藏表面1699 Å2由SARS-CoV RBD (869 Å)贡献2)和ACE2 (830 Å)2)也在SARS-CoV RBD-ACE2接口上观察到。在距离截止为4 Å的情况下,RBD共有17个残基与ACE2的20个残基相接触(图1)。2和扩展数据表2).对SARS-CoV RBD和ACE2之间的界面进行分析发现,SARS-CoV RBD共有16个残基与ACE2的20个残基相接触。2和扩展数据表2).在与两种不同rbd相互作用的20个ACE2残基中,有17个残基在两种相互作用之间共享,大多数接触残基位于n端螺旋(图1)。2和扩展数据表2).
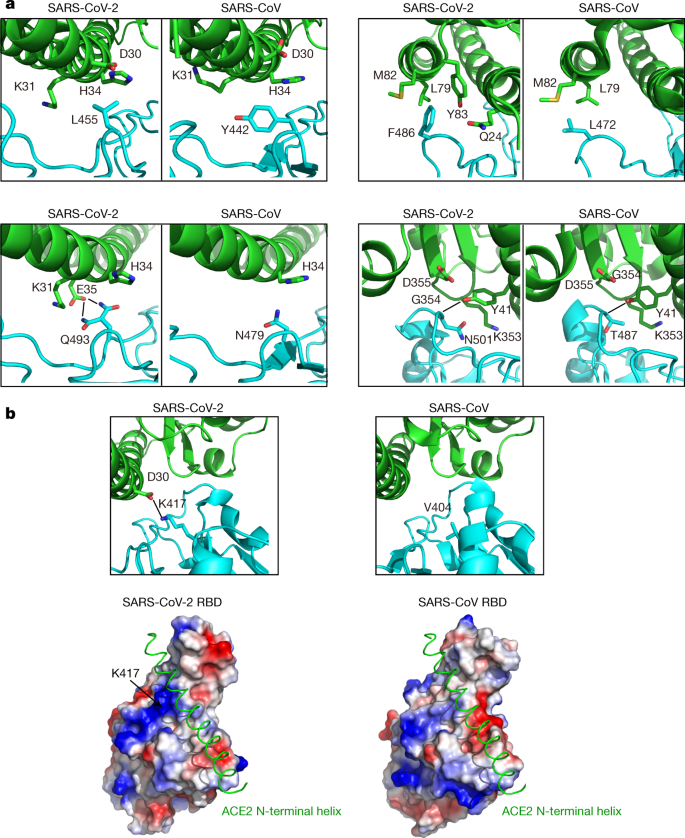
为了比较SARS-CoV-2和SARS-CoV rbd上ace2相互作用的残基,我们使用结构引导序列比对,并将它们映射到各自的序列(图1)。2 b).在两个RBMs与ACE2相互作用共用的14个氨基酸位置中,有8个在两个RBMs之间有相同的残基,分别是SARS-CoV-2/SARS-CoV的Tyr449/Tyr436、Tyr453/Tyr440、Asn487/Asn473、Tyr489/Tyr475、Gly496/Gly482、Thr500/Thr486、Gly502/Gly488和Tyr505/Tyr491(图1)。2 b).SARS-CoV-2/SARS-CoV的Leu455/Tyr442、Phe456/Leu443、Phe486/Leu472、Gln493/Asn479和Asn501/Thr487这5个位点的残基虽然侧链不同,但具有相似的生化性质(图4)。2 b).其余位置在Gln498/Tyr484位置(图。2 b),其中SARS-CoV-2的Gln498和SARS-CoV的Tyr484都与ACE2的Asp38、Tyr41、Gln42、Leu45和Lys353相互作用。在6个残基发生变化的RBD位置中,SARS-CoV残基Tyr442、Leu472、Asn479和Thr487此前已被证明对结合ACE2至关重要18.在Leu455/Tyr442位点,SARS-CoV-2的Leu455和SARS-CoV的Tyr442与ACE2的Asp30、Lys31和His34具有类似的相互作用(图1)。3).在Phe486/Leu472位点,SARS-CoV-2的Phe486与ACE2的Gln24、Leu79、Met82和Tyr83相互作用,而SARS-CoV的Leu472与ACE2的Leu79和Met82相互作用较少(图4)。3).在Gln493/Asn479位置,SARS-CoV-2的Gln493与ACE2的Lys31、His34和Glu35相互作用,与Glu35形成氢键;SARS-CoV的Asn479只与ACE2的His34相互作用。3).在Asn501/Thr487位置,这两个残基与ACE2的Tyr41、Lys353、Gly354和Asp355有相似的相互作用(图1)。3).SARS-CoV-2的Asn501和Thr487都与ACE2的Tyr41形成氢键。3).在RBM外,SARS-CoV-2中有一个独特的ACE2相互作用残基(Lys417),它与ACE2的Asp30形成盐桥相互作用(图4)。3 b).这个位置被SARS-CoV RBD中的缬氨酸取代,该缬氨酸不能参与ACE2结合(图2)。2b,3 b).此外,对表面静电势的比较还发现,在SARS-CoV-2 RBD上有一个由Lys417贡献的带正电荷的斑块,而在SARS-CoV RBD上没有(图4)。3 b).这些微妙不同的ACE2相互作用可能导致了SARS-CoV-2和SARS-CoV对ACE2受体的结合亲和力的差异(分别为4.7 nM和31 nM)(扩展数据图)。4).
一个在RBM中,SARS-CoV-2和SARS-CoV位置周围的相互作用与残基改变。SARS-CoV-2和SARS-CoV rbd以青色显示。ACE2用绿色表示。b,分别在2型和2型rbd的RBM和静电势图之外的rbd的K417和V404位置周围的相互作用。K417在SARS-CoV-2 RBD中的位置用黑色箭头表示。ACE2的n端螺旋如图所示为绿色丝带。SARS-CoV RBD-ACE2复合体的蛋白数据库(PDB)代码为2 ajf.
RBD-ACE2界面的一个显著和共同特征是亲水性相互作用网络。在SARS-CoV-2 RBD-ACE2界面上有13个氢键和2个盐桥,在SARS-CoV RBD-ACE2界面上有13个氢键和3个盐桥(表2)1).第二个共同特征是多个酪氨酸残基参与其中,与极性羟基形成氢键相互作用。其中包括来自SARS-CoV-2 RBD的Tyr449、Tyr489和Tyr505,以及来自SARS-CoV RBD的Tyr436、Tyr475和Tyr4911).第三个共同特征可能存在于ACE2的asn90连接聚糖中,它们与不同的rbd结合。在SARS-CoV RBD-ACE2复合物的结构中,有一条asn90连接的NAG-NAG - β-链d-甘露糖与SARS-CoV RBD的Thr402接触(扩展数据图。5),这种甘氨酸- RBD相互作用被认为在ACE2结合SARS-CoV RBD中发挥重要作用4,19.在SARS-CoV-2 RBD - ACE2结构中,密度只支持连接到ACE2 Asn90的第一个NAG的建模,并且没有观察到该NAG与SARS-CoV-2 RBD之间的相互作用(扩展数据图)。5 b).然而,这并不排除第一个NAG后的聚糖可能与SARS-CoV-2 RBD相互作用,并可能在ACE2结合SARS-CoV-2 RBD中发挥重要作用。总之,我们的研究结果表明,尽管在RBM内外观察到的一些ACE2相互作用不同,但SARS-CoV-2 RBD-ACE2和SARS-CoV RBD-ACE2界面在埋藏表面积、相互作用残基的数量和亲水性相互作用网络方面具有很大的相似性(图1)。3 a, b).这种相似性有力地证明了SARS-CoV-2和SARS-CoV RBD结构的趋同进化,以提高对同一ACE2受体的结合亲和力,尽管SARS-CoV-2并不聚集在SARS-CoV和SARSr-CoV之间Betacoronavirus属。
与较高的结构相似性一致,我们发现ACE2与SARS-CoV-2和SARS-CoV rbd之间的结合亲和力也处于相似的范围。具体来说,平衡解离常数(KDACE2和SARS-CoV-2的RBD为4.7 nM, ACE2和SARS-CoV的RBD为31 nM(扩展数据图)。4).其他小组也报告了类似的结果13,16.然而,这与最近的一份报告略有不同,该报告发现ACE2与SARS-CoV-2刺突三聚体之间的结合增加了约20倍(KD为14.7 nM),与ACE2和SARS-CoV RBD-SD1 (KD的325海里)17.这可能是由于实验中使用了不同的蛋白质,也可能是因为其他未知的原因。然而,仅结合亲和力不太可能解释SARS-CoV-2不寻常的传播能力。其他因素,如SARS-CoV-2刺突蛋白S1-S2边界上独特的“rar”furin裂解位点,可能在促进SARS-CoV-2的快速人际传播方面发挥更重要的作用。
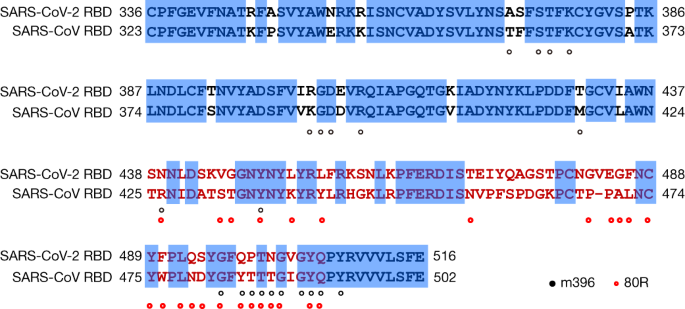
中和抗体是免疫系统对抗病毒感染的重要组成部分。据报道,马抗SARS-CoV血清和来自SARS-CoV感染患者的恢复期血清可以交叉中和SARS-CoV-21,15增强了SARS-CoV-2和SARS-CoV的rbd之间的结构相似性。这种相似性也增加了先前鉴定的SARS-CoV单克隆抗体在临床快速应用的希望。然而,目前尚无针对SARS-CoV的抗体(m396、S230、80R和CR3014)对SARS-CoV-2的刺突蛋白或RBD具有显著的交叉结合和中和活性16,17,20.,21,22,23.一个例外是SARS-CoV抗体CR3022,它与SARS-CoV-2 RBD结合KD虽然其对SARS-CoV-2的中和活性尚未有报道16.目前,我们还不确定SARS-CoV或SARS-CoV-2的rbd上CR3022的表位具体位于哪里。在三种不能与SARS-CoV-2 RBD结合的抗体中,有两种抗体(m396和80R)的表位通过SARS-CoV RBD - fab复合体的高分辨率晶体结构测定得到了解析20.,21.通过将这些表位残基映射到SARS-CoV RBD序列上,使其与SARS-CoV-2 RBD序列一致。4),我们发现抗体m396在SARS-CoV-2 RBD的21个表位位置中有7个残留变化(图。4).在SARS-CoV-2抗体80R的25个表位位置中,有16个残留变化(图1)。4).这可能为m396和80R与SARS-CoV-2缺乏交叉反应提供了结构性基础。马抗SARS-CoV血清与康复患者血清或血浆的交叉中和揭示了在识别这两种冠状病毒之间具有交叉反应性的抗体方面的巨大潜力1,15.刺突蛋白中保守的非rbd区域,如S2亚基,是交叉反应抗体的潜在靶点。尽管RBD保守性较差,但SARS-CoV-2和SARS-CoV RBD之间存在相同的残基,即使是在更可变的RBM中(图1)。4).考虑到RBD是受体结合的重要区域,针对RBD中保守表位的抗体也将为开发对抗包括SARS-CoV-2在内的多种冠状病毒的高效交叉反应治疗药物提供巨大潜力。
方法
数据报告
没有使用统计方法来预先确定样本量。实验不是随机的,研究人员在实验和结果评估过程中没有对分配进行盲法。
蛋白的表达和纯化
利用bacto - bac杆状病毒系统(Invitrogen)表达SARS-CoV-2的RBD和人ACE2的n端肽酶结构域。将带有n端gp67信号肽分泌和c端6×His标签纯化的SARS-CoV-2 RBD(残基Arg319-Phe541)插入pFastBac-Dual载体(Invitrogen)。将构建物转化为细菌DH10Bac感受态细胞,提取的bacmid用Cellfectin II Reagent (Invitrogen)转染Sf9细胞。收集低滴度病毒,扩增成高滴度病毒株,以2 × 10的密度感染Hi5细胞6在感染60小时后收集含有分泌的SARS-CoV-2 RBD的细胞培养上清,浓缩和缓冲交换到HBS (10 mM HEPES, pH 7.2, 150 mM NaCl)。用Ni-NTA树脂(GE Healthcare)捕获SARS-CoV-2 RBD,用500 mM咪唑在HBS缓冲液中洗脱。然后使用与HBS缓冲液预平衡的Superdex 200柱(GE Healthcare)通过凝胶过滤层析纯化SARS-CoV-2 RBD。收集含有SARS-CoV-2 RBD的组分。
人类ACE2的n端肽酶结构域(残基Ser19-Asp615)表达和纯化的方法与SARS-CoV-2 RBD的基本相同。为了纯化SARS-CoV-2 RBD - ACE2复合物,ACE2与SARS-CoV-2 RBD在HBS缓冲液中冰上孵育1小时,然后对混合物进行凝胶过滤色谱。含有复合物的组分被汇集和浓缩到13毫克毫升−1.
结晶和数据收集
结晶在室温下,在含有100 mM MES, pH 6.5, 10% PEG 5000 MME和12% 1-丙醇的井中成功生长。将200 nl SARS-CoV-2 RBD-ACE2复合物混合在20 mM Tris pH 7.5, 150 mM NaCl和200 nl well溶液中制成滴剂。收集晶体,在100 mM MES、pH 6.5、10% PEG 5000 MME、12% 1-丙醇和20%甘油中短暂浸泡,然后在液氮中快速冷冻。衍射数据是在上海同步加速器研究设施的BL17U1光束线上采集的,波长为1.07180 Å,波长为100 K。衍射数据采用水族管道自动处理24数据处理统计列于扩展数据表中1.
结构确定和细化
用CCP4组份中的PHASER分子置换法确定结构25.使用的搜索模型包括ACE2细胞外结构域和SARS-CoV RBD (PDB代码)2 ajf).利用ARP/wARP对原子进行更新和细化,从而改善密度图26.随后分别使用COOT和PHENIX进行模型构建和细化27,28.最终的Ramachandran统计数据:96.44%赞成,3.56%允许,0.00%异常值。结构细化统计信息列在扩展数据表中1.所有的结构图都是用PyMol生成的29.
表面等离子体共振实验
使用Biacore T200 (GE Healthcare)和由10 mM HEPES pH 7.2、150 mM NaCl和0.05% Tween-20组成的流动缓冲液,将ACE2固定在CM5传感器芯片(GE Healthcare)上,达到约500个响应单位的水平。连续稀释的SARS-CoV RBD和SARS-CoV-2 RBD以62.5 - 1.9 nM的浓度通过。使用Biacore评估软件(GE Healthcare)将所得数据拟合为1:1绑定模型。
报告总结
关于研究设计的进一步信息可在自然研究报告摘要链接到这篇论文。
数据可用性
SARS-CoV-2 RBD-ACE2复合体的坐标和结构因子文件已存入蛋白质数据库(PDB),登录号如下6 m0j.
参考文献
周,P.等。与一种可能源自蝙蝠的新型冠状病毒有关的肺炎爆发。自然579, 270 - 273(2020)。
吴,F.等。中国发现一种与人类呼吸道疾病有关的新型冠状病毒。自然579, 265 - 269(2020)。
朱,N.等。2019年,来自中国肺炎患者的一种新型冠状病毒。心血管病。j .地中海.382, 727 - 733(2020)。
李芳,李伟,法尔zan M. & Harrison S. C. SARS冠状病毒刺突受体结合域与受体复合物的结构。科学309, 1864 - 1868(2005)。
卢,R.等。2019新型冠状病毒的基因组特征和流行病学:对病毒起源和受体结合的影响。《柳叶刀》395, 565 - 574(2020)。
黄,C.等。武汉市新型冠状病毒感染患者的临床特征《柳叶刀》395,497 - 506 (2020)。
Liu, K.等。湖北省三级医院新型冠状病毒感染病例的临床特征下巴。地中海。j .(英格兰)。https://doi.org/10.1097/CM9.0000000000000744(2020)。
王,D.等。武汉市138例新型冠状病毒感染的肺炎住院患者的临床特征j。医学协会。.323, 1061 - 1069(2020)。
桂,M.等。SARS-CoV刺突糖蛋白的低温电子显微镜结构揭示了受体结合的先决构象状态。细胞Res.27, 119 - 129(2017)。
宋伟,桂明,王晓霞,向阳。SARS冠状病毒棘突糖蛋白与宿主细胞受体ACE2复合物的低温电镜结构。公共科学图书馆Pathog.14e1007236(2018)。
柯什多弗,R. N.等。稳定的冠状病毒尖刺对受体识别或蛋白质水解引起的构象变化具有抗性。科学。代表.815701(2018)。
袁,杨,等。中东呼吸综合征冠状病毒和sars冠状病毒刺突糖蛋白的Cryo-EM结构揭示了受体的动态结合结构域。Nat。Commun.815092(2017)。
墙壁,a.c.等。新冠病毒穗状糖蛋白的结构、功能和抗原性。细胞https://doi.org/10.1016/j.cell.2020.02.058(2020)。
Letko, M., Marzi, A. & Munster, V. SARS-CoV-2和其他B系β冠状病毒的细胞进入和受体使用的功能评估。Nat。Microbiol.5, 562 - 569(2020)。
霍夫曼等人。SARS-CoV-2细胞进入依赖于ACE2和TMPRSS2,并被临床证实的蛋白酶抑制剂阻断。细胞https://doi.org/10.1016/j.cell.2020.02.052(2020)。
田,x,等。SARS冠状病毒特异性人单克隆抗体与2019新型冠状病毒刺突蛋白的有效结合。紧急情况。微生物感染.9, 382 - 385(2020)。
Wrapp, D.等。预融合构象中2019-nCoV尖刺的Cryo-EM结构。科学367, 1260 - 1263(2020)。
万勇,尚杰,Graham, R., Baric, R. S.,李峰,武汉新型冠状病毒的受体识别:基于近十年SARS结构研究的分析。j .性研究.94e00127-20(2020)。
Li, W.等。sars冠状病毒适应人类ACE2的受体和病毒决定因素EMBO J.24, 1634 - 1643(2005)。
Prabakaran, p等。与中和抗体复合物的严重急性呼吸综合征冠状病毒受体结合域的结构。生物。化学.281, 15829 - 15836(2006)。
黄,W. C.等。人抗严重急性呼吸综合征刺突蛋白抗体80R中和的结构基础。生物。化学.281, 34610 - 34616(2006)。
墙壁,a.c.等。意想不到的受体功能模拟阐明了冠状病毒融合的激活。细胞176, 1026 - 1039(2019)。
van den Brink, E. N.等。与严重急性呼吸综合征冠状病毒刺突蛋白和核衣壳蛋白结合的人单克隆抗体的分子和生物学特性j .性研究.79, 1635 - 1644(2005)。
于,F.等。水族:生物大分子晶体学束线的自动数据处理和实验信息管理系统。j:。Crystallogr.52, 472 - 477(2019)。
McCoy, A. J.等。移相器晶体软件。j:。Crystallogr.40, 658 - 674(2007)。
科恩,s.x等人。ARP/wARP和分子替换:下一代。Acta Crystallogr。D6449-60(2008)。
埃姆斯利,P. &考坦,K.库特:分子图形的模型构建工具。Acta Crystallogr。D60,2126 - 2132 (2004)。
亚当斯,P. D.等。构建新的自动晶体结构测定软件。Acta Crystallogr。D58, 1948 - 1954(2002)。
Janson, G., Zhang, C., Prado, m.g. & Paiardini, A. PyMod 2.0: PyMOL中蛋白质序列结构分析和同源建模的改进。生物信息学33, 444 - 446(2017)。
确认
我们感谢上海同步加速器研究中心BL17U1光束线的工作人员对数据的收集和处理。我们感谢清华大学蛋白质研究技术中心x射线晶体学平台的工作人员提供的设备支持。本研究得到国家科学研究与发展重点计划项目(批准号:2016YFD0500307)资助。清华大学创新科研计划项目(20201080053)、国家自然科学基金奖励项目(81530065)、北京市科委(171100000517-001、171100000517-003)、腾讯基金、水地基金、TH资本资助。
作者信息
作者和联系
贡献
J.L.和J.G.在J.Y.和S.S. Q.Z.的帮助下进行蛋白表达、纯化、结晶、衍射数据收集和结构确定,X.S.帮助蛋白表达和纯化。s.f., H.Z.和Q.W.帮助收集结晶和衍射数据。x.w和l.z构思、设计和指导了这项研究。j.l., j.g., j.y., s.s., L.Z.和X.W.分析数据,生成数据并撰写手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争利益。
额外的信息
同行审查的信息自然感谢李俊荣和其他匿名审稿人对这项工作的同行评审所做的贡献。同行评审报告可用。
出版商的注意施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
扩展的数据图和表
图1纯化SARS-CoV-2 RBD-ACE2复合体。
一个,复合物的凝胶过滤色谱。1、SARS-CoV-2 RBD二聚体- ace2;2、SARS-CoV-2 RBD单体- ace2;3、SARS-CoV-2 RBD单体。b, SDS-PAGE凝胶的配合物。
图2电子密度图。
2Fo−Fc电子密度图等高线为1.5σ图中为SARS-CoV-2 RBD(红色)与ACE2(绿色)的结合界面。
图3 SARS-CoV-2和SARS-CoV的rbd结构比较及其与ACE2受体的结合方式。
一个, SARS-CoV-2 RBD(核心为青色,RBM为红色)和SARS-CoV RBD(核心为橙色,RBM为蓝色)结构的对齐。bSARS-CoV-2 RBD-ACE2和SARS-CoV RBD-ACE2复合物的结构比对。SARS-CoV-2 RBD为青色和红色,其相互作用的ACE2为绿色。SARS-CoV RBD显示为橙色和蓝色,其相互作用的ACE2显示在鲑鱼中。SARS-CoV RBD-ACE2复合体的PDB代码为2 ajf.
图4表面等离子体共振传感器图。
固定化人ACE2与SARS-CoV-2 RBD(左)和SARS-CoV RBD(右)的结合曲线。数据用不同颜色的线表示,数据与1:1绑定模型的最佳拟合用黑色表示。
图5 ACE2的asn90 -连接聚糖。
一个, ACE2(鲑鱼)asn90连接聚糖与SARS-CoV RBD(橙色)之间的界面。bACE2的Asn90-linked glycan(绿色)与SARS-CoV-2 RBD(青色)之间的界面。2Fo−Fc电子密度的轮廓为1.5σ也会显示出来。
权利和权限
关于这篇文章
引用这篇文章
兰,杰,葛,杰,余,杰。et al。SARS-CoV-2与ACE2受体结合的刺突受体结合域的结构。自然581, 215 - 220(2020)。https://doi.org/10.1038/s41586-020-2180-5
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586-020-2180-5
这篇文章被引用
SARS-CoV-2刺突受体结合域的突变景观和硅结构模型揭示了病毒-宿主相互作用的关键分子决定因素
分子与细胞生物学(2022)
COVID-19疗法:我们是否看到实质性进展?
细胞与分子生物学通讯(2022)
血管紧张素转换酶2 (ACE2)基因变异在COVID-19感染中的作用:文献综述
埃及医学人类遗传学杂志(2022)
用于COVID-19治疗和SARS-CoV-2检测的单克隆抗体
生物医学科学杂志(2022)
疫苗之外:小分子和肽抗covid - 19武器库一瞥
生物医学科学杂志(2022)