摘要
了解肠道微生物组功能需要培养细菌进行实验验证和参考细菌基因组序列来解释宏基因组数据集并指导功能分析。我们提出了人类胃肠道细菌培养集(HBC),这是一套完整的737个全基因组测序的细菌分离物,代表人类胃肠道微生物群中发现的31个科的273个物种(105个新物种)。HBC使来自人类胃肠道微生物群的细菌基因组数量增加了37%。由此产生的全球人类胃肠道细菌基因组收集(HGG)在13490个鸟枪测序宏基因组样本中按丰度分类了83%的属,与人类微生物组计划(HMP)基因组收集相比,分类分类提高了61%,并实现了近50%序列的亚种级分类。胃肠道细菌参考序列资源的改进避免了对宏基因组从头组装的依赖,并实现了对人类胃肠道微生物群的精确和经济有效的鸟枪宏基因组分析。
主要
人类胃肠道拥有一个多样化和动态的微生物群落,直接影响人类生物学和健康1,2,3..这个复杂的生态系统以细菌为主,但也包括病毒、古生菌、真菌和其他真核生物。宏基因组测序是用于研究胃肠道微生物群和其他自然和建筑环境中的微生物群的主要方法1,2,4.扩增子测序,针对16s核糖体RNA (rRNA)基因,能够表征分类水平的细菌和古菌组成,并可以检测微生物群落的结构变化。然而,即使在同一物种的高度相关的菌株之间,生物学相关的表型差异也存在5这些菌株差异通常不能通过扩增子测序来区分。使用鸟枪宏基因组测序,评估任何微生物组的整个基因组内容并实现精确的分类学分类和准确的功能分配是可行的,但前提是可以解释宏基因组序列以揭示所有存在的物种和菌株1.
计算方法可以应用于从宏基因组样本中提取物种甚至亚种级别的信息1,2,3.,6,7,8,9,10;然而,这些方法从根本上受到深度序列覆盖的要求和无法区分密切相关的细菌类群的限制11.此外,与从纯培养物中产生的高质量参考基因组不同,从新宏基因组组合中获得的基因组可能是不完整的或可能代表嵌合物种种群12.这些因素限制了使用宏基因组来源的基因组进行高分辨率分类学分类和功能分析的准确性。有证据表明,许多人的胃肠道菌群中含有多种相同种类的细菌6这意味着迫切需要提高宏基因组分析的精度和准确性,以实现开发基于微生物组的治疗方法所需的功能验证13,14.
全面收集参考质量的细菌基因组,能够进行准确的、基于参考的宏基因组分析(RBMA),并实现微生物组细菌组成的物种、亚种和菌株级别的分类分类。大量的工作已经用于组装来自不同环境的细菌参考基因组15包括人类微生物组计划(HMP),该计划对来自18个人体部位的细菌分离物进行了测序16;然而,由于个体之间的多样性和以往培养方法的限制,大多数物种仍有待分离、存档和基因组测序。随着细菌培养方法的最新进展,现在有可能在实验室中从人类胃肠道中培养和纯化大多数细菌17,18,19,20..
除了基因组序列之外,获取存档的细菌分离物进行功能实验有助于从基于序列的相关研究过渡到预测细菌功能的致病表型验证13.我们报告了人类胃肠道细菌培养集(HBC)的编译和测序,其中包含来自人类胃肠道的分离株,应该能够进行精确的宏基因组分析,而不需要从头组装或超深度测序和实验验证。
结果
组装一种胃肠道细菌培养物
为了从人类胃肠道中收集全面的细菌分离物,我们从英国的20名成年人的粪便样本中培养和纯化细菌分离物(n= 8)和北美(n= 12)。总的来说,我们选择了超过10,000个细菌分离株,然后使用16个S rRNA基因测序进行分类。结合我们之前报道的234株胃肠道分离物17, 737个纯化和存档的分离株现在被纳入HBC。该标本来自放线菌门31科(53个基因组)的273种(105个新物种);拟杆菌门(143个基因组;厚壁菌门(496个基因组;203种)和变形菌门(45个基因组;14种)(补充表1).HBC中每个分离物的基因组序列是可用的。
我们将我们的HBC基因组与通过国家生物技术信息中心(NCBI)基因组数据库获得的617个公开的、高质量的人类胃肠道相关细菌基因组结合起来,生成了人类胃肠道微生物群基因组集(HGG;补充表1).值得注意的是,HGG基因组中53%的物种被存档在HBC中。许多目前在HBC中不存在但在HGG中存在的剩余物种包括梭杆菌门、变形杆菌门和增效菌门的成员,这些成员通常在发达国家的健康个体中不存在。这表明,需要从更多样化的健康捐赠者和受疾病影响的人群中进一步进行有针对性的培养,以详尽地存档人类胃肠道微生物群的细菌成分。
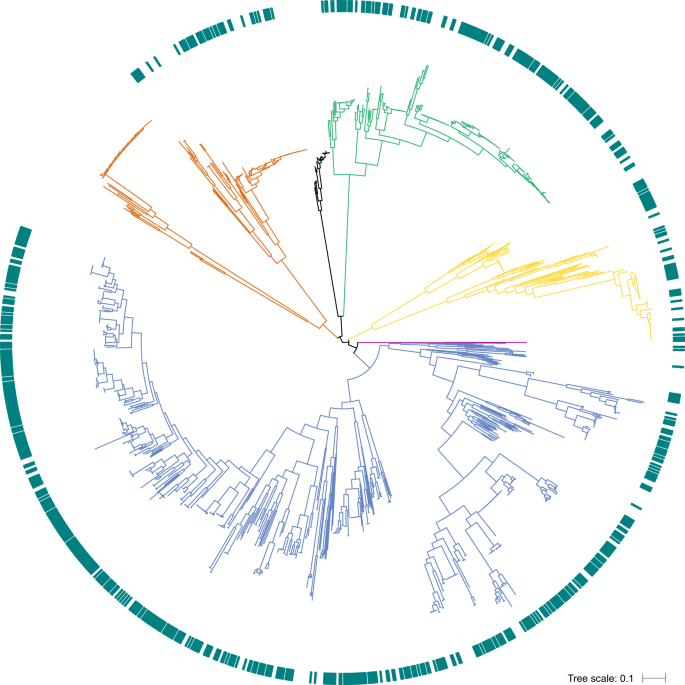
总的来说,HGG中的1354个基因组代表放线菌门57个科的530个物种(129个基因组;55种),拟杆菌门(231个基因组;69种),厚壁菌门(772个基因组;339种),Fusobacteria(26个基因组;9种),变形菌门(194个基因组;56种)和增效菌(2个基因组;2种)(补充图。1).为了了解这些类群之间的系统发育关系,我们提取了40个通用核心基因21并进行系统发育分析(图;1;补充图。2).总体而言,厚壁菌门(Firmicutes)的系统发育多样性最大,以Clostridia、丹毒纲(Erysipelotrichia)和Negativicutes最为明显;然而,广泛的物种和系统发育类群代表了所有门(图。1;补充图。2).
HGG改善胃肠宏基因组分析
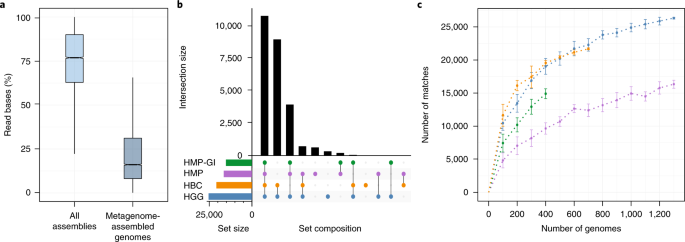
在缺乏参考基因组的情况下,最先进的宏基因组测序分析依赖于原始reads的从头组装,然后进行contig binning以生成宏基因组组装基因组序列(MAGs)。为了比较从头组装和装箱的分类学分类与RBMA分析的效率,我们考虑了13490个公共可访问的(补充表2)从粪便中提取鸟枪宏基因组,有足够的读覆盖来进行从头组装。从头组装和contig分箱鉴定出11,892个样本(88.2%),这些样本的质量足以产生长度大于或等于2,000个碱基对(bp)的contig。从9548个组件中获得了39913个完整性为>90%且污染<5%的容器,以下称为MAGs(补充表)3.).在这些mag中,81%至少有15个tRNAs,进一步强调了它们的高完整性;然而,只有16.1%(四分位范围,IQR = 31.2%-8.2%)的读基对这些mag有贡献(图2)。2).
一个,宏基因组样本中读碱基使用率占总读碱基的百分比(n= 13490),可以映射到它们各自的从头组装contigs(最小,22.23;Q1: 62.87;值,76.89;第三,89.99;max。,99.98) and metagenome-assembled genomes (MAGs; min., 0.16; Q1, 8.17; median, 16.09; Q3, 31.16; max., 65.64).b,使用HGG的分类箱总数(蓝色),仅从HBC收集的基因组(HBC;橙色)、HMP(紫色)和HMP胃肠道分离株(HMP- gi;绿色)。c使用HGG(蓝色)、HBC(橙色)、HMP(紫色)和HMP- gi(绿色)基因组的次样本集分类的39913个mas的总数。误差条显示平均值和s.d (n= 100个bootstrap)。
为了评估单独的HBC基因组集合和完整的HGG集合与现有的HMP基因组相比如何,我们接下来考虑使用每个集合作为参考数据库可以识别哪些MAGs。HGG能够识别25,085个mag,而仅与HBC对应的基因组只能识别20,772个mag。与此同时,从所有18个体点的HMP收集中鉴定出16,476个mas,如果仅包括来自胃肠道体点(HMP- gi)的HMP分离株,则发现15,156个mas。这表明,与完整的HMP相比,使用HGG集合作为参考时,提高了52.3%。2 b).由于HGG基因组集合比HBC、HMP和HMP- gi基因组集合大得多,我们接下来对每个基因组数据库进行了自举子采样,并将所选基因组与之前确定的mag基因组进行了平均核苷酸同一性(ANI > 95%)比较。考虑到400个基因组的子样本(HMP- gi可用的最后一个数据点),HGG达到19,545个匹配,HBC基因组收集19,036个匹配,相比之下,HMP- gi有14,906个匹配,完整HMP有9,655个匹配(图5)。2摄氏度).在完整的HMP中分类受到阻碍,因为它包括非胃肠道物种的基因组。值得注意的是,使用HGG和HBC基因组实现的更大匹配表明,在这些数据集中也存在更具有代表性的系统发育多样性。因此,我们的分析表明,与现有基因组相比,HGG的分类潜力增加了61.1%。
宏基因组中基于系统发育的基因组覆盖估计
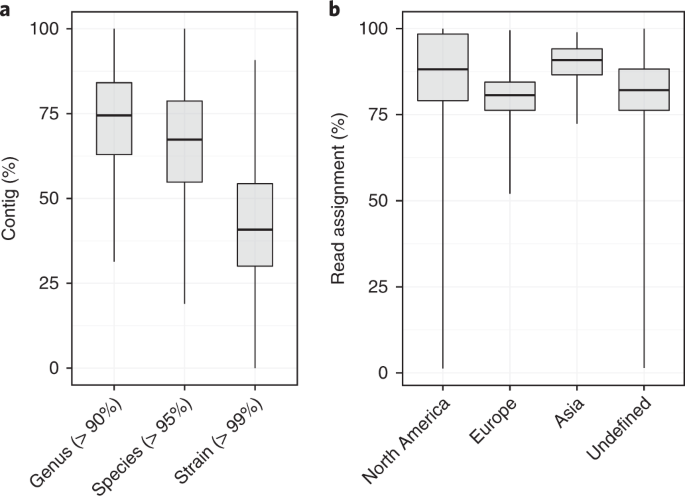
虽然可以使用从头组装和分箱方法生成mag,但该方法仍然无法分配本研究分析的13490个霰弹枪宏基因组测序样本中考虑的83.9%的reads。为了解决这一限制,我们接下来将所有从头组装的contigs与HGG进行了比较,以确定对更大比例的输入数据进行分类的能力。应用该方法,我们能够在大约相当于属的水平上绘制74.5% (IQR = 84.1%-62.9%)的contigs,而在种水平上绘制67.3% (IQR = 78.7%-54.8%)的contigs (95% cutoff;无花果。3).值得注意的是,40.8%(54.3% - 30.0%)可以归类为低于物种水平(99%的截点),尽管不包括HGG中从这些样本中培养的任何分离株(图2)。3).
一个,来自13490个属宏基因组样本的Contig赋值(90%;分钟,31.35;Q1, 62.92;值,74.48;第三,84.10;max。,100.0), species (95%; min., 18.94; Q1, 54.80; median, 67.35; Q3, 78.73; max., 100.0) and strain (99%; min., 0.0; Q1, 30.03; median, 40.82; Q3, 54.35; max., 90.77) identity compared to the HGG.b、北美宏基因组测序样本的分类(n= 2064;分钟,1.31;Q1, 79.07;值,88.16;第三,98.42;max。,99.97), Europe (n= 1431;分钟,52.07;Q1, 76.28;值,80.66;第三,84.47;max。,99.52), Asia (n= 191;分钟,72.37;Q1, 86.56;值,90.84;第三,94.13;max。,98.93.) and the other undefined locations (n= 9804;分钟,1.45;Q1, 76.28;值,82.14;第三,88.25;max。,99.94).
考虑到HGG提供的分类改进,我们接下来采用最低共同祖先RBMA来确定同一数据集的整体分类分类效率。与从头宏基因组组装和分箱方法相比,RBMA对低样本覆盖率更有弹性,因为它需要较浅的测序深度来自信地将序列分配到参考基因组。使用这些数据集,大规模霰弹枪宏基因组数据集的RBMA对每个样本的处理时间中位数为7.3分钟,而等效从头组装所需的时间为12.19小时。所需计算性能的大幅降低提供了一种处理更多样本的方法,并克服了阻碍许多宏基因组研究的统计能力的限制。
由于不正确的读取分配,细菌种群和群落中移动元件的水平基因转移会限制我们识别真实物种组成的能力。为了解决与水平基因转移相关的混杂因素,并提供更精确的分类学覆盖范围估计,我们还生成了在欧洲核苷酸档案(ENA)中发现的移动元件、插入序列和质粒的综合列表。22.结合HGG中预测的可移动元素,这代表了在人类胃肠道微生物群中发现的已知可移动元素的全面数据库。该数据库包括2803个质粒和489个转座子和插入序列,它们被隐藏在基因组中,并在最低共同祖先分类之前从宏基因组读取中过滤,以最大限度地提高系统发育信号(补充表4).当我们应用最低共同祖先RBMA和移动元素过滤后的HGG时,原始reads的分类在属水平上实现了82.9%的平均分类学分配,在种水平上实现了78.7%的平均分类学分配。综上所述,这些分析表明,即使考虑来自不同地理人群的样本,使用HGG也可以实现对来自人类胃肠道微生物群的大多数宏基因组reads的高分辨率分类(图2)。3 b).
人类胃肠道中的细菌多样性
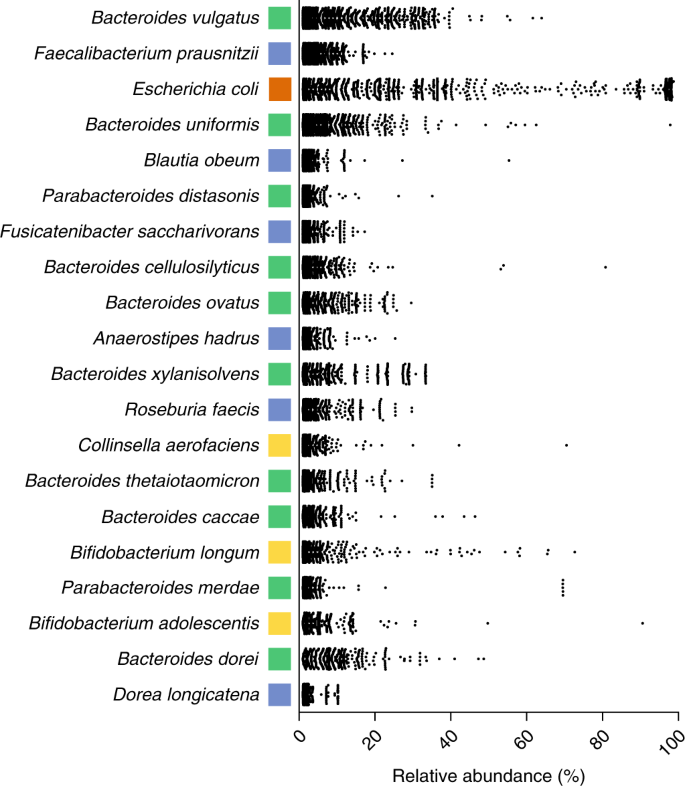
接下来,我们试图了解哪些物种在人类胃肠道微生物群中最普遍使用HGG。为了做到这一点,我们询问了所有13940个来自人类粪便的高质量鸟枪宏基因组样本(补充表2).尽管这种分析可能会受到粪便样本储存条件和DNA提取方法变化的影响23,我们推断,那些在许多个体的样本中高度普遍的物种可能在人类生物学中发挥重要作用,应该成为进一步研究的重点。仅考虑在任何样本中存在水平大于0.01%的物种,我们在两个以上不相关的样本中确定了165个物种(补充表)5).这组优势种包括拟杆菌门(n= 41)、厚壁菌门(n= 82),变形菌门(n= 27)和放线菌(n= 15)。考虑到每个门的背景流行率,这表明拟杆菌门(P< 0.05),厚壁菌门(Firmicutes)的物种数量明显不足(P< 0.01)。
考虑到所有在背景水平以上检测到的物种,大多数优势物种仍然是拟杆菌门的成员。总的来说,20个最流行的物种中有8个是该物种的成员拟杆菌属(拟杆菌vulgatus,拟杆菌均匀化,拟杆菌cellulosilyticus,拟杆菌ovatus,拟杆菌xylanisolvens,叫多形拟杆菌,拟杆菌caccae而且拟杆菌dorei).当对每个系统发育类群内的物种数量进行校正时,拟杆菌门一般为,而拟杆菌门为拟杆菌而且Parabacteroides属(Parabacteroides distasonis而且Parabacteroides merdae),更具体地说,明显过高(P< 0.001;无花果。4).尽管厚壁菌门中有三个超过346个物种,但只有6个远亲厚壁菌门物种在许多个体中都有很高的代表性(Fecalibacterium prausnitzii,Blautia obeum,Fusicatenibacter saccharivorans,Anaerostipes hadrus,Roseburia faecis而且Dorea longicatena;无花果。4).总的来说,所有在厚壁菌门内检测到的属在统计上的发生率不足。同样,变形菌门中唯一在样本中高度流行的成员是大肠杆菌,大多数变形菌门没有在样品中检测到。有趣的是,在考虑的检测水平上,没有发现梭杆菌或增殖菌的成员普遍存在,这表明它们只在本分析中不包括的某些条件或生命阶段被发现。
这些数据表明,拟杆菌在人类胃肠道中的特定成员具有潜在的关键作用。相比之下,在另一个优势门厚壁菌门(Firmicutes)中观察到的显著更大的多样性表明,这是一个高度可变的、潜在的功能冗余的类群,与先前关于该类群中许多类群动态孢子介导的传播和翻转的报道一致17,24.虽然通过这项研究鉴定的许多关键物种的基于实验室的表型分析仍然有限,但现在可以通过访问HBC中存档的分离株来解决这一问题。
考虑到HGG中包含的新基因组的多样性,我们接下来试图了解这些物种在整个群落中的患病率。重要的是,这些基因组的可用性使我们能够可靠地评估这些物种在宏基因组样本中的流行程度。总的来说,173个新基因组中的106个(60.9%)在13490个公共宏基因组样本中至少有一个样本的丰度大于0.001%。值得注意的是,近一半(87;48.6%),但不足四分之一(39;在> 1000份样品中检出21.8%)。有趣的是,在近一半的分析样本中发现了梭状芽孢杆菌中的三种新物种。在7797份(55.9%)和7074份(50.7%)样品中分别发现2个Lachnospiraceae新种,在6777份(48.6%)样品中发现1个Ruminococcaceae新种。总的来说,这些数据表明,通过这项工作确定的许多新物种和基因组经常出现在人类群体中,并可能代表人类胃肠道微生物群的组成部分,值得进一步研究。
人体胃肠道细菌的功能
这种扩展的基因组测序细菌分离物的收集能够进行高分辨率的功能和分类学分析。我们首先进行了一组同源蛋白群(COG)注释25对蛋白质序列进行分析,以确定HGG细菌中普遍存在的特征。该分析确定了4,696个不同的同源类群至少在一个分离物中。正如预期的那样,细菌的内务功能,包括核糖体蛋白功能、氨基酸合成和其他翻译相关功能,主导了收集中所有细菌的30种功能(补充表)6).
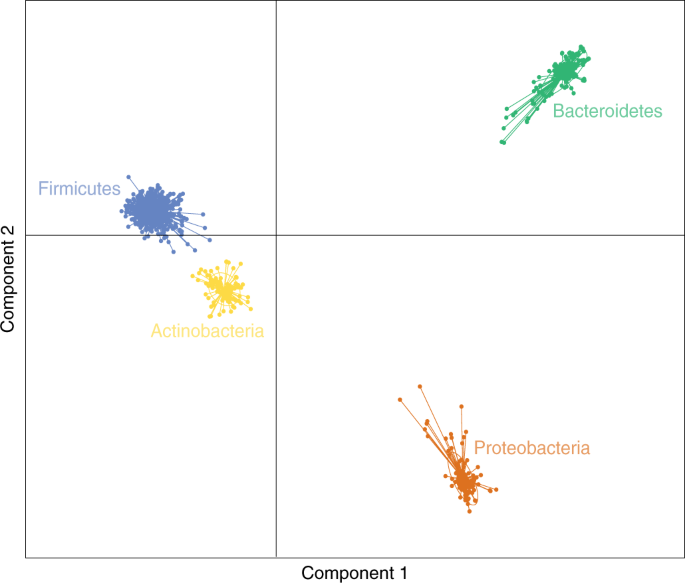
为了了解胃肠道菌群的四个主要细菌门(拟杆菌门、厚壁菌门、放线菌门和变形菌门)成员在功能作用上的差异,我们使用主成分鉴别分析(DAPC)比较了经COG分析鉴定的4,696个同源类群。这一比较表明,人类胃肠道微生物群的关键门之间存在明显的功能差异(图2)。5).接下来,我们进行了富集分析,以确定相对于HGG中存在的所有功能,在每个门中代表过多的功能。该分析确定了8、122、152和389个统计富集功能(问在放线菌门、拟杆菌门、厚壁菌门和变形菌门中分别< 0.001)(补充表7).放线菌的富集功能有限,但已确定的主要与脂质有关(问< 1.99 × 10−83)和碳水化合物代谢(问< 7.57 × 10−77).对拟杆菌门特定功能的等效分析确定了许多关键功能,包括铁(问< 1.18 × 10−114)和硫转运功能(问< 6.82 × 10−97)和特定的钠转运NADH泛醌氧化还原酶(问< 3.47 × 10−124).厚壁菌门以无特征功能为主;然而,孢子的形成(问< 3.48 × 10−123)和硫胺素(问< 2.76 × 10−101)和核黄素(问< 7.04 × 10−101)运输都非常丰富。变形菌门以果糖二磷酸酶(问< 4.50 × 10−140)、葡萄糖激酶(问< 4.55 × 10−125)和铁簇形成的调节因子(问< 9.20 × 10−98).这些结果表明,人类胃肠道微生物群的关键门所提供的独特功能存在明显差异;然而,尚未鉴定的功能的流行进一步证明了需要更好的基因组注释和功能基因组学来理解这些细菌。
HGG收集的基因组来自173个以前没有从人类胃肠道中分离出来的物种。这包括来自HBC内105个新物种的基因组,以及来自68个已知物种的基因组,其中以前不存在从人类胃肠道分离的基因组测序(补充表)1).为了了解在这173个物种中发现了什么功能,而在先前报道的基因组测序物种中不存在,我们进行了功能分析。共鉴定出45种新发现的功能,其中41种是在厚壁菌门中发现的。虽然这些功能主要由未鉴定的蛋白质主导,但新功能包括与四氢甲蝶呤相关的功能年代-甲基转移酶(存在于5个物种中),前蛋白转位酶(也存在于5个物种中)和甲烷发生所必需的甲醛激活酶(在4个以前未鉴定的厚壁菌门中发现)。此外,83.2%的新测序分离株和85.8%的新物种预计将在先前定义的基因组特征的基础上形成孢子17.
最后,我们试图了解哪些功能预计会出现在某个特定门的新基因组测序成员中,但在该门的所有现有基因组中都不存在。该分析确定了拟杆菌门中III、IV和VI型分泌系统成分,这些成分在之前测序的胃肠道拟杆菌门中没有发现,但在现有的变形菌门和厚壁菌门基因组中被识别出来。同样,在变形菌门现有基因组中发现的ABC转运蛋白功能在新测序的胃肠道厚壁菌门中也被发现,但在任何先前测序的分离物中都没有发现。这表明在微生物群落动态和宿主-微生物群相互作用中具有潜在重要的冗余作用的门的特定成员之间可能存在进一步的功能重叠。
讨论
我们提出了一个胃肠道细菌基因组和培养集,大大增加了在发达国家宏基因组学样本中发现的物种的比例。所使用的YCFA培养基达到了广泛代表原始样品的细菌生长水平,因此有必要将YCFA与选择性培养技术结合起来,以针对特定的细菌表型;例如,抗生素耐药性、产孢、碳水化合物利用和个别粪便样本中罕见细菌种类的分离。散弹枪宏基因组测序尚未对世界上许多人口进行,因此目前还不可能准确评估整个人群中培养细菌的比例。我们提出,需要开展一项扩大的、协调的全球培养活动,特别关注来自发展中国家和发达国家更多样化社区的样本和细菌分离物。收集和存储与这些宏基因组样本相关的元数据也是必不可少的:尽管努力制定标准26,27在美国,许多保存在公共序列集合中的基因组和宏基因组序列存在不正确、不一致、缺失或有限的元数据,这从根本上限制了它们的使用。
除了改进物种分类之外,获得全面的基因组测序分离株从根本上改变了功能分析的方法、分辨率和准确性。经基因组测序的分离株能够从参考基因组的遗传库中推断出功能能力。这消除了执行超深宏基因组测序的需要,并确保完整的功能通路包含在单个细菌中。除了提高准确性,该方法还能够提高功能分析的灵敏度,允许检测功能,尽管不普遍,但可能代表研究队列之间的根本差异。
尽管对病原体和模式生物的广泛描述主导了过去100年的微生物学研究,但对与人类健康相关的共生细菌的研究却落后了。如本文所述,培养、基因组测序和分离存档将为人类胃肠道和其他潜在位点的基于微生物群的分析提供实质性改进28.传统的微生物学方法可以继续获得急需的细菌分离物,以进行实验表征和验证,并提高我们对重要的人类相关微生物群落的理解。
方法
细菌培养和纯化
用补充的YCFA培养基进行细菌培养29有或没有乙醇预处理,如前所述17.简单地说,样品处理和培养是在37°C的Whitley DG250工作站的厌氧条件下进行的,使用磷酸盐缓冲盐水和在使用前在厌氧条件下孵育24小时的培养基。从20名健康成年人(北美、n= 12;英国,n= 8)在过去六个月内没有服用抗生素的患者。样品在培养前冷冻运输并保存在−80°C。将粪便样本在还原PBS中均质(0.1 g粪便/ ml PBS),连续稀释并直接镀于YCFA上29琼脂添加0.002 g ml−1将葡萄糖、麦芽糖和纤维素二糖分别放入直径13.5厘米的培养皿中。采集菌落,重划至纯度,并使用16s rRNA基因测序进行鉴定。16s rRNA基因序列同源性阈值为>97.8%30..这些分离品可从威康桑格研究所或相关的公共文化收藏中获得。
基因组测序与注释
基因组DNA是从球团细胞中提取的,使用苯酚-氯仿方法31.如前所述,在Wellcome Sanger研究所使用Illumina Hi-Seq平台制备和测序DNA,文库片段大小为200-300 bp,读取长度为100或125 bp32.使用前面描述的管道生成带注释的程序集33.对于每个示例,序列读取用于使用Velvet v1.2创建多个程序集。34)和VelvetOptimiser v2.2.5 (https://github.com/tseemann/VelvetOptimiser).装配改进步骤应用于N50最佳的装配,并使用SSPACE对contigs进行支架35和序列间隙填充使用GapFiller36.使用PROKKA v1.11执行自动注释。37).少于400个contigs的基因组、基因组大小小于8 Mb和存在同源性大于97.5%的16s rRNA序列被认为是纯的,并纳入进一步分析。我们收集的所有基因组都可以在项目访问下通过EBI欧洲核苷酸档案公开获取ERP105624而且ERP012217(补充表1).当NCBI中的分离源是粪便或胃肠道相关时,包括公共样本,并且序列来自纯分离物。所有基因组都按照内部基因组的描述进行了质量筛选,只有那些通过这些标准的基因组才被纳入进一步分析。
系统发育分析
通过提取40个通用核心标记基因的氨基酸序列进行系统发育分析38,39从每个基因组的细菌收集使用SpecI21.蛋白质序列与MAFFT v. 7.20(参考文献)进行连接和比对。40),使用RAxML v. 8.2.8构建极大似然树(参考文献)。41),采用标准LG模型,进行100次快速自举重复。使用FastTree可视化树42其次是iTOL43.
从头宏基因组分析
对于宏基因组分析,我们首先从欧洲核苷酸档案中可用的人类肠道样本中提取了13490个宏基因组测序(补充表)4).为了评估从头组装方法的效率,使用metaSPAdes v3.10.0(参考文献)组装原始读取。44),并随后与MetaBAT 2 (v2.12.1)绑定45,最小contig长度阈值为2000bp。通过使用BWA v0.7.16将原始读取映射回程序集或容器,可以推断测序覆盖率和读基使用情况。46),然后使用SAMtools v1.5检索映射的读基的百分比(参考。46)和MetaBAT 2中的jgi_summarize_bam_contig_depth函数(参考。45).使用INFERNAL v1.1.2检测核糖体rna (rrna)。47)使用细菌5 S、16 S和23 S rrna的Rfam协方差模型。总对齐长度由所有非重叠命中的总和推断出来。如果MAG中包含超过80%的序列,则认为每个基因存在。用tRNAscan-SE v2.0(参考文献)鉴定转移rna (tRNAs)。48)使用细菌tRNA模型和默认参数。根据CheckM的估计,>的完整性为90%,污染< 5%12进一步与人类微生物组计划(HMP)、HBC基因组和完整HGG进行分析。
基因组比较
使用完整的HGG集合、hbc衍生基因组和HMP基因组与mag进行比较。完整的HMP集,以及从HMP项目目录检索到的一组人体肠道特定参考文献(https://www.hmpdacc.org/hmp/catalog/grid.php?dataset=genomic),进行分析。对于每个数据库,Mash v2.0(参考。49)用于将所有参考基因组转换为默认设置的MinHash草图(mash草图)。然后,计算mag与每组参考之间的Mash距离,以找到最佳匹配(即具有最低Mash距离的基因组)。参考集中的每个MAG及其最近的相关物与MUMmer 3.23中的dnadiff v1.3(参考文献)对齐。50),以比较每对碱基与ANI对齐的百分比。mag长度的75%以上与ANI长度的95%以上被认为是正匹配。为了进一步对赋值性能进行基准测试,我们从每个数据库中以100个基因组的增量对参考基因组进行次采样,并创建了草图大小为100,000的MinHash草图(Mash sketch - 10万).然后,我们评估了与Mash距离低于0.05的每个下样本集相匹配的mag的数量(ANI > 95%)。使用UpSet R包对数据进行可视化51.
最低共同祖先宏基因组分析
最低共同祖先分析使用自定义生成的Kraken数据库,其中包含HBC中的所有基因组。宏基因组样本使用Trimmomatic 0.35进行质量筛选。52)和通过bowtie2映射到人类参考基因组(hg19)过滤的人类污染reads(参考文献)。53),过滤后的样本阅读数少于100万。如前所述,使用最低共同祖先分析在属和种水平上对过滤的序列进行分类54.
功能基因组分析
为了识别基因组中的蛋白质结构域,我们使用COG数据库进行了RPS-BLAST(访问2017年11月)25.利用COG数据库将所有蛋白结构域按不同功能分类25并进行主成分判别分析(DAPC)。55在R包Adegenet v2.0.1中实现。56).结构域和功能富集分析采用单边Fisher精确检验PR . 3.2.2中Hochberg方法调整的值。
报告总结
有关研究设计的进一步资料,请参阅自然研究报告摘要链接到这篇文章。
数据可用性
序列数据保存在ENA项目编号下ERP105624而且ERP012217.细菌分离物已存放在莱布尼茨研究所dsmz -德国微生物和细胞培养集(http://www.dsmz.de)、瑞典哥德堡大学文化馆藏(http://www.ccug.se)、由根特大学微生物实验室(BCCM/LMG)主办的比利时协同微生物收集(http://bccm.belspo.be/)和日本微生物收藏(JCM;http://jcm.brc.riken.jp/en/).每个基因组的培养集合标识符和ENA登录号在补充表中提供1.宏基因组组装的基因组可从ftp://ftp.ebi.ac.uk/pub/databases/metagenomics/hgg_mags.tar.gz.
参考文献
劳埃德-普莱斯,J.等人。扩展的人类微生物组计划中的菌株、功能和动态。自然550, 61-66(2017)。
秦,等。宏基因组测序建立的人体肠道微生物基因目录。自然464, 59-65(2010)。
肖尔茨等人。来自鸟枪宏基因组学的菌株水平微生物流行病学和种群基因组学。Nat方法。13, 435-438(2016)。
帕克斯,D. H.等。近8000个宏基因组组装基因组的恢复大大扩展了生命之树。Microbiol Nat。2, 1533-1542(2017)。
Kamada, N., Chen, g.y., Inohara, N. & Núñez, G.肠道微生物群对病原体和病原菌的控制。Immunol Nat。14, 685-690(2013)。
李,s.s.等。粪便菌群移植后供体和受体菌株的持久共存。科学352, 586-589(2016)。
Truong, D. T., Tett, A., Pasolli, E., Huttenhower, C. & Segata, N.微生物菌株水平的种群结构和宏基因组的遗传多样性。基因组Res。27, 626-638(2017)。
尼尔森,H. B.等人。在不使用参考基因组的情况下鉴定和组装复杂宏基因组样本中的基因组和遗传元件。生物科技Nat。》。32, 822-828(2014)。
Truong, D. T.等。用于增强宏基因组分类分析的MetaPhlAn2。Nat方法。12, 902-903(2015)。
Nayfach, S., Rodriguez-Mueller, B., Garud, N. & Pollard, K. S.一个用于菌株分析的集成宏基因组学管道揭示了细菌传播和生物地理学的新模式。基因组Res。26, 1612-1625(2016)。
昆斯,C.,沃克,A. W.,辛普森,J. T.,洛曼,N. J. &塞格塔,N.散弹宏基因组学,从取样到分析。生物科技Nat。》。35, 833-844(2017)。
Parks, D. H., Imelfort, M., Skennerton, C. T., Hugenholtz, P. & Tyson, g.w. CheckM:评估从分离物、单细胞和宏基因组中恢复的微生物基因组的质量。基因组Res。25, 1043-1055(2015)。
内维尔,B. A.福斯特,S. C.和劳利,T. D.科赫的假设:在人类微生物研究中建立因果关系。咕咕叫。当今。Microbiol。42, 47-52(2018)。
沃克,A. W.,邓肯,S. H.,路易斯,P. &弗林特,H. J.人类肠道微生物群的系统发育、培养和宏基因组学。Microbiol趋势。22, 267-274(2014)。
Mukherjee, S.等人1003个细菌和古细菌分离株的参考基因组扩大了生命树的覆盖范围。生物科技Nat。》。35, 676-683(2017)。
人类微生物组项目联盟。健康人体微生物群的结构、功能和多样性。自然486, 207-214(2012)。
布朗,h.p.等人。“不可培养的”人类微生物群的培养揭示了新的分类群和广泛的孢子。自然533, 543-546(2016)。
拉格尔,J. C.等。用培养组学方法培养以前未培养的人体肠道菌群成员。Microbiol Nat。1, 16203(2016)。
古德曼,a.l.等。广泛的个人人类肠道菌群培养集合的特征和操作的gnotobiomice。国家科学院学报美国108, 6252-6257(2011)。
刘,J. T.等。通过培养富集分子图谱捕获人类肠道微生物群的多样性。基因组医学。8, 72(2016)。
孟德,苏娜川,S.,泽勒,G. &博克,P.原核生物物种的精确和普遍描述。Nat方法。10, 881-884(2013)。
西尔维斯特,N.等人。2017年欧洲核苷酸档案。核酸测定。46, d36-d40(2017)。
Costea, p.i.等。朝着宏基因组研究中人类粪便样本处理的标准迈进。生物科技Nat。》。35, 1069-1076(2017)。
布朗,H. P.,内维尔,B. A.,福斯特,S. C.和劳利,T. D.肠道微生物群的传播:健康的传播。Nat. Rev.微生物。15, 531-543(2017)。
Tatusov, R. L., Galperin, M. Y., Natale, D. a . & Koonin, E. V. COG数据库:用于蛋白质功能和进化基因组规模分析的工具。核酸测定。28, 33-36(2000)。
伊尔马兹,P.等人。关于标记基因序列(MIMARKS)的最小信息和关于任何(x)序列(MIxS)规格的最小信息。生物科技Nat。》。29, 415-420(2011)。
鲍尔斯,r.m.等。细菌和古生菌的单个放大基因组(MISAG)和宏基因组组装基因组(MIMAG)的最少信息。生物科技Nat。》。35, 725-731(2017)。
托马斯-怀特,K.等人。女性膀胱细菌的培养揭示了一个相互关联的泌尿生殖系统微生物群。Commun Nat。9, 1557(2018)。
Duncan, s.h., Hold, g.l., hamsen, h.j., Stewart, c.s. & Flint, h.j.梭菌属prausnitzii,并建议将其重新归类为Faecalibacterium prausnitziigen nov.,梳子。11月。Int。j .系统。另一个星球。Microbiol。52, 2141-2146(2002)。
Yarza, P.等人。结合16S rRNA基因序列对培养菌和未培养菌及古菌进行分类。Nat. Rev.微生物。12, 635-645(2014)。
格林,m.r.,桑布鲁克,J. &桑布鲁克,J.。分子克隆:实验室手册.第4版(冷泉港实验室出版社,2012)。
哈里斯,s.r.等人。MRSA在医院传播和洲际传播期间的演变。科学327, 469-474(2010)。
佩奇,A. J.等人。Illumina数据的高通量原核细胞组装和改进管道。活细胞。染色体组。2, e000083(2016)。
Zerbino, D. R. & Birney, E. Velvet:使用de Bruijn图的从头短读组装算法。基因组Res。18, 821-829(2008)。
Boetzer, M, Henkel, c.v, Jansen, H. J, Butler, D. & Pirovano, W.使用SSPACE搭建预组装的contigs。生物信息学27, 578-579(2011)。
Boetzer, M. & Pirovano, W.使用GapFiller接近封闭基因组。基因组医学杂志。13, r56(2012)。
Seemann, T. Prokka:快速原核基因组注释。生物信息学30., 2068-2069(2014)。
Ciccarelli, f.d.等人。实现高分辨率生命树的自动重建。科学311, 1283-1287(2006)。
Sorek, R.等人。水平基因转移障碍的全基因组实验测定。科学318, 1449-1452(2007)。
Katoh, K. & Standley, D. M. MAFFT多序列比对软件版本7:性能和可用性的改进。摩尔。杂志。另一个星球。30., 772-780(2013)。
Stamatakis, a . RAxML版本8:用于系统发育分析和大系统发育后分析的工具。生物信息学30., 1312-1313(2014)。
Price, M. N. Dehal, P. S. & Arkin, a . P. FastTree:用剖面代替距离矩阵计算大型最小进化树。摩尔。杂志。另一个星球。26, 1641-1650(2009)。
Letunic, I. & Bork, P.交互式生命树2:使系统发育树的在线注释和显示变得容易。核酸测定。39, w475-w478(2011)。
Nurk, S., Meleshko, D., Korobeynikov, a . & Pevzner, P. a . metaSPAdes:一种新的多功能宏基因组组装器。基因组Res。27, 824-834(2017)。
Kang, d.d., Froula, J., Egan, R. & Wang, Z. MetaBAT,一种从复杂微生物群落中精确重建单基因组的有效工具。PeerJ3., e1165(2015)。
李,H. & Durbin R.快速和准确的长读对齐与Burrows-Wheeler变换。生物信息学26, 589-595(2010)。
Nawrocki, e.p.和Eddy, s.r. Infernal 1.1: 100倍快的RNA同源性搜索。生物信息学29, 2933-2935(2013)。
Lowe, t.m.和Chan, P. P. tRNAscan-SE在线:整合搜索和上下文分析转移RNA基因。核酸测定。44, w54-w57(2016)。
Ondov, B. D.等人。Mash:使用MinHash快速估计基因组和宏基因组距离。基因组医学杂志。17132(2016)。
库尔茨,S.等人。通用和开放的软件比较大的基因组。基因组医学杂志。5, r12(2004)。
Lex, A., Gehlenborg, N., Strobelt, H., Vuillemot, R. & Pfister, H. UpSet:相交集的可视化。IEEE反式。粘度第一版。图。20., 1983-1992(2014)。
Bolger, a.m., Lohse, M. & Usadel, B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学30., 2114-2120(2014)。
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。Nat方法。9, 357-359(2012)。
福斯特,s.c.等。HPMCD:基于宏基因组数据集和微生物参考基因组的人类微生物群落数据库。核酸测定。44, d604-d609(2016)。
Jombart, T., Devillard, S. & Balloux, F.主成分判别分析:一种分析遗传结构群体的新方法。BMC麝猫。11, 94(2010)。
Jombart, T. adegenet:用于遗传标记的多元分析的R包。生物信息学24, 1403-1405(2008)。
确认
这项工作得到了威康基金会(098051)的支持;英国医学研究委员会(PF451 to T.L.)、英国生物科学研究委员会(BB/M011755/1 to R.D.F.)、欧洲分子生物学实验室、欧洲生物信息学研究所(EMBL-EBI)和澳大利亚国家卫生和医学研究委员会(1141564 to S.C.F.)。S.C.F.由澳大利亚国家卫生和医学研究委员会(1091097)和维多利亚州政府的业务基础设施支持计划支持。博士学位由医学研究委员会博士训练补助金(MR/J004111/1)资助。作者还想感谢威康桑格研究所病原信息学和核心测序团队的支持。
作者信息
作者及隶属关系
贡献
s.c.f., B.A.N, n.k., R.D.F.和T.D.L.构思了这项研究。同上,S.C.F, N.K B.A.N, H.P.B,汽车,R.D.F. T.D.L.和写的手稿。s.c.f., B.A.N, B.O.A, e.v., m.d.s., m.d., h.p.b., y.s., L.J.P.和T.L.收集样品,纯化细菌并进行基因组测序。s.c.f., n.k., a.a., a.l.m., t.t.m., A.Z.和R.D.F.进行了计算分析。所有作者都阅读并批准了手稿。
相应的作者
道德声明
相互竞争的利益
s.c.f., b.a.n., m.d., R.D.F.和T.D.L.是Microbiotica Pty Ltd.的员工或顾问。
额外的信息
出版商的注意:施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
综合补充信息
补充图1 HGG内物种和基因组序列计数。
每种放线菌的种类和基因组序列的计数(n= 129个基因组,55个物种),拟杆菌门(n= 231个基因组,69个物种),厚壁菌门(n= 772个基因组,339种),梭杆菌门(n= 26个基因组,9个物种),变形菌门(n= 194个基因组,56个物种)和增效菌(n= 2个基因组,2个物种)。
补充图2人类胃肠道微生物群基因组集的系统发育多样性。
最大似然树,包括物种名称,由来自737个HBC基因组(绿色外圈)的40个通用核心基因和来自人类胃肠道样本的617个高质量公共基因组生成,它们共同构成HGG。分支颜色区分了放线菌门(金色)、拟杆菌门(绿色)、厚壁菌门(蓝色)、梭杆菌门(棕色)、协同菌门(黑色)和变形菌门(红色)。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
福斯特,南卡罗来纳,库马尔,北卡罗来纳,阿诺耶,B.O.et al。用于改进宏基因组分析的人类肠道细菌基因组和培养集。生物科技Nat》37, 186-192(2019)。https://doi.org/10.1038/s41587-018-0009-7
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41587-018-0009-7
这篇文章被引用
微生物参考基因组的性状偏倚
科学数据(2023)
7302个人类微生物基因组代谢重建,用于个性化医疗
自然生物技术(2023)
CKD中的肠道微生物组研究:机会、缺陷和治疗潜力
自然肾科学评论(2023)
肠道微生物群和高血压
自然肾科学评论(2023)
宿主介导的基因工程和基于微生物群的可持续农业和环境技术优化
功能与整合基因组学(2023)