文摘gydF4y2Ba
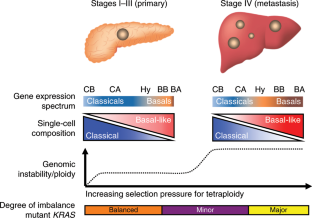
胰腺腺癌表现为光谱的高攻击性疾病患者。这种疾病异质性的基础已经被证明很难解决由于肿瘤细胞性差和广泛的基因组不稳定性。为了解决这个问题,整个基因组和转录组的数据集从纯化的上皮主要生成和转移性肿瘤。转录组分析表明分子亚型的基因表达连续驱动的混合瘤内亚种群,这证实了单细胞分析。集成的全基因组分析发现,分子亚型都与特定的拷贝数畸变等基因突变gydF4y2Ba喀斯特gydF4y2Ba和gydF4y2BaGATA6gydF4y2Ba。通过映射肿瘤遗传历史,tetraploidization成为这些事件背后的关键突变过程。综上所述,这些数据支持的前提下基因畸变的星座肿瘤产生的分子亚型,这疾病异质性是由于持续的基因组不稳定性在进展。gydF4y2Ba
这是一个预览的订阅内容,gydF4y2Ba通过访问你的机构gydF4y2Ba
相关的文章gydF4y2Ba
开放获取文章引用这篇文章。gydF4y2Ba
Pacpaint: histology-based深学习模型揭示胰腺腺癌的广泛intratumor分子异质性gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2023年6月13日gydF4y2Ba
磷脂运输车PITPNC1链接喀斯特MYC预防自噬在肺癌和胰腺癌gydF4y2Ba
分子癌症gydF4y2Ba开放获取gydF4y2Ba2023年5月20gydF4y2Ba
Irbesartan克服耐吉西他滨在胰腺癌中通过抑制具备干细胞通过抑制和铁代谢的河马/ YAP1 c-Jun轴gydF4y2Ba
实验和临床癌症研究杂志》上gydF4y2Ba开放获取gydF4y2Ba2023年5月04gydF4y2Ba
访问选项gydF4y2Ba
访问其他自然组合期刊性质和54gydF4y2Ba
得到自然+,请求书在线访问订阅gydF4y2Ba
29.99美元gydF4y2Ba/ 30天gydF4y2Ba
取消任何时候gydF4y2Ba
订阅本杂志gydF4y2Ba
收到12印刷问题和网络访问gydF4y2Ba
每年189.00美元gydF4y2Ba
只有15.75美元的问题gydF4y2Ba
本文租或购买gydF4y2Ba
价格不同的文章类型gydF4y2Ba
从gydF4y2Ba1.95美元gydF4y2Ba
来gydF4y2Ba39.95美元gydF4y2Ba
价格可能受当地税收计算在结帐gydF4y2Ba
数据可用性gydF4y2Ba
原始数据是免费的从EGA加入以下代码可用gydF4y2BaEGAS00001002543gydF4y2Ba。gydF4y2Ba
代码的可用性gydF4y2Ba
没有独特的代码是在这项研究中开发的。R表示用于相关的脚本或功能gydF4y2Ba结果gydF4y2Ba,图传说或gydF4y2Ba方法gydF4y2Ba。gydF4y2Ba
改变历史gydF4y2Ba
2020年2月12日gydF4y2Ba
这篇论文已经发表的校正:gydF4y2Bahttps://doi.org/10.1038/s41588 - 020 - 0588 - 3gydF4y2Ba
引用gydF4y2Ba
Waddell: et al。整个基因组重新定义胰腺癌的突变景观。gydF4y2Ba自然gydF4y2Ba518年gydF4y2Ba,495 - 501 (2015)。gydF4y2Ba
勒,d . t . et al . PD-1封锁在错配修复缺陷的肿瘤中。gydF4y2Ba心血管病。j .地中海。gydF4y2Ba372年gydF4y2Ba,2509 - 2520 (2015)。gydF4y2Ba
Yu, J。,Blackford, A. L., Dal Molin, M., Wolfgang, C. L. & Goggins, M. Time to progression of pancreatic ductal adenocarcinoma from low-to-high tumour stages.肠道gydF4y2Ba64年gydF4y2Ba,1783 - 1789 (2015)。gydF4y2Ba
Hruban, r . H。郭金,M。,Parsons, J. & Kern, S. E. Progression model for pancreatic cancer.中国。癌症Res。gydF4y2Ba6gydF4y2Ba,2969 - 2972 (2000)。gydF4y2Ba
琼斯,s . et al .核心信号通路在人类胰腺癌揭示了全球基因组分析。gydF4y2Ba科学gydF4y2Ba321年gydF4y2Ba,1801 - 1806 (2008)。gydF4y2Ba
Waddell: et al .胰腺癌基因揭示畸变在轴突引导途径的基因。gydF4y2Ba自然gydF4y2Ba491年gydF4y2Ba,399 - 405 (2012)。gydF4y2Ba
Witkiewicz, a . k . et al . Whole-exome测序定义遗传多样性和罹患胰腺癌的治疗目标。gydF4y2BaCommun Nat。gydF4y2Ba6gydF4y2Ba6744 (2015)。gydF4y2Ba
Makohon-Moore, a . p . et al。有限的已知驱动基因突变的异质性在胰腺癌患者的转移。gydF4y2BaNat,麝猫。gydF4y2Ba49gydF4y2Ba,358 - 366 (2017)。gydF4y2Ba
康纳,a . a . et al .整合基因组和转录特性在胰腺癌中揭示了细胞周期进展增加转移。gydF4y2Ba癌症细胞gydF4y2Ba35gydF4y2BaP267-P282 (2019)。gydF4y2Ba
麦当劳,o . g . et al .外遗传性改变在胰腺癌进展联系合成葡萄糖代谢远处转移。gydF4y2BaNat,麝猫。gydF4y2Ba49gydF4y2Ba,367 - 376 (2017)。gydF4y2Ba
Notta, f . et al .新一轮模型基于基因组重排的胰腺癌进化模式。gydF4y2Ba自然gydF4y2Ba538年gydF4y2Ba,378 - 382 (2016)。gydF4y2Ba
Cortes-Ciriano, et al . 2658年chromothripsis综合分析使用全基因组测序人类癌症。预印在gydF4y2BabioRxivgydF4y2Bahttps://doi.org/10.1101/333617gydF4y2Ba(2018)。gydF4y2Ba
拉斐尔,b . j . et al .集成化的胰腺导管腺癌基因特征。gydF4y2Ba癌症细胞gydF4y2Ba32gydF4y2Ba,185 - 203。e13(2017)。gydF4y2Ba
莫菲特,r . a . et al。虚拟显微解剖标识不同的肿瘤,stroma-specific胰腺导管腺癌的亚型。gydF4y2BaNat,麝猫。gydF4y2Ba47gydF4y2Ba,1168 - 1178 (2015)。gydF4y2Ba
Collisson, e·a . et al .胰腺导管腺癌的亚型和对治疗的反应不同。gydF4y2BaNat,地中海。gydF4y2Ba17gydF4y2Ba,500 - 503 (2011)。gydF4y2Ba
贝利,p . et al .基因组分析识别分子亚型的胰腺癌。gydF4y2Ba自然gydF4y2Ba531年gydF4y2Ba47-52 (2016)。gydF4y2Ba
威尔克森,m . d . &海耶斯,d . n . ConsensusClusterPlus:一个类发现工具有信心评估和项目跟踪。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba,1572 - 1573 (2010)。gydF4y2Ba
Puram, s . v . et al .单细胞转录组分析,主要在头部和颈部癌症和转移性肿瘤的生态系统。gydF4y2Ba细胞gydF4y2Ba171年gydF4y2Ba,1611 - 1624。e24 (2017)。gydF4y2Ba
Muraro, m . j . et al。人类胰腺的单细胞转录组图集。gydF4y2Ba细胞系统。gydF4y2Ba3gydF4y2Ba,385 - 394。e3 (2016)。gydF4y2Ba
Martinelli, p . et al . Gata6is所需完整的外分泌胰腺腺泡的分化和维护在成年老鼠。gydF4y2Ba肠道gydF4y2Ba62年gydF4y2Ba,1481 - 1488 (2013)。gydF4y2Ba
穆勒,et al。进化路线和喀斯特剂量定义胰腺癌表型。gydF4y2Ba自然gydF4y2Ba554年gydF4y2Ba62 (2018)。gydF4y2Ba
扎克,t . i . et al . Pan-cancer体细胞拷贝数改变的模式。gydF4y2BaNat,麝猫。gydF4y2Ba45gydF4y2Ba,1134 - 1140 (2013)。gydF4y2Ba
Krasinskas, a . M。莫泽,a·J。萨卡人,B。,Adsay, N. V. & Chiosea, S. I. KRAS mutant allele-specific imbalance is associated with worse prognosis in pancreatic cancer and progression to undifferentiated carcinoma of the pancreas.国防部,病理学研究。gydF4y2Ba26gydF4y2Ba,1346 - 1354 (2013)。gydF4y2Ba
坎贝尔,p . j . et al .基因组不稳定性的模式和动态转移性胰腺癌。gydF4y2Ba自然gydF4y2Ba467年gydF4y2Ba,1109 - 1113 (2010)。gydF4y2Ba
昂,k l . et al . Genomics-driven精密医学先进的胰腺癌:从指南针试验初步结果。gydF4y2Ba中国。癌症Res。gydF4y2Ba24gydF4y2Bahttps://doi.org/10.1158/1078 - 0432. - ccr - 17 - 2994gydF4y2Ba(2017)。gydF4y2Ba
Cejalvo, j . m . et al .内在亚型和基因表达谱原发性和转移性乳腺癌。gydF4y2Ba癌症Res。gydF4y2Ba77年gydF4y2Ba,2213 - 2221 (2017)。gydF4y2Ba
辛格,A . et al .基因表达特征与“k - ras基因成瘾”揭示了相关监管机构EMT和肿瘤细胞的生存。gydF4y2Ba癌症细胞gydF4y2Ba15gydF4y2Ba,489 - 500 (2009)。gydF4y2Ba
央行,s . f . et al .瀑样模型的人类和小鼠导管胰腺癌。gydF4y2Ba细胞gydF4y2Ba160年gydF4y2Ba,324 - 338 (2015)。gydF4y2Ba
克尔,e . M。Gaude E。,Turrell, F. K., Frezza, C. & Martins, C. P. Mutant喀斯特gydF4y2Ba拷贝数定义代谢重编程和治疗脆弱的感情。gydF4y2Ba自然gydF4y2Ba531年gydF4y2Ba,110 - 113 (2016)。gydF4y2Ba
Ligorio, m . et al .基质微环境的形状的瘤内架构胰腺癌。gydF4y2Ba细胞gydF4y2Ba178年gydF4y2Ba,160 - 175。e27(2019).
Aiello: m . et al . EMT亚型影响上皮细胞可塑性和模式的细胞迁移。gydF4y2BaDev细胞。gydF4y2Ba45gydF4y2Ba681 (2018)。gydF4y2Ba
罗伊,js。et al。增强重组促进胰腺癌转移。gydF4y2Ba细胞gydF4y2Ba170年gydF4y2Ba,875 - 888。e20.(2017)。gydF4y2Ba
Puleo, f . et al .分层的胰腺导管腺癌肿瘤微环境特性。gydF4y2Ba胃肠病学gydF4y2Ba155年gydF4y2Ba,1999 - 2013。e3 (2018)。gydF4y2Ba
康纳,a . a . et al .协会截然不同的突变特征相关的增加胰腺导管腺癌的免疫活动。gydF4y2BaJAMA杂志。gydF4y2Ba3gydF4y2Ba,774 - 783 (2017)。gydF4y2Ba
布雷:L。,Pimentel, H., Melsted, P. & Pachter, L. Near-optimal probabilistic RNA-seq quantification.生物科技Nat。》。gydF4y2Ba34gydF4y2Ba,525 - 527 (2016)。gydF4y2Ba
Soneson C。,爱,我。&Robinson, M. D. Differential analyses for RNA-seq: transcript-level estimates improve gene-level inferences.F1000Res。gydF4y2Ba4gydF4y2Ba1521 (2015)。gydF4y2Ba
爱,我。,Huber, W. & Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2.基因组医学杂志。gydF4y2Ba15gydF4y2Ba,550 - 21 (2014)。gydF4y2Ba
f . et al . Scikit-learn Pedregosa:机器学习在Python中。gydF4y2Baj·马赫。学习。Res。gydF4y2Ba12gydF4y2Ba,2825 - 2830 (2011)。gydF4y2Ba
萨勃拉曼尼亚,等。基因集富集分析:一个基于知识的方法解释全基因组表达谱。gydF4y2BaProc。《科学。美国gydF4y2Ba102年gydF4y2Ba,15545 - 15550 (2005)。gydF4y2Ba
Tiriac, h . et al .瀑样分析识别常见的急救员在胰腺癌化疗。gydF4y2Ba癌症。gydF4y2Ba8gydF4y2Ba,1112 - 1129 (2018)。gydF4y2Ba
斯图亚特,t . et al .单细胞数据综合集成。gydF4y2Ba细胞gydF4y2Ba177年gydF4y2Ba,1888 - 1902。e21 (2019)。gydF4y2Ba
麦克因尼斯,l·希利& j . UMAP:统一歧管近似和投影降维。预印在gydF4y2Bahttps://arxiv.org/abs/1802.03426gydF4y2Ba(2018)。gydF4y2Ba
李,h &杜宾,r .快速和准确的短阅读符合burrows - wheeler变换。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba,1754 - 1760 (2009)。gydF4y2Ba
麦肯纳,a . et al .基因组分析工具包:一个MapReduce框架分析下一代DNA测序数据。gydF4y2Ba基因组Res。gydF4y2Ba20.gydF4y2Ba,1297 - 1303 (2010)。gydF4y2Ba
桑德斯,c . t . et al . Strelka:准确的体细胞small-variant打来的测序tumor-normal样本对。gydF4y2Ba生物信息学gydF4y2Ba28gydF4y2Ba,1811 - 1817 (2012)。gydF4y2Ba
Cibulskis, k . et al .敏感体点突变的检测不洁净和异构癌症样本。gydF4y2Ba生物科技Nat。》。gydF4y2Ba31日gydF4y2Ba,213 - 219 (2013)。gydF4y2Ba
王,j . et al .波峰地图体细胞在癌症基因组结构变异碱基对决议。gydF4y2BaNat方法。gydF4y2Ba8gydF4y2Ba,652 - 654 (2011)。gydF4y2Ba
Rausch t . et al . del:通过集成paired-end和split-read分析发现结构变体。gydF4y2Ba生物信息学gydF4y2Ba28gydF4y2Bai333-i339 (2012)。gydF4y2Ba
Lawrence m . s . et al .异质性突变在癌症和寻找新的癌症相关的基因。gydF4y2Ba自然gydF4y2Ba499年gydF4y2Ba,214 - 218 (2013)。gydF4y2Ba
雷瑟尔森·m·d·m . et al . Pan-cancer网络分析确定组合的罕见的体细胞突变途径和蛋白质复合物。gydF4y2BaNat,麝猫。gydF4y2Ba47gydF4y2Ba,106 - 114 (2014)。gydF4y2Ba
Scrucca, L。Fop, M。,Murphy, T. B. & Raftery, A. E. mclust 5: clustering, classification and density estimation using Gaussian finite mixture models.r . J。gydF4y2Ba8gydF4y2Ba,289 - 317 (2016)。gydF4y2Ba
确认gydF4y2Ba
我们感谢Notta实验室的所有成员和PanCuRx项目安大略癌症研究所(OICR)手稿的评论,和OICR基因技术项目技术支持。我们还要感谢f .真实和b·斯坦格对知识输入和批判性讨论关于手稿。我们也承认病人和他们的家属的参与,和波格游戏和胚芽团队。本研究进行的支持下OICR (PanCuRx转化研究计划)通过安大略省政府提供的资金,华莱士麦凯恩胰腺癌中心的玛格丽特公主癌症基金会的支持下,特里福克斯研究所,加拿大癌症协会研究所和加拿大胰腺癌基础。研究也支持了慈善捐款的加拿大朋友希伯来大学(a, Soyka)。信号发生器是一名调查员的接受者从OICR奖。G.Z.的临床研究学者魁北克矫揉造作的en健康基础。支持F.N. Gattuso-Slaight个性化癌症医学基金会,资助OICR,加拿大卫生研究院的研究(没有。388785)和癌症研究协会(没有。23383)。 G.Z. is a Clinician-Scientist of the Fonds de la Recherche en Santé du Québec. P.J.C. is a Wellcome Trust Senior Clinical Fellow and L.D.S. and S.G. are recipients of Senior or Clinician-Scientist Awards from OICR.
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
数据分析和解释被M.C.-S.-Y完成。,J.C.K.,S.G. and F.N. Samples were processed by K.N., E.F.F., J.M., J.M.W., S.-B.L. and D.C. M.C.-S.-Y., J.C.K., G.W.W., K.N., R.E.D., G.H.J., A.Z., P.M.K., V.S., B.H.-K. and L.D.S. were responsible for genomics. S.-B.L., D.C., I.L. and J.M.S.B. were responsible for LCM. Single-cell RNA-seq was done by M.C.-S.-Y. and K.N. S.K., R.C. and S.E.F. were responsible for pathology. Sample acquisition and clinical annotation were done by G.M.O., A.A.C., A.D., R.C.G., J.M.W., A.B., G.Z., J.J.K. and S.G. Data were validated by H.T., F.E.M.F., J.M.K., J.T.T., D.J.R., D.F.S., S.J.M.J, M.A.M., J.L. and D.A.T. The manuscript was written by M.C.-S.-Y., J.C.K. and F.N. The manuscript was edited by E.F.F., H.T., F.E.M.F., P.J.C. and D.A.T. The project was led and supervised by S.G. and F.N.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然保持中立在发表关于司法主权地图和所属机构。gydF4y2Ba
扩展数据gydF4y2Ba
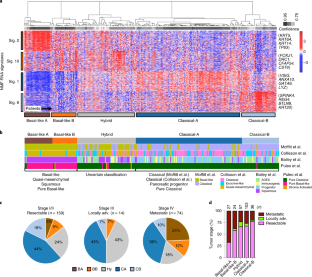
扩展数据图1生存和肿瘤可切除的响应数据,根据以往的子类型化先进的疾病的计划。gydF4y2Ba
一个gydF4y2Ba,为可切除的生存分析(左列)和先进的基于先前莫菲特(右列)患者gydF4y2Ba等gydF4y2Ba14gydF4y2Ba,CollissongydF4y2Ba等gydF4y2Ba15gydF4y2Ba和贝利gydF4y2Ba等gydF4y2Ba16gydF4y2Ba分类方案。假定值是两面的生存率较。gydF4y2BabgydF4y2Ba,瀑布的最好的肿瘤反应评估RECIST 1.1标准的66名胰腺癌患者罗盘试验根据以往的分类方案。假定值来自双边Wilcoxon rank-sum测试。gydF4y2Ba
扩展数据图2混合肿瘤基因表达分析证明他们土地上祝福表达连续性。gydF4y2Ba
一个gydF4y2Ba,只有8基因差异表达在混合动力车相比其他群体(热图所示)。这些8基因基本上限于Classical-B肿瘤的一个子集。被定义为基因的差异表达基因的调整假定值< = 0.05(双边瓦尔德测试之后,罗斯福校正)和绝对日志gydF4y2Ba2gydF4y2Ba(折叠变化(FC)) > = 1。gydF4y2BabgydF4y2Ba汇总表的数量差异表达基因中确定混合动力车和其他亚型。被定义为基因的差异表达基因的调整假定值< = 0.05(双边瓦尔德测试之后,罗斯福校正)和绝对日志gydF4y2Ba2gydF4y2Ba(FC) > = 1。gydF4y2BacgydF4y2Ba调节基因的热图(b)下令亚型。左侧面板描述比较每个基因调节的。gydF4y2BadgydF4y2Ba,维恩图解显示重叠(b)的调节基因识别。没有一般调节基因在混合动力车相比其他亚型。gydF4y2BaegydF4y2Ba跨子类型,箱线图显示EMT分数(混合Basal-likeB Basal-likeA = 27日= 24日= 57,Classical-like-A = 103和Classical-like-B = 36)。假定值来自双边Wilcoxon rank-sum测试。盒须图表示中位数,第一和第三个四分位数。胡须代表最极端点不超过1.5 *差。gydF4y2Ba
扩展数据图3亚型生存和化疗反应从无花果。gydF4y2Ba1gydF4y2Ba。gydF4y2Ba
一个gydF4y2Ba,生存分析比较Basal-like-B和混合肿瘤可切除的患者的三个分类(官腔,莫菲特gydF4y2Ba14gydF4y2Ba;quasi-mesenchymal, CollissongydF4y2Ba15gydF4y2Ba;和鳞状,贝利gydF4y2Ba16gydF4y2Ba)。gydF4y2BabgydF4y2Ba对可切除的病人生存分析基于5疾病亚型(I / II阶段;双边生存率较,p = 0.0004)。在每个子类型的患者数量。gydF4y2BacgydF4y2Ba,对先进的病人生存分析比较Basal-like-A (n = 14)和其他(n = 66) (III / IV阶段;双边生存率较,p = 0.12)。gydF4y2BadgydF4y2Ba66年先进的病人,RECIST瀑布图基于5疾病亚型。患者接受修改FOLFIRINOX或基于Gemcitabline / nab-paclitaxel化疗。假定值从两面Wilcoxon rank-sum测试Basal-like-A肿瘤和休息。gydF4y2Bae和fgydF4y2Ba,RECIST瀑布(假定值从两面Wilcoxon rank-sum测试)和生存的土地(假定值从双边生存率较先进的病人gydF4y2Bae)gydF4y2Ba修改FOLFIRINOX或gydF4y2Baf)gydF4y2BaGemcitabine-based治疗。gydF4y2Ba
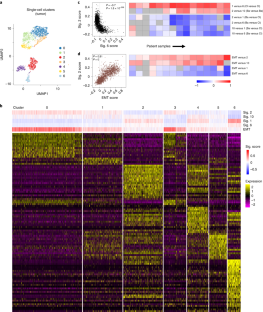
扩展数据图4单细胞RNA-seq分析病人的肿瘤。gydF4y2Ba
一个gydF4y2Ba负选择工作流是用来丰富肿瘤上皮细胞之前scRNA-seq 10 x平台。数据采集和处理。gydF4y2Bab和cgydF4y2Ba,在gydF4y2BabgydF4y2BaUMAP块单细胞集群从95373年病人负选择后细胞(n = 2252)。在gydF4y2BacgydF4y2Ba,谱系标记高亮显示上皮(gydF4y2BaEPCAM, KRT19gydF4y2Ba)、免疫细胞(gydF4y2BaPTPRC, CD33的gydF4y2Ba)和成纤维细胞(gydF4y2BaTHY1, ACTA2gydF4y2Ba)所示。这个分析是重复14日额外的样品。gydF4y2Bad和egydF4y2Ba鉴定上皮细胞后,细胞(n = 2121)(上图),然后试图识别各种类型的胰腺上皮细胞。在d, UMAP情节强调97727年病人上皮细胞集群。在gydF4y2BaegydF4y2Ba然后,上皮细胞集群外分泌标有谱系标记(gydF4y2BaSPP1,雌性生殖道gydF4y2Ba)和内分泌(gydF4y2BaGCG, INSgydF4y2Ba)细胞。这个分析是重复14日额外的样品。gydF4y2BafgydF4y2Ba,UMAP情节病人85948细胞(n = 2235)显示单个细胞集群。细胞类型识别的阴谋。gydF4y2BaggydF4y2Ba,单细胞RNA-seq数据搜索读着gydF4y2Ba喀斯特gydF4y2Ba突变。Mutation-positive细胞只存在于恶性肿瘤细胞集群(从gydF4y2BafgydF4y2Ba)。gydF4y2BahgydF4y2Ba,推断单细胞CNV的热图(gydF4y2Bahttps://github.com/broadinstitute/inferCNVgydF4y2Ba)从一个案例来验证分为恶性上皮细胞非整倍性。前跟踪显示了正常上皮的拷贝数概要文件。底跟踪底跟踪显示肿瘤中的拷贝数变化。gydF4y2Ba我gydF4y2Ba堆叠柱形图,4肿瘤签名分数为每个标识单个细胞集群从15个样本。集群分为三种类型;基底集群包含> 75%团体。2和10,经典集群包含团体> 75%。1和6,和混合集群由其余表达混合物的基底和经典签名。星号表示的一个集群缺乏表达4签名。gydF4y2Ba
扩展数据图5基因组分析转录子类型。gydF4y2Ba
一个gydF4y2Ba、肿瘤与同源重组缺陷(HRD,黄色)和错配修复(MMR),蓝色)缺乏突出显示。HRD, MMR推断使用特定基因组标准轮廓gydF4y2Ba9gydF4y2Ba(例如indels、sv和SNV负载突变签名(团体。3],体细胞和生殖细胞的突变基因在HRD / MMR pathway-ex。gydF4y2Ba乳腺癌易感基因1gydF4y2Ba,gydF4y2BaBRCA2, PALB2gydF4y2Ba或gydF4y2Ba一种MSH2、MSH6 PMS1 PMS2gydF4y2Ba)。gydF4y2BabgydF4y2Ba、基本亚型的基因分析。盒须图表示中位数+ / - 95%的置信区间。SNV-single核苷酸变异;SV-structural变异;BA-Basal-like-A (n = 23);BB-Basal-like-B (n = 23);Hy-hybrid (n = 52);CA-Classical-A (n = 94);Classical-B (n = 33)。盒须图所示值和第一/第三四分位数与所有点。gydF4y2BacgydF4y2Ba、频率显著突变基因的发现在WGS MutSigCV队列(n = 303)gydF4y2Ba49gydF4y2Ba。gydF4y2BadgydF4y2Ba,被MutSigCV循环Log10假定值的突变基因。假定值被从正反获得确切概率测试之间的确定基因突变的存在Basal-like-A / B (n = 46) vs Classical-A / B (n = 127)、Basal-like-A (n = 23)和休息(n = 202)。gydF4y2BaegydF4y2Ba,显著突变基因网络检测到HOTNET2 WGS队列(n = 303)。红色,蓝色规模描述意义的突变基因网络(炽热的;Blue-cold)。假定值计算的算法gydF4y2Ba50gydF4y2Ba。gydF4y2BafgydF4y2Ba中发现的基因,Oncoprint HOTNET2网络(见gydF4y2BaegydF4y2Ba基于亚型(n = 225)。gydF4y2Ba
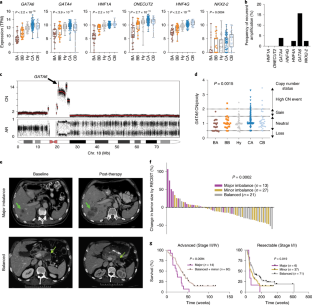
扩展数据图6通道gydF4y2BaGATA6gydF4y2Ba我们组的表达分析。gydF4y2Ba
一个gydF4y2Ba,丰富通路被GSEA Basal-like-A / B和Classical-A / B的肿瘤。gydF4y2BabgydF4y2Ba、基因频率不变gydF4y2BaSMAD4gydF4y2Ba等位基因(左面板),gydF4y2BaTP53gydF4y2Ba突变(中间面板)和完整的损失gydF4y2BaCDKN2AgydF4y2Ba(右面板)的亚型。假定值是双面的确切概率法(n = 225组;个人n值图所示)。gydF4y2BacgydF4y2Ba,gydF4y2BaGATA6gydF4y2Ba从图放大倍数性(归一化)。gydF4y2Ba3 dgydF4y2Ba基于初级(n = 170)分离和转移性肿瘤(n = 58)。假定值是克鲁斯卡尔-沃利斯的测试。gydF4y2BadgydF4y2Ba,gydF4y2BaGATA6gydF4y2Ba根据亚型表达原发性和转移性肿瘤。按照与上面相同的肿瘤。假定值是克鲁斯卡尔-沃利斯的测试。盒须图所示值和第一/第三四分位数与所有点。主要(n = 170);大都会(n = 58)。gydF4y2BaegydF4y2Ba、相关的gydF4y2BaGATA6gydF4y2Ba表达和拷贝数。占倍性相关的变化,的拷贝数gydF4y2BaGATA6gydF4y2Ba是标准化的倍性。双面的斯皮尔曼相关系数,假定值和肿瘤的数量在每个亚型在图所示。gydF4y2BafgydF4y2Ba(n = 3例),代表RNAish图像gydF4y2BaGATA6gydF4y2Ba相邻的外分泌组织的表达(管/腺泡的细胞)从病人正常的实质。gydF4y2Ba
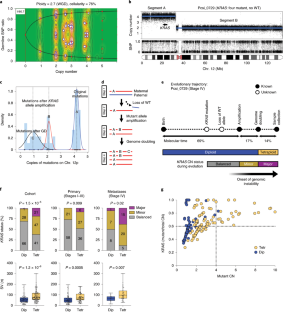
扩展数据图7肿瘤SV、倍性gydF4y2Ba喀斯特gydF4y2Ba不平衡的标准。gydF4y2Ba
一个gydF4y2Ba、突变频率gydF4y2Ba喀斯特gydF4y2Ba不平衡在原发性和转移性疾病亚型。被定义为不平衡(野生型等位基因拷贝数+ 1)。gydF4y2BabgydF4y2Ba、倍性分布主要与转移性疾病(四期)基于队列gydF4y2Ba(我)gydF4y2Ba和分子亚型gydF4y2Ba(2)gydF4y2Ba。假定值来自双边确切概率法。Pa-primary (n = 148);Met-metastatic (n = 56)。gydF4y2BacgydF4y2Ba、结构变体(SV)负担原发性和转移性disease-broken队列gydF4y2Ba(我)gydF4y2Ba并通过分子亚型gydF4y2Ba(2)gydF4y2BaBA-Basal-like-A;BB-Basal-like-B;Hy-hybrid;CA-Classical-A;Classical-B。只有肿瘤gydF4y2Ba喀斯特gydF4y2Ba变异进行了分析。假定值来自双边Wilcoxon rank-sum测试。盒须图所示值和第一/第三四分位数与所有点。群组数字一样gydF4y2BabgydF4y2Ba。gydF4y2Ba维,我gydF4y2Ba。拷贝数变异的情节gydF4y2Ba喀斯特gydF4y2Ba等位基因只在肿瘤的不平衡。有两个不同的集群和疲软的第三集群用更少的肿瘤。这支持,至少有两个不同的拷贝数不平衡分类(红色箭头)。两个不同的类别gydF4y2Ba喀斯特gydF4y2Ba不平衡。这个原理是用于开发三层系统(主要、次要、平衡)所示gydF4y2Ba二世gydF4y2Ba。gydF4y2Ba二世gydF4y2Ba。定义三种不同的拷贝数变异的不平衡gydF4y2Ba喀斯特gydF4y2Ba。失衡的程度是绘制突变CN(轴)和突变CN/Total CN(轴)。CN -拷贝数。gydF4y2Ba
扩展数据图8所示gydF4y2Ba喀斯特gydF4y2Ba失衡肿瘤亚型根据疾病阶段。gydF4y2Ba
一个gydF4y2Ba,突变体不平衡块gydF4y2Ba喀斯特gydF4y2Ba根据临床亚型阶段(前面板:四期;底板:》初级阶段)。在左边底部面板Basal-like-B只值显示在括号中。看到补充图。gydF4y2Ba8 fgydF4y2Ba。信息Basal-like-A肿瘤。gydF4y2BabgydF4y2Ba,对先进的病人生存分析分层根据程度的变异gydF4y2Ba喀斯特gydF4y2Ba不平衡(III / IV阶段;双边生存率较,p = 0.13)。gydF4y2BacgydF4y2Ba,盒须图比较的数量结构变异突变体的不同类别gydF4y2Ba喀斯特gydF4y2Ba不平衡在初级(主要= 9,次要= 62和平衡= 117)和转移性样品(主要= 21,次要= 36和平衡= 23)。假定值来自双边Wilcoxon rank-sum测试之间的平衡和专业。盒须图表示中位数,第一和第三个四分位数。胡须代表最极端点不超过1.5 *差。gydF4y2Ba
扩展数据图9生存和肿瘤响应分析基于肿瘤倍性和结构变异。gydF4y2Ba
一个gydF4y2Ba瀑布,RECIST块肿瘤反应基于倍性的肿瘤。假定值来自双边Wilcoxon rank-sum测试。gydF4y2BabgydF4y2Ba瀑布,RECIST块肿瘤反应基于类别被Waddell结构变体gydF4y2Ba等gydF4y2Ba。gydF4y2Ba1gydF4y2Ba。假定值来自双边Wilcoxon rank-sum测试。gydF4y2BacgydF4y2Ba生存分析基于肿瘤倍性或种族隔离结构变异的临床肿瘤阶段。假定值是两面的生存率较。数量的肿瘤和统计检验用图所示。gydF4y2Ba
扩展数据图10的分析两个病人显示肿瘤亚型的变化与疾病进展。gydF4y2Ba
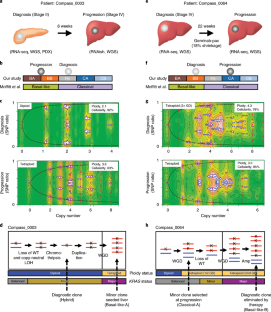
一个gydF4y2Ba,Compass_0003和Compass_0064肿瘤分子亚型集群系统树图的共识。gydF4y2BabgydF4y2Ba,RNAishgydF4y2BaGATA6gydF4y2Ba从Compass_0003肝脏活检。缺乏gydF4y2BaGATA6gydF4y2Ba的表情证实了官腔表型。gydF4y2BacgydF4y2Ba、基因组分析Compass_0003 Compass_0064。酒吧SNVs和indels情节显示数字。圆环图显示全基因组sv。gydF4y2BadgydF4y2Ba、拷贝数和生殖系SNP情节chr12从诊断(顶部面板)和肝转移(下半部分)。一个独特的重复事件放大变异gydF4y2Ba喀斯特gydF4y2Ba发现只有在转移。gydF4y2BaegydF4y2Ba综合基因组查看器(进口)的基因组区域形象显示paired-end读取(绿色)表示的重复事件(gydF4y2BadgydF4y2Ba)在肝转移Compass_0003(下半部分)。相同的重复不在诊断(顶面板),但出现在的主要肿瘤提取异种移植(PDX;中间面板)。gydF4y2BafgydF4y2Ba电影情节,PDX Compass_0003原发性肿瘤的生成。gydF4y2BaggydF4y2Ba、拷贝数和生殖系SNP块12号染色体显示gydF4y2Ba喀斯特gydF4y2Ba状态诊断(顶部面板)和Compass_0064进展(下半部分)。gydF4y2Ba
补充信息gydF4y2Ba
权利和权限gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
Chan-Seng-Yue, M。、金、J.C.,Wilson, G.W.et al。gydF4y2Ba胰腺癌的表型是由基因组转录事件在肿瘤的进化。gydF4y2BaNat麝猫gydF4y2Ba52gydF4y2Ba,231 - 240 (2020)。https://doi.org/10.1038/s41588 - 019 - 0566 - 9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41588 - 019 - 0566 - 9gydF4y2Ba
本文引用的gydF4y2Ba
针对p53通路:机制、结构和先进的治疗gydF4y2Ba
信号转导和有针对性的治疗gydF4y2Ba(2023)gydF4y2Ba
胰腺导管腺癌基因表达分析的开源内容管理平台(pdacR)支持two-subtype模型gydF4y2Ba
通信生物学gydF4y2Ba(2023)gydF4y2Ba
应用高通量single-nucleus DNA测序在胰腺癌gydF4y2Ba
自然通讯gydF4y2Ba(2023)gydF4y2Ba
新辅助治疗胰腺癌gydF4y2Ba
自然评论临床肿瘤学gydF4y2Ba(2023)gydF4y2Ba
RBFOX2调节可变剪接的签名转移性胰腺癌gydF4y2Ba
自然gydF4y2Ba(2023)gydF4y2Ba
