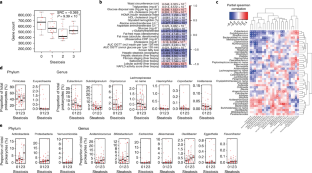
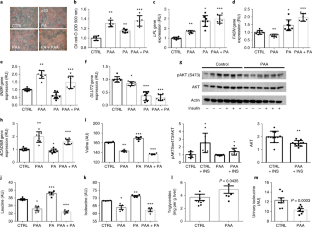
摘要
肝脂肪变性是一种多因素的疾病,常在肥胖患者中观察到,是非酒精性脂肪性肝病的前奏。在这里,我们将粪便宏基因组的鸟枪测序与分子表型学(肝脏转录组、血浆和尿液代谢组)结合起来,对两组特征良好的肥胖症女性进行FLORINASH研究。我们揭示了连接肠道微生物组和宿主表型与肝脏脂肪变性的分子网络。脂肪变性患者的微生物基因丰富度较低,在处理膳食脂类和内毒素生物合成(主要来自变形菌门)、肝脏炎症和芳香和支链氨基酸代谢失调方面具有较高的遗传潜力。我们证明,粪便微生物群移植和苯乙酸(芳香族氨基酸代谢的微生物产物)的长期治疗成功地触发脂肪变性和支链氨基酸代谢。分子表型特征是可预测的(曲线下面积= 87%),并且与肠道微生物组对脂肪变性表型的影响一致(>75%共享变异),因此,可通过基于微生物的治疗采取行动。
这是订阅内容的预览,通过你的机构获取
相关的文章
引用本文的开放存取文章。
非酒精相关性脂肪肝/脂肪性肝炎(NAFL/NASH)的靶向治疗和新的信号通路
信号转导与靶向治疗开放获取2022年8月13日
微生物-肠道-大脑轴:改善大脑健康的途径。关于我们知道什么,我们需要研究什么以及如何将知识付诸实践的观点
细胞与分子生命科学开放获取2022年1月19日
肠道微生物群在慢性疾病中的作用:述评
欧洲临床营养学杂志开放获取2021年9月28日
访问选项
订阅Nature+
立即在线访问整个自然家族的50多种期刊
29.99美元
每月
订阅期刊
获得1年的完整期刊访问权限
99.00美元
每期仅需8.25美元
所有价格均为净价格。
增值税将在稍后的结帐中添加。
税金计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问权限。
32.00美元
所有价格均为净价格。




改变历史
2018年8月09
在本文最初发布的版本中,接收日期缺失。它应该被列为2018年1月2日。该错误已在本文的HTML和PDF版本中得到纠正。
参考文献
Saltiel, a.r. & Kahn, c.r.胰岛素信号转导与糖脂代谢的调节。自然414, 799-806(2001)。
卡恩,S. E,赫尔,R. L.和Utzschneider, K. M.肥胖与胰岛素抵抗和2型糖尿病的联系机制。自然444, 840-846(2006)。
肝因子:非酒精性脂肪肝与胰岛素抵抗的关系。内分泌内分泌素。13, 509-520(2017)。
Petersen, m.c., Vatner, d.f. & Shulman, g.i.健康和疾病中肝脏葡萄糖代谢的调节。内分泌内分泌素。13, 572-587(2017)。
亚当斯,l.a, Anstee, Q. M, Tilg, H. & Targher, G.非酒精性脂肪肝及其与心血管疾病和其他肝外疾病的关系。的直觉。66, 1138-1153(2017)。
小仲马,M.-E。et al。代谢分析揭示了肠道菌群对胰岛素抵抗小鼠脂肪肝表型的贡献。美国国家科学院学报。美国103, 12511-12516(2006)。
勒罗伊,T.等人。肠道菌群决定小鼠非酒精性脂肪肝的发展。的直觉。62, 1787-1794(2013)。
秦,N.等。肝硬化患者肠道菌群的改变。自然513, 59-64(2014)。
Turnbaugh, P. J.等。一种与肥胖相关的肠道微生物群,其能量收集能力增强。自然444, 1027-1031(2006)。
Turnbaugh, P. J.等。肥胖和瘦弱双胞胎的核心肠道微生物群。自然457, 480-484(2009)。
Ridaura, V. K.等。肥胖双胞胎肠道菌群不协调调节小鼠代谢。科学341, 1241214(2013)。
微生物将胆碱转化为三甲胺需要一种甘氨酸自由基酶。美国国家科学院学报。美国109, 21307-21312(2012)。
霍伊尔斯等人。三甲胺的代谢反转化N-氧化物和肠道菌群微生物组。6, 73(2018)。
王铮等。磷脂酰胆碱的肠道菌群代谢促进心血管疾病。自然472, 57-63(2011)。
小仲马,M.-E。et al。微生物-宿主共同代谢物是预测行为、肥胖和糖耐量受损的表型异质性的前驱标志物。细胞的代表。20., 136-148(2017)。
Lonardo, A.等人。脂肪肝与糖尿病和心血管疾病风险的增加有关,证据来自三种不同的疾病模型:NAFLD、HCV和HIV。世界J.胃肠醇。22, 9674-9693(2016)。
Houle, Govindaraju, D. R. & Omholt, S.表型学:下一个挑战。Nat. Rev. Genet。11, 855-866(2010)。
小仲马,M.-E。,Kinross, J. & Nicholson, J. K. Metabolic phenotyping and systems biology approaches to understanding metabolic syndrome and fatty liver disease.胃肠病学146, 46-62(2014)。
勒夏特列等人。人肠道菌群丰富度与代谢标志物相关。自然500, 541-546(2013)。
纽加德,C. B.等。一种支链氨基酸相关的代谢特征,区分肥胖和瘦人,有助于胰岛素抵抗。细胞金属底座。9, 311-326(2009)。
张,C.等。支链氨基酸代谢物驱动血管脂肪酸运输,引起胰岛素抵抗。Nat,地中海。22, 421-426(2016)。
Karlsson, F. H.等。欧洲葡萄糖控制正常、受损和糖尿病妇女的肠道宏基因组。自然498, 99-103(2013)。
Forslund, K.等。解开2型糖尿病和二甲双胍在人类肠道菌群中的治疗特征。自然528, 262-266(2015)。
吴,H.等。二甲双胍改变了未治疗的2型糖尿病患者的肠道微生物群,有助于该药物的治疗效果。Nat,地中海。23, 850-858(2017)。
欧洲肝脏研究协会(EASL),欧洲糖尿病研究协会(EASD),欧洲肥胖研究协会(EASO)非酒精性脂肪肝的临床实践指南。Diabetologia59, 1121-1140(2016)。
欧洲肝脏研究协会(EASL),欧洲糖尿病研究协会(EASD),欧洲肥胖研究协会(EASO)非酒精性脂肪肝的临床实践指南。j .乙醇。64, 1388-1402(2016)。
克莱纳,D. E.等人。非酒精性脂肪性肝病组织学评分系统的设计与验证。肝脏病学41, 1313-1321(2005)。
Pedersen, H. K.等。人肠道微生物影响宿主血清代谢组和胰岛素敏感性。自然535, 376-381(2016)。
秦杰等。2型糖尿病患者肠道菌群的宏基因组相关性研究。自然490, 55-60(2012)。
福尔摩斯,E.等。人类代谢表型多样性及其与饮食和血压的关系。自然453, 396-400(2008)。
彭颖,梁鸿昌,姚淑敏,陈凤英利。IDBA-UD:一种用于深度高度不均匀的单细胞和宏基因组测序数据的全新汇编器。生物信息学28, 1420-1428(2012)。
推导基因发现模型的启发式方法。核酸储备。27, 3911-3920(1999)。
Zhu, W., Lomsadze, A. & Borodovsky, M.宏基因组序列中Ab initio基因的鉴定。核酸储备。38, e132(2010)。
李,J.等。人类肠道微生物菌群中参考基因的综合目录。生物科技Nat。》。32, 834-841(2014)。
Cani, P. D.等。代谢性内毒素血症引发肥胖和胰岛素抵抗。糖尿病56, 1761-1772(2007)。
Shoaie, s。定量饮食诱导的人体肠道微生物群代谢变化。细胞金属底座。22, 320-331(2015)。
斯宾塞,医学博士等人。人胃肠菌群组成与脂肪肝胆碱缺乏症发展之间的关系。胃肠病学140, 976-986(2011)。
Pallister, T.等人。河马酸盐作为肠道微生物群多样性的代谢组标志物:受饮食的调节和与代谢综合征的关系。科学。代表。7, 13670(2017)。
卡达尔,H.等人。用超高效液相色谱-高分辨率质谱联用的高通量血清甲胺的多目标定量分析方法。拱门。物化学。Biophys。59712-20(2016)。
普罗维耶,H.等。一种纯化的膜蛋白Akkermansia muciniphila或者巴氏杀菌细菌可以改善肥胖和糖尿病小鼠的新陈代谢。Nat,地中海。23, 107-113(2017)。
舒格,R. C.等。产生tmao的含黄素的单加氧酶3调节肥胖和白色脂肪组织的褐变。细胞的代表。19, 2451-2461(2017)。
Tarca, A. L.等。一种新的信号通路影响分析。生物信息学25, 75-82(2009)。
Davidovic, L.等。脆性X综合征小鼠模型大脑的代谢组学和系统生物学视角。基因组Res。21, 2190-2202(2011)。
Rodriguez-Martinez等人。代谢信号:一种基于网络的方法,用于通过代谢和信号通路进行代谢调节的拓扑分析。生物信息学33, 773-775(2017)。
比丁格,S. B.等。肝脏胰岛素抵抗直接促进胆固醇胆结石的形成。Nat,地中海。14, 778-782(2008)。
迈克尔,m.d.等人。肝细胞中胰岛素信号的丢失导致严重的胰岛素抵抗和进行性肝功能障碍。摩尔。细胞6, 87-97(2000)。
Fujisaka, S.等。抗生素对肠道菌群和代谢的影响取决于宿主。j .中国。投资.126, 4430-4443(2016)。
尼古拉斯,S.等。肠道不良菌群的转移对宿主肝脏代谢有有益作用。摩尔。系统。医学杂志。13, 921(2017)。
Grasset, E.等人。2型糖尿病小鼠特异性肠道菌群失调通过肠内no依赖和肠-脑轴机制诱导GLP-1耐药。细胞金属底座。25, 1075 - 1090。e5(2017)。
拉托尔等人。在NAFLD肥胖受试者中,脂质代谢降低但FA生物合成增加与肝microRNAs的变化相关。Int。j . ob。(Lond)。41, 620-630(2017)。
歌迪亚等。膳食干预对肠道微生物基因丰富度的影响。自然500, 585-588(2013)。
Sharifnia, T.等人。肥胖NAFLD中的肝脏TLR4信号转导点。j .杂志。Gastrointest。肝脏杂志。309, g270-g278(2015)。
泰斯,C. A.等人。持续的微生物组改变调节节食后体重恢复的速度。自然540, 540-551(2016)。
Zeevi, D.等。通过预测血糖反应的个性化营养。细胞163, 1079-1094(2015)。
Boursier, J.等。非酒精性脂肪性肝病的严重程度与肠道菌群失调和肠道菌群代谢功能的改变有关。肝脏病学63, 764-775(2016)。
伦巴,R.等人。基于肠道微生物的宏基因组特征用于非酒精性脂肪肝晚期纤维化的无创检测细胞金属底座。25, 1054 - 1062。e5(2017)。
塞里诺等人。对高脂肪饮食的代谢适应与肠道菌群的变化有关。的直觉。61, 543-553(2012)。
克莱森,M. J.等。老年人肠道菌群组成与饮食和健康相关。自然488, 178-184(2012)。
法洛尼,G.等。肠道微生物组变异的人群水平分析。科学352, 560-564(2016)。
格拉萨,m.d.m.等人。SHBG结合睾酮和雌二醇的性别和病态肥胖的调节。欧元。j .性。176, 393-404(2017)。
美国糖尿病协会。糖尿病的分类和诊断:糖尿病医疗护理标准-2018。糖尿病护理41, s13-s27(2018)。
Dieterle, F., Ross, A., Schlotterbeck, G. & Senn, H.概率商归一化作为解释复杂生物混合物稀释的稳健方法。应用程序在1H NMR代谢组学。肛交。化学。78, 4281-4290(2006)。
Fleige, S. & Pfaffl, M. W. RNA完整性及其对实时qRT-PCR性能的影响。摩尔方面医学。27, 126-139(2006)。
史密斯。limma:微阵列数据的线性模型使用R和Bioconductor的生物信息学和计算生物学解决方案.生物与健康统计丛书(mr . R., Carey, V., Huber, W., Irizarry, R., Dudoit, S.) 397-420(施普林格,纽约,纽约,2005)。
张建东和魏曼。KEGGgraph: R和生物导体中KEGG通路的图解方法。生物信息学25, 1470-1471(2009)。
陈爱云,陈爱云等。richr:交互式和协作的HTML5基因列表丰富分析工具。BMC生物信息学14, 128(2013)。
Kuleshov, m.v.等。全面基因集富集分析web服务器2016更新。核酸储备。44, w90-w97(2016)。
Lluch, J.等。利用优化的16S宏基因组测序管道对新型组织微生物群进行表征。《公共科学图书馆•综合》10, e0142334(2015)。
Escudié, F.等。青蛙:用星系解决方案快速找到OTUs。生物信息学34, 1287-1294(2018)。
Segata, N.等。使用独特的进化枝特异性标记基因的宏基因组微生物群落分析。Nat方法。9, 811-814(2012)。
Truong, d.t.等。用于增强宏基因组分类分析的MetaPhlAn2。Nat方法。12, 902-903(2015)。
搜索和聚类的数量级比BLAST快。生物信息学26, 2460-2461(2010)。
小仲马,M.-E。et al。分析重现性的评估1基于H NMR的代谢组学用于大规模流行病学研究:INTERMAP研究。肛交。化学。78, 2199-2208(2006)。
Smilde, a.k, Kiers, h.a.l, Bijlsma, S., Rubingh, c.m. & van Erk, m.j.高维数据的矩阵相关性:修正的rv系数。生物信息学25, 401-405(2009)。
Cloarec, O.等。对潜在结构模型的局限性的正交投影的评估,由化学位移的可变性和改进的生物标志物变化的可视化1H NMR光谱代谢组学研究。肛交。化学。77, 517-526(2005)。
布莱斯,B. J.等。Metabotyping的秀丽隐杆线虫揭示潜在的表现型。美国国家科学院学报。美国104, 19808-19812(2007)。
确认
我们感谢N. J. Gooderham对手稿的批判性阅读。本研究得到EU-FP7 FLORINASH (Health-F2-2009-241913)对R.B, M.F, j -M.F - r的支持。,F.F., C.P., E.H. and J.K.N. This work used the computing resources of the UK MEDical BIOinformatics partnership—aggregation, integration, visualization and analysis of large, complex data (UK MED-BIO), which is supported by the Medical Research Council (grant number MR/L01632X/1). L.H. is in receipt of an MRC Intermediate Research Fellowship in Data Science (MR/L01632X/1, UK MED-BIO). This work was also supported by funding to R.B. ((Région Midi-Pyrénées 2009-2014 RPV09003BBA), Agence Nationale de la Recherche ANR 09-GEN TRANSFLORAP and GAD 08-2_378258), to M.F. (Ministry of University (MIUR) Progetti di Ricerca di Interesse Nazionale (PRIN) protocol number 2015MPESJS_004, Ministry of Health Ricerca Finalizzata RF-2011-02349921, Fondazione Roma call for Non-Communicable Diseases NCD 2014), to J.-M.F.-R. (Ministry of Health FIS project 15/01934, CIBERobn Pathophysiology of Obesity and Nutrition and FEDER funds) and to M.-E.D. (EU METACARDIS under agreement HEALTH-F4-2012-305312, Neuron II under agreement 291840 and the MRC MR/M501797/1). We acknowledge the support of the Imperial College National Institute of Biomedical Research andthe Clinical Phenotyping Centre for support.
作者信息
作者和隶属关系
贡献
R.B J.-M.F.-R。,M.F., F.L., F.F., C.P., E.H. and J.K.N. designed the study and supervised all parts of the project. R.B. is the project leader and chaired the consortium. M.-E.D. led data integration and elaborated the primary interpretation of analytical outcomes with L.H., in close collaboration with M.F., J.-M.F.-R. and R.B. L.H. implemented the microarray data analysis workflow. C.T. and M.W. developed the data repository. J.A. developed the metagenomic data analysis pipeline in collaboration with L.H. S.A.B. supervised the development of the data repository and the pipeline. L.H., J.A., R.H.B. and M.-E.D. performed the data analyses. J.-M.F.-R. and M.F. designed the clinical protocol and oversaw the clinical activities. M.C., F.D., I.C., O.P., P.G., J.P., G.X. and W.R. recruited and phenotyped patients and collected biological samples and physiological data. M.S., V.A. and V.B.-B. performed the RNA and DNA extractions. R.B. and M.S. supervised the DNA sequencing and gene profiling. J.L.L. and J.M.M.-N. performed the cell culture experiments. F.B., E.A., J.Charpentier and C.H. performed the animal work. R.H.B., J.Chilloux and L.M.-G. performed the metabolic profiling of plasma and urine by1核磁共振。E.H.和J.K.N.负责代谢分析。A.M.进行了甲胺定量。L.H.和m.e.d.起草了论文的第一个版本,m.f.、j.m. f.r对论文做出了重要和实质性的贡献。在整个项目中,所有作者都提供了支持和建设性的批评,并批准了论文的最终版本。
相应的作者
道德声明
相互竞争的利益
L.H J.-M.F.-R。,M.F., R.H.B., J.L.L., E.H., J.K.N., C.P., R.B. and M.-E.D. are named as co-inventors on pending patents held by INSERM Transfert, INSERM, University of Rome Tor Vergata, University of Girona and Imperial College on NAFLD diagnostics and have the right to receive royalty payments for inventions or discoveries related to NAFLD diagnostics.
额外的信息
出版商的注意:施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
权利与权限
关于本文
引用本文
霍尔斯,L., Fernández-Real, JM。费德里奇(Federici), M。et al。非糖尿病肥胖女性肝脏脂肪变性的分子表型学和宏基因组学。Nat地中海24, 1070-1080(2018)。https://doi.org/10.1038/s41591-018-0061-3
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41591-018-0061-3
进一步的阅读
肠道微生物群在慢性疾病中的作用:述评
欧洲临床营养学杂志(2022)
非酒精相关性脂肪肝/脂肪性肝炎(NAFL/NASH)的靶向治疗和新的信号通路
信号转导与靶向治疗(2022)
2型糖尿病小鼠肠道菌群失调损害GLP-1敏感性的肠道日常节律
Acta Diabetologica(2022)
宿主-微生物在代谢性疾病中的相互作用:从饮食到免疫
微生物学杂志(2022)
微生物-肠道-大脑轴:改善大脑健康的途径。关于我们知道什么,我们需要研究什么以及如何将知识付诸实践的观点
细胞与分子生命科学(2022)
