摘要
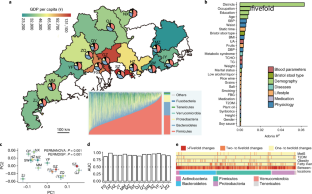
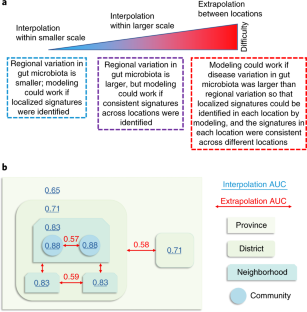
生态失调,即肠道微生物组脱离健康状态,已被认为是疾病发病率和进展的有力生物标志物1,2,3..已提出在炎症性肠病诊断和预后方面的诊断应用4,结直肠癌预检5以及黑色素瘤的治疗选择6.无创采样可以促进大规模的公共卫生应用,包括代谢的早期诊断和风险评估7心血管疾病8.为了了解基于微生物群的代谢性疾病诊断模型的广泛性,我们对来自中国一个省14个地区的7,009人的肠道微生物群进行了特征分析。在表型中,宿主定位与菌群变异的相关性最强。在一个地方开发的基于微生物群的代谢疾病模型在其他地方使用时失败了,这表明这种模型不能外推。插值模型表现得更好,特别是在具有明显微生物群相关特征的疾病中。插值效率随着地理规模的增加而降低,这表明需要建立局部基线和疾病模型来预测代谢风险。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
社会与生物在哪里相遇:生物社会种族的新本体论
综合开放获取2023年1月04日
大规模肥胖人群肠道微生物生物标记物发现和调节分析的机器学习框架
BMC基因组学开放获取2022年12月23日
口腔真菌菌群与胰腺导管腺癌
BMC癌症开放获取12月2日
访问选项
访问《自然》和其他54种《自然》杂志
获取Nature+,我们最超值的在线订阅
每月29.99美元
随时取消
订阅这本杂志
收到12个印刷问题和在线访问
每年189.00美元
每期只要15.75美元
租或购买这篇文章
只要这篇文章,只要你需要它
39.95美元
价格可能受当地税收的影响,在结账时计算

改变历史
2018年9月24日
在本文最初发表的版本中,在“将相同的方法应用于肥胖(图2b), MetS(图2c)和脂肪肝(图2d)产生了类似的结果”这句话中,两个图面板被错误引用。肥胖数据见图2c,代谢当量数据见图2b。这句话已经在文章的印刷版、PDF版和HTML版中得到了正确的引用。
参考文献
Lynch, S. V. & Pedersen, O.人体肠道微生物群在健康和疾病中的作用。心血管病。j .地中海。375, 2369-2379(2016)。
吉尔伯特,J. A.等。微生物群范围内的关联研究将动态微生物群与疾病联系起来。自然535, 94-103(2016)。
吉尔伯特,J. A.等。目前对人类微生物群的了解。Nat,地中海。24, 392-400(2018)。
Dubinsky, M. & Braun, J.炎症性肠病的诊断和预后微生物生物标志物。胃肠病学149, 1265 - 1274。e3(2015)。
Konstantinov, s.r., Kuipers, e.j. & Peppelenbosch, m.p.肠道微生物群用于CRC筛查的功能基因组分析。胃肠醇。乙醇。10, 741-745(2013)。
Routy, B.等人。肠道微生物影响pd -1免疫治疗上皮性肿瘤的疗效科学359, 91-97(2018)。
Bouter, K. E, van Raalte, D. H., Groen, A. K. & Nieuwdorp, M.肠道微生物组在肥胖和肥胖相关代谢功能障碍发病机制中的作用。胃肠病学152, 1671-1678(2017)。
唐伟华等。磷脂酰胆碱肠道微生物代谢与心血管风险。心血管病。j .地中海。368, 1575-1584(2013)。
格雷厄姆,C.,马伦,a . &惠兰,K.肥胖和胃肠道微生物群:关联和机制的回顾。减轻。牧师。73, 376-385(2015)。
Wieland, A, Frank, D. N, Harnke, B. & Bambha, K.系统综述:微生物失调与非酒精性脂肪肝。滋养品。杂志。其他。42, 1051-1063(2015)。
Forslund, K.等人。解开人类肠道菌群中的2型糖尿病和二甲双胍治疗特征。自然528, 262-266(2015)。
Yatsunenko, T.等。不同年龄和地理位置的人类肠道微生物组。自然486, 222-227(2012)。
Martínez, I.等。巴布亚新几内亚农村的肠道微生物群:组成、多样性模式和生态过程。细胞的报道11, 527-538(2015)。
Gupta, V. K, Paul, S. & Dutta, C.人类微生物组组成和多样性的地理、种族或生存特定差异。前面。Microbiol。8, 1162(2017)。
Winglee, K.等人。中国最近的城市化与编码毒性和抗生素耐药性基因的西方化微生物组有关。微生物组5, 121(2017)。
Costea, p.i.等。朝着宏基因组研究中人类粪便样本处理的标准迈进。生物科技Nat。》。35, 1069-1076(2017)。
法洛尼,G.等。肠道微生物组变异的人群水平分析。科学352, 560-564(2016)。
卡波拉索,J. G.等。QIIME允许分析高通量群落测序数据。Nat方法。7, 335-336(2010)。
Zhernakova, A.等人。基于人群的宏基因组分析揭示了肠道微生物组组成和多样性的标记。科学352, 565-569(2016)。
Anderson, m.j. & Walsh, d.c.i. PERMANOVA, ANOSIM,以及面对异质分散的Mantel检验:你在检验什么零假设?生态。Monogr。83, 557-574(2013)。
周,Y.等。肠道菌群在炎症性肠病诊断和英夫利昔单抗反应预测中提供了跨种族的通用生物标志物。mSystems3., e00188-e00117(2018)。
于,J.等。粪便微生物组的宏基因组分析可作为结直肠癌靶向非侵入性生物标志物的工具。肠道66, 70-78(2017)。
Walters, W. A., Xu, Z. & Knight, R.与肥胖和IBD相关的人类肠道微生物的元分析。2月。588, 4223-4233(2014)。
麦克唐纳等人。美国肠道:公民科学微生物组研究的开放平台。mSystems3., e00031-18(2018)。
斯塔格曼,K.等。市场整合在经济发展的梯度上预测人类肠道微生物组的属性。mSystems3., e00122-17(2018)。
科斯特洛,E. K.,斯塔格曼,K.,德瑟莱夫森,L.,博汉南,B. J. &雷尔曼,D. A.生态学理论在理解人类微生物群方面的应用。科学336, 1255-1262(2012)。
廖,M.等。中国孤立人群瑶族、壮族和汉族农村人群粪便微生物群16sRNA测序比较分析科学。代表。8, 1142(2018)。
沃尔特斯,W.等人。改进的细菌16S rRNA基因(V4和V4-5)和真菌内部转录间隔标记基因引物用于微生物群落调查。mSystems1, e00009-e00015(2015)。
Frank, D. N. BARCRAWL和BARTAB:用于设计和实现高度多路DNA测序的条形码引物的软件工具。BMC生物信息学10, 362(2009)。
麦克唐纳,D,伯明翰,A.和奈特,R.环境和人类微生物组。微生物组3., 52(2015)。
搜索和聚类速度比BLAST快几个数量级。生物信息学26, 2460-2461(2010)。
麦克唐纳等人。一种改进的绿基因分类学,用于细菌和古生菌的生态和进化分析。ISME。J。6, 610-618(2012)。
他,Y.等人。操作分类单位的稳定性:分析微生物多样性的一个重要但被忽视的特性。微生物组3., 20(2015)。
麦克唐纳等人。生物观察矩阵(BIOM)格式或者:我如何学会停止担忧,爱上某物。Gigascience1, 7(2012)。
摩根,X. C.等。炎症性肠病肠道微生物功能障碍及其治疗基因组。医学杂志。13, r79(2012)。
谢,H.等。250对成年双胞胎的鸟枪宏基因组揭示了遗传和环境对肠道微生物群的影响。细胞系统。3., 572-584(2016)。
Lim, M. Y.等。遗传性和宿主遗传学对肠道菌群和代谢综合征的影响。肠道66, 1031-1038(2017)。
确认
我们感谢308名当地疾控中心调查员和注册护士在收集点维护、元数据和粪便样本收集方面的帮助。我们感谢所有参与这个项目的志愿者。国家重大传染病预防控制项目(2017ZX10103011 (H.W.Z.))、国家自然科学基金(NSFC31570497 (H.W.Z.)、31322003 (H.W.Z.)、81671171 (J.Y.))、中国博士后科学基金(C1090132 (Y.H.))、广东省科技计划项目(2015A030401055 (W.W.))资助。
作者信息
作者及隶属关系
贡献
y.h.、w.w.、w.j.m.和h.w.z.设计了这项研究;w.w.、c.z. h .、z - h.c .、g.y.j .、y.j.x .和w.j.m .组织数据收集和培训当地疾控中心调查员;M.-X.C Z.-D.-X.Z。下午,X.-J.C, Z.-H.R, L.-Y.L.和纽约州处理样品;,12 H.-M.Z。,P.L H.-F.S, X.W, C.-B.W,古滑坡体,J.Y. H.-W.Z.分析数据;y.h.、d.m.、w.j.m.、R.K.和h.w.z.起草了手稿;R.K.和J.R.就研究设计、数据分析和手稿写作提供了建议。
相应的作者
道德声明
相互竞争的利益
l.y.l .、X.W.及c.b.w .为深圳市Fun-Poo生物科技有限公司员工。
额外的信息
出版商的注意:施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
关于本文
引用本文
何,Y.,吴,W.,郑,HM.。et al。区域差异限制了健康肠道微生物组参考范围和疾病模型的应用。Nat地中海24, 1532-1535(2018)。https://doi.org/10.1038/s41591-018-0164-x
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41591-018-0164-x
这篇文章被引用
社会与生物在哪里相遇:生物社会种族的新本体论
综合(2023)
机器学习用于人体肠道微生物组的数据集成
微生物细胞工厂(2022)
食管鳞状细胞癌中食管微生物群的特征及相互作用
BMC癌症(2022)
年龄越大,控制体重对2型糖尿病的潜在好处就越少
BMC老年病学(2022)
长期和短期植物性饮食模式和心脏代谢健康的肠道微生物群特征:一项前瞻性队列研究
BMC医学(2022)
