摘要
相关研究已将微生物组的改变与许多人类疾病联系起来。然而,他们并不总是报告一致的结果,因此需要进行交叉研究比较。在这里,对八项地理上和技术上不同的结肠直肠癌粪便散弹枪宏基因组研究(CRC,n= 768),在几个混杂因素的控制下,鉴定出一个核心的29个物种CRC宏基因组显著富集(错误发现率(FDR) < 1 × 10−5).从单一研究中得到的CRC特征在其他研究中保持了准确性。通过多项研究的训练,我们提高了CRC的检测准确性和疾病特异性。CRC宏基因组的功能分析显示,蛋白质和粘蛋白分解代谢基因富集,碳水化合物降解基因缺失。此外,我们推断CRC宏基因组中次生胆汁酸的产生增加,这表明癌症相关的肠道微生物与富含脂肪和肉类的饮食之间存在代谢联系。通过广泛的验证,该元分析牢固地建立了全球可推广的、可预测的分类和功能微生物组CRC特征,作为未来诊断的基础。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
重复使用已付费健康计划的粪便免疫化学测试,以检测结肠直肠腺瘤与微生物组的相关性
科学报告开放获取2022年8月31日
只需添加数据:用于知识发现和特征选择的自动预测建模
npj精密肿瘤学开放获取2022年6月16日
大规模微生物组数据集成可实现可靠的生物标志物识别
自然计算科学开放获取2022年5月23日
访问选项
订阅《自然》+
立即在线访问整个自然家族的50多种期刊
29.99美元
每月
订阅期刊
获得1年的完整期刊访问权限
99.00美元
每期只要8.25美元
所有价格均为净价格。
增值税稍后将在结帐时添加。
税务计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问。
32.00美元
所有价格均为净价格。
数据可用性
德国研究中样本的原始测序数据以前没有发表过(见方法)可从欧洲核苷酸档案(研究编号:PRJEB27928.这些示例的元数据可在补充表中找到6.
对于当前研究中包括的其他研究,原始测序数据可以在以下欧洲核苷酸档案标识符下找到:PRJEB10878Yu等人。11;PRJEB12449为Vogtmann等人。10;ERP008729为Feng等。9;ERP005534为Zeller等。8.独立的验证队列可以在Sequence Read Archive的标识符号下找到。SRP136711托马斯等人。27并存入日本DNA数据库,编号为。DRA006684.
作为统计建模管道输入的过滤分类和功能概要文件可在补充数据中获得1.
代码和所有分析结果可以在下面找到https://github.com/zellerlab/crc_meta.
参考文献
Tringe, s.g. & Rubin, e.m.宏基因组学:环境样本的DNA测序。Nat. Rev. Genet。6, 805-814(2005)。
Tremaroli, V. & Bäckhed, F.肠道微生物群与宿主代谢之间的功能相互作用。自然489, 242-249(2012)。
Lynch, S. V. & Pedersen, O.人体肠道微生物群在健康和疾病中的作用。心血管病。j .地中海。375, 2369-2379(2016)。
秦,等。2型糖尿病患者肠道菌群宏基因组范围的关联研究。自然490, 55-60(2012)。
卡尔森,F. H.等。正常、受损和糖尿病血糖控制的欧洲女性的肠道宏基因组。自然498, 99-103(2013)。
秦,等。宏基因组测序建立的人体肠道微生物基因目录。自然464, 59-65(2010)。
席尔默等人。炎症性肠病肠道微生物群中代谢的动态。Microbiol Nat。3., 337-346(2018)。
泽勒,G.等。粪便微生物群在结直肠癌早期检测中的潜力。摩尔。系统。医学杂志。10, 766(2014)。
冯,Q.等。肠道微生物组沿结直肠腺瘤-癌序列的发展。Commun Nat。6, 6528(2015)。
沃格特曼,E.等人。结直肠癌和人类肠道微生物组:全基因组霰弹枪测序的可重复性。《公共科学图书馆•综合》11, e0155362(2016)。
于,J.等。粪便微生物组的宏基因组分析可作为结直肠癌靶向非侵入性生物标志物的工具。肠道66, 70-78(2017)。
贝达夫,J. R.等。微生物和病毒肠道宏基因组变化在早期L-DOPA-naïve帕金森病患者中的功能意义。基因组医学。9, 39(2017)。
施密特,T. S. B.,瑞斯,J.和博克,P.人体肠道微生物群:从关联到调节。细胞172, 1198-1215(2018)。
Forslund, K.等人。解开人类肠道菌群中的2型糖尿病和二甲双胍治疗特征。自然528, 262-266(2015)。
Costea, p.i.等。朝着宏基因组研究中人类粪便样本处理的标准迈进。生物科技Nat。》。35, 1069-1076(2017)。
Lozupone, c.a.等。人类微生物群研究的元分析。基因组Res。23, 1704-1714(2013)。
Duvallet, C., Gibbons, S. M., Gurry, T., Irizarry, R. A. & Alm, E. J.肠道微生物组研究的元分析确定了疾病特异性和共同反应。Commun Nat。8, 1784(2017)。
Shah, m.s.等。利用基于序列的粪便微生物群落调查数据确定结直肠癌的复合生物标志物。肠道67, 882-891(2018)。
Pasolli, E., Truong, D. T., Malik, F., Waldron, L. & Segata, N.大型宏基因组数据集的机器学习元分析:工具和生物学见解。公共科学图书馆第一版。医学杂志。12, e1004977(2016)。
戴,Z.等。结直肠癌宏基因组的多队列分析确定了人群中改变的细菌和通用的细菌标记物。微生物组6, 70(2018)。
Maier, L.等。非抗生素药物对人体肠道细菌的广泛影响。自然555, 623-628(2018)。
米兰尼斯等人。用mOTUs2进行微生物丰度、活性和种群基因组分析。Commun Nat。10, 1014(2019)。
Kultima, J. R.等。MOCAT2:宏基因组组装、注释和分析框架。生物信息学32, 2520-2523(2016)。
霍桑,T.等人。一个用于条件推理的乐高系统。点。统计。60, 257-263(2006)。
Mandal, S.等人。微生物组成分析:研究微生物组成的一种新方法。活细胞。生态。健康说。26, 27663(2015)。
Tjalsma, H., Boleij, A., Marchesi, J. R. & Dutilh, B. E.结直肠癌的细菌司机-乘客模型:超出通常的怀疑。Nat. Rev.微生物。10, 575-582(2012)。
托马斯,a.m.等。大肠癌数据集的宏基因组分析确定了跨队列微生物诊断特征和与胆碱降解的联系。Nat,地中海。https://doi.org/10.1038/s41591-019-0405-7(2019)。
Huerta-Cepas, J. et al.eggNOG 4.5:一个具有改进的真核、原核和病毒序列功能注释的分层矫形学框架。核酸测定。44, d286-d293(2016)。
Kanehisa等人。数据、信息、知识和原理:回到KEGG的新陈代谢。核酸测定。42, d199-d205(2014)。
李,J.等。人类肠道微生物组内参基因的综合目录。生物科技Nat。》。32, 834-841(2014)。
Vieira-Silva, S.等人。物种-功能关系塑造了人类肠道微生物群的生态特性。Microbiol Nat。1, 16088(2016)。
平山等人。用毛细管电泳飞行时间质谱分析结肠癌和胃癌微环境的定量代谢组谱。癌症Res。69, 4918-4925(2009)。
Denkert, C.等人。人结肠癌的代谢物分析:TCA循环和氨基酸周转的失调。摩尔。癌症7, 72(2008)。
马,M., Koh, P. K., Cheah, P. Y. & Chan E. C.用二维气相色谱质谱分析人类结直肠癌的代谢分型。分析的Bioanal。化学。403, 483-493(2012)。
Weir, t.l.等。结直肠癌患者与健康成人粪便微生物组和代谢组的差异《公共科学图书馆•综合》8, e70803(2013)。
戈德特,j.j.等人。粪便代谢组学:检测性能及其与结直肠癌的相关性。致癌作用35, 2089-2096(2014)。
红肉和结直肠癌。肿瘤防治杂志。牧师。9288(2015)。
饮食、营养、体育活动与癌症:全球视角。一个第三次专家报告摘要(世界癌症研究基金会,2018)。
Dutilh, B. E., Backus, L., van Hijum, S. A. & Tjalsma, H.筛选metatranscriptomes的毒素基因作为人类结直肠癌的功能驱动因素。Pract最好。中国>,杂志。27, 85-99(2013)。
西尔斯,C. L. & Garrett, W. S.微生物,微生物群与结肠癌。细胞宿主微生物15, 317-328(2014)。
Ridlon, J. M., Harris, S. C., Bhowmik, S., Kang, D. J. & Hylemon, P. B.肠道细菌胆盐生物转化的后果。肠道微生物7, 22-39(2016)。
Yoshimoto, S.等人。肥胖诱导的肠道微生物代谢产物通过衰老分泌体促进肝癌的发生。自然499, 97-101(2013)。
Ajouz, H., Mukherji, D. & Shamseddine, A.次生胆汁酸:一种未被认识到的结肠癌病因。世界J.外科医生。12, 164(2014)。
Boleij, A.等。的脆弱拟杆菌结直肠癌患者的结肠黏膜普遍存在毒素基因。中国。感染。说。60, 208-215(2015)。
吴,S.等。人类结肠共生体通过激活辅助性17型T细胞反应促进结肠肿瘤的发生。Nat,地中海。15, 1016-1022(2009)。
Dejea, C. M.等。家族性腺瘤性息肉病患者的结肠生物膜中含有致瘤细菌。科学359, 592-597(2018)。
李德龙,康德杰,海莱蒙,彭波。一种胆汁酸诱导的7α-去羟基操纵子的分离与表征hylemonae梭状芽胞杆菌TN271。厌氧生物16, 137-146(2010)。
马隆尼,怀特,W. B.和Hylemon, P. B.一种胆汁酸诱导操纵子的克隆和测序真细菌sp.菌株VPI 12708。j . Bacteriol。172, 7011-7019(1990)。
Ocvirk, S. & O 'Keefe, S. J. D.胆汁酸对结直肠癌风险的影响:饮食-肠道微生物群相互作用介导的潜在机制。咕咕叫。减轻。代表。6, 315-322(2017)。
Gevers, D.等人。新发克罗恩病中未治疗的微生物群。细胞宿主微生物15, 382-392(2014)。
维也纳,S.等人。炎症性肠病中的结肠癌:近期趋势、问题和答案。杂志。中国。医学杂志。33, s190-s201(2009)。
鲁宾斯坦,m.r.等。梭菌属nucleatum通过FadA粘连蛋白调节E-cadherin/β-catenin信号通路促进结直肠癌的发生。细胞宿主微生物14, 195-206(2013)。
Kostic, a.d.等人。梭菌属nucleatum增强肠道肿瘤发生和调节肿瘤免疫微环境。细胞宿主微生物14, 207-215(2013)。
亚瑟,J. C.等。肠道炎症的目标是微生物群的致癌活性。科学338, 120-123(2012)。
饮食与胆汁酸的排泄。癌症Res。41, 3766-3768(1981)。
Ogino, S.等。精准医疗的外源性、内源性、肿瘤和免疫因素综合分析。肠道67, 1168-1180(2018)。
Ogino, S., Chan, A. T., Fuchs, C. S. & Giovannucci, E.结直肠肿瘤的分子病理流行病学:一个新兴的跨学科和跨学科领域。肠道60, 397-411(2011)。
Hannigan, g.d., Duhaime, m.b., Ruffin, m.t. 4th, Koumpouras, c.c. & Schloss, p.d.结直肠癌病毒群的诊断潜力和相互作用动力学。MBio9, e02248-18(2018)。
zur Hausen, H.红肉消费和癌症:怀疑牛感染因素参与结直肠癌的原因。Int。j .癌症130, 2475-2483(2012)。
Shkoporov, A. N.等。人类粪便噬菌体宏基因组分析的可重复性方案。微生物组6, 68(2018)。
Böhm, J.等。发现新型血浆蛋白作为结直肠癌患者中线切口后切口疝发展的生物标志物:ColoCare研究手术161, 808-817(2017)。
利森菲尔德,d.b.等人。代谢组学和转录组学鉴定结直肠癌患者内脏和皮下脂肪组织之间的途径差异:ColoCare研究点。j .中国。减轻。102, 433-443(2015)。
痘,c.p.等。全国结直肠癌结肠镜筛查项目的疗效。胃肠病学142, 1460 - 1467。e2(2012)。
Furet, J. P.等。利用实时定量PCR对人类和农场动物粪便微生物群的比较评估。《。生态。68, 351-362(2009)。
门德,D. R.等。原核生物物种的准确和普遍的描述。Nat方法。10, 881-884(2013)。
Sunagawa等人。利用通用系统发育标记基因进行宏基因组物种分析。Nat方法。10, 1196-1199(2013)。
李,H. & Durbin R.快速和准确的短读对齐与Burrows-Wheeler变换。生物信息学25, 1754-1760(2009)。
通过LASSO的回归收缩和选择。J. R.统计社B58, 267-288(1996)。
史玛洛夫斯基,P.弗莱曼,D.和克雷默,S.监督特征选择的陷阱。生物信息学26, 440-443(2010)。
Benjamini, Y. & Hochberg, Y.控制错误发现率:多重测试的一种实用而强大的方法。J. R.统计社B57, 289-300(1995)。
Robin, X.等人pROC:用于R和S+分析和比较ROC曲线的开源包。BMC生物信息学12, 77(2011)。
Oksanen, J. et al. vegan:社区生态包(综合R档案网络,2018)。
考斯泰,P. I.,泽勒,G., Sunagawa, S. &博克,P.一个公平的比较。Nat方法。11, 359(2014)。
哈斯蒂,T.,蒂布谢拉尼,R. & J.弗里德曼。统计学习的要素:数据挖掘、推断和预测(施普林格,2009)。
彭海华,龙飞,丁志刚。基于互信息的特征选择:最大依赖、最大相关和最小冗余的准则。IEEE反式。模式肛门。马赫。智能。27, 1226-1238(2005)。
Edgar, r.c. MUSCLE:多序列比对,高精度,高通量。核酸测定。32, 1792-1797(2004)。
Altschul, s.f.等人。gapping BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸测定。25, 3389-3402(1997)。
艾迪,s。r。加速剖面HMM搜索。公共科学图书馆第一版。医学杂志。7, e1002195(2011)。
Rice, P., Longden, I. & Bleasby, A. EMBOSS:欧洲分子生物学开放软件套件。趋势麝猫。16, 276-277(2000)。
卡波拉索,J. G.等。16S rRNA多样性的全球模式在每个样本数百万序列的深度。国家科学院学报美国108, 4516-4522(2011)。
确认
我们感谢Zeller, Bork和Arumugam小组的成员,他们激发了讨论。此外,我们感谢袁光平和EMBL信息技术核心设施对高性能计算的支持,以及EMBL基因组学核心设施对测序的支持。我们也非常感谢EMBL统计数据分析中心B. Klaus提供的建议。我们感谢EMBL、德国癌症研究中心、亨茨曼癌症基金会、国家癌症研究所的校内研究计划、ETH Zürich以及以下外部来源的资助:欧洲研究理事会(CancerBiome资助号:ERC-2010-AdG_20100317到p.b., Microbios批准号。ERC-AdG-669830到P.B, Meta-PG批准号。ERC-2016-STG-716575 to N.S.);诺和诺德基金会(no。NNF10CC1016515 to M.A.);由诺和诺德基金会和TARGET研究计划支持的丹麦糖尿病学会(丹麦战略研究委员会no。 0603-00484B to M.A.); the Matthias-Lackas Foundation (to C.M.U.); the National Cancer Institute (grant nos. R01 CA189184, R01 CA207371, U01 CA206110, and P30 CA042014 to C.M.U.); the Federal Ministry of Education and Research (BMBF; the de.NBI network no. 031A537B to P.B. and the ERA-NET TRANSCAN project no. 01KT1503 to C.M.U.); the Helmut Horten Foundation (to S.Sunagawa); and the Fundação de Amparo à Pesquisa do Estado de São Paulo (grant no. 16/23527-2 to A.M.T.). For the Italy validation cohorts, funding was provided by the Lega Italiana per La Lotta contro i Tumori. For the Japan validation cohort, funding was provided to T.Y. and S.Y. by the National Cancer Center Research and Development Fund (grant nos. 25-A-4,28-A-4, and 29-A-6); Practical Research Project for Rare/Intractable Diseases from the Japan Agency for Medical Research and Development (grant no. JP18ek0109187); Japan Science and Technology Agency-PRESTO (grant no. JPMJPR1507); Japan Society for the Promotion of Science KAKENHI (grant nos. 16J10135, 142558, and 221S0002); Joint Research Project of the Institute of Medical Science, University of Tokyo; and the Takeda Science and Suzuken Memorial Foundations.
作者信息
作者及隶属关系
贡献
g.z., m.a.和P.B.构想并监督了这项研究。p.s.k., n.h., C.M.U, h.b., e.v.和R.S.招募了参与者并收集了样本。e.k., A.Y.V, s.s anagawa和P.B.生成了宏基因组数据。a.m., p.t.p., j.s.f., a.p., s.s anagawa, l.p.c., g.z.和M.A.开发了宏基因组分析工作流程和/或进行了分类和功能分析。j.w., g.z., k.z., p.t.p., a.k., m.a.和N.S.进行了统计分析和/或开发了统计分析工作流程。E.K.和R.P.设计并执行了验证实验。A.M.T点。,S.G个终身制,伺服电动机,h, S.Shiba, t, S.Y, T.Y, L.W,蒋振声,含硝酸钠提供额外的验证数据。j.w., g.z., m.a., p.t.p.和P.B.设计了这些人物。g.z., j.w., m.a.和P.B.撰写了手稿,p.t.p., a.m., s.s sunagawa, l.p.c., e.k., A.Y.V, e.v., r.s., p.s.k., h.b., e.n., N.S.和L.W.所有作者讨论并批准了手稿。
相应的作者
道德声明
相互竞争的利益
P.B、G.Z、A.Y.V和s.s anagawa被命名为一项专利的发明者(EP2955232A1:基于分析肠道微生物组诊断腺瘤和/或结直肠癌(CRC)的方法)。
额外的信息
出版商的注意:施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
扩展数据
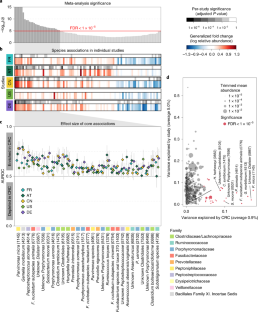
扩展数据图2研究异质性对alpha和beta多样性有很强的影响。
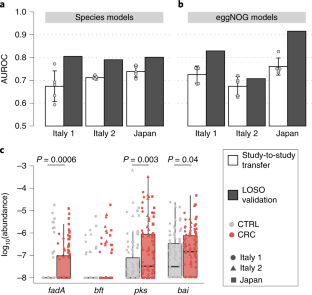
一个,用香农指数计算所有肠道微生物物种的Alpha多样性(n= 849),参考mOTUs (n= 246),以及元motus (n= 603)。P值计算使用双面Wilcoxon检验,而整体P值(顶部)的计算采用双面阻塞Wilcoxon检验(n= 575个独立观测值;看到方法).面板下方的方差分析f -统计量使用R函数“aov”计算。b,基于Bray-Curtis距离的所有五个纳入研究样本的主坐标分析;该研究用颜色编码,疾病状态(CRC与CTRL)用填充/未填充的圆圈表示。侧面和下方的箱形图分别显示了投射到前两个主要坐标上的样本,这些坐标由研究和疾病状况分解。P数值的计算采用疾病状态的双侧Wilcoxon检验和研究用的Kruskal-Wallis检验(n= 575个独立观测)。对于所有箱形图,框表示IQR,中位数为粗黑线,胡须延伸到1.5倍IQR内的最极端点。国家代码如图所示。1 b.
广义折叠变化扩展了已建立的(基于中值的)折叠变化,以在稀疏的微生物组数据中提供更高的分辨率。
一个的对数相对丰度拟杆菌dorei / vulgatus,微小微单胞菌,f . nucleatum亚种animalis -一个高流行种和两个低流行种的例子显示为CTRL组和CRC组的群图。粗竖线表示不同组的中位数,黑色水平线表示两组中位数的差异,对应经典(基于中位数的)折叠变化。自f . nucleatum亚种animalis在超过50%的癌症病例中未检测到,CTRL和CRC中位数之间没有差异;因此,折叠变化为0。下面一行显示了相同的数据,但不是只有中位数(或第50百分位数),9个分位数范围从10%到90%由较细的垂直线显示。广义折叠变化再次由水平黑线表示,计算为两组中相应分位数之间差异的平均值。对于稀疏数据(例如,f . nucleatum), 70、80和90%分位数的差异导致广义折叠变化大于0。b中位数折叠变化与新开发的所有微生物物种的广义折叠变化进行对比。(微生物CRC标记物种的核心集以橙色突出显示。)边缘直方图显示了折叠变化和广义折叠变化的分布。c,显示折叠变化和广义折叠变化与ROC曲线下面积(AUROC)或CRC和CTRL之间患病率变化的关系的散点图,并在左上角添加了斯皮尔曼秩相关性(rho);广义折叠变化提供了更高的分辨率(在0附近分布更广),并且与非参数AUROC效应量测量以及流行度偏移有更好的相关性,后者捕捉了CRC宏基因组相对于CTRL宏基因组中一个物种的流行度差异。
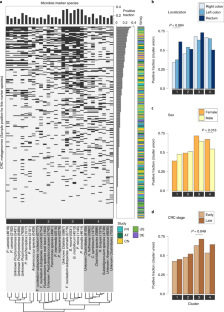
基于CRC宏基因组的共现性,CRC富集微生物种的核心集可分为四个簇。
一个,热图显示杰卡德指数(通过比较标记阳性样本计算;看到方法)的核心微生物标记物种,只以结直肠癌个案计算。聚类使用R函数“hclust”中实现的Ward算法进行。插图显示了每个类群内和背景的Jaccard相似性的分布(不属于同一类群的物种之间的所有相似性)。b,c, Barplots显示CRC样本中标记物种聚类(定义为阳性标记物种的联合)阳性的比例,根据BMI (b)及年龄(c)(见图。两个罪犯对于其他患者亚组)。使用Cochran-Mantel-Haenszel测试阻塞“研究”和“结肠镜检查”来测试CRC亚群和标记物种簇之间关联的意义。(没有检测到显著的关联。)d对于具有基因组参考的核心微生物物种集,超氧化物歧化酶、过氧化物酶和过氧化氢酶的存在(红色)或不存在(白色)显示为热图。酶的存在是通过检查参考项目的蛋白质注释(见NCBI BioProject ID)来确定的http://progenomes.embl.de/.
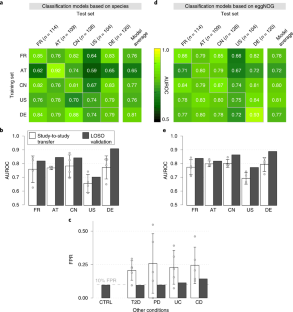
扩展数据图6 LOSO LASSO逻辑回归模型的系数与个别研究训练模型的比较。
一个,在单个研究(颜色编码)上训练的LASSO交叉验证模型的平均系数(特征权重)被绘制到每个物种特征的单特征AUROC上。横线突出显示了在交叉验证中超过50%的模型中选择的微生物物种,并且在至少10%的交叉验证模型中占绝对模型权重的10%以上(至少一项研究如此)。b类似地,b显示了在LOSO设置中训练的模型的相同结果(参见方法).颜色表示哪个研究被排除在训练集之外(并用于验证)。LOSO模型的权重分布在更多的物种中;因此,一般来说,如果在至少10%的交叉验证模型中,较低的物种的权重解释了绝对模型的2.5%以上,并且在交叉验证中超过50%的模型中选中了它们,那么它们就会被水平线突出显示。c,插图显示了所有交叉验证模型中非零系数的数量分布。d,柱状高度分别表示每个研究或遗漏研究的平均模型之间共享的非零系数的数量。e,交叉验证单研究模型的研究间差异(计算为平均模型中单个物种的模型权重之间的所有成对差异的中位数)与LOSO模型的相同测量值相对照。交叉验证模型中研究与研究之间差异超过0.02的物种被突出显示和注释,与LOSO模型相比,单个研究训练的模型之间的变异性要大得多。国家代码如图所示。1 b.
图7 LOSO模型对预测偏差的分析。
一个为了检验物种和基因家族级别的分类模型是否混淆,即偏向于某些患者亚组,LOSO模型的预测分数被分解为每个临床参数的层(例如,性别为女性和男性)。对每个变量的预测偏倚进行Wilcoxon(性别和BMI)或Kruskal-Wallis(所有其他)检验,同时将阻塞作为混杂因素进行研究。方框表示IQR,中间值为水平黑线,晶须延伸到1.5倍IQR内的最极端点。仅CRC期预测评分有显著差异。这种阶段偏差在基因家族中比在物种模型中更为明显。b,为了进一步检查CRC阶段偏差,barplot显示了对应于总体10% FPR的真阳性率(另见图)。3 c),在物种和基因家族模型中,对晚期CRC的分类敏感性均略高。
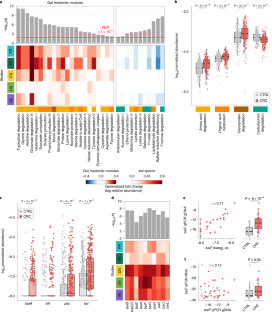
扩展数据图9白宏基因组中的基因。
假定的白在宏基因组IGC中鉴定的基因在宏基因组中通过共丰度聚类来推断基因组连锁(见方法)来推断操纵子的完整性和起源的种类。一个对于每个由此产生的假定胆汁酸转换基因簇,平均相对丰度与已知胆汁酸转换基因的全球比对所获得的蛋白质相同的平均百分比进行了绘制c . scindens而且c . hylemonae(见方法).完备性,也就是11个不同的白基因功能在每个簇中表示,每个簇内相对丰度的平均基因-基因相关性分别由点大小和颜色编码(见图例)。4个蛋白簇的平均蛋白同源性> 75%为已知白包含操纵子的基因组被包括在后续的分析中,并用最高度相关的mOTU标记(见b).b,基因簇丰度与相关性最高的物种(在对数空间中)的相对丰度之间的Pearson相关性由中鉴定的四个基因簇的条形高度给出一个.相关性最高的物种用深灰色突出显示(见中基因簇的标记)一个).c,对数转换的所有丰度白基因和四个物种的鉴定b显示为ctrl(灰色)和CRC情况(红色)的箱线图。评估CRC和CTRLs之间差异的意义(使用用于“研究”和“结肠镜检查”的Wilcoxon检验)表明聚集的宏基因组的CRC富集更为显著白基因丰度高于它们所属的梭状芽孢杆菌个体。d, ROC曲线为qPCR定量baiF基因在德国研究的一个样本子集的基因组DNA(见方法和无花果。4 e).
源数据
图1 .来源数据
统计来源数据
图2 .来源数据
统计来源数据
图3 .源数据
统计来源数据
图4 .来源数据
统计来源数据
图5 .来源数据
统计来源数据
图1 .源数据扩展数据
统计来源数据
图2 .扩展数据
统计来源数据
图3 .扩展数据
统计来源数据
图4 .扩展数据
统计来源数据
图5 .源数据扩展数据
统计来源数据
图6 .源数据扩展数据
统计来源数据
图7 .源数据扩展数据
统计来源数据
图8 .源数据扩展数据
统计来源数据
图9 .源数据扩展数据
统计来源数据
图10 .源数据扩展数据
统计来源数据
权利和权限
关于本文
引用本文
韦贝尔,J.,派尔,p.t.,卡尔塔尔,E.。et al。粪便宏基因组的元分析揭示了结直肠癌特有的全球微生物特征。Nat地中海25, 679-689(2019)。https://doi.org/10.1038/s41591-019-0406-6
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41591-019-0406-6
进一步的阅读
人类肠道微生物组中硫代谢基因的多样性和分布及其与结直肠癌的关系
微生物组(2022)
热量限制对肠道微生物群的影响与免疫衰老有关
微生物组(2022)
建立宏基因组学机器学习可解释模型,以理解从腺瘤到结直肠癌的转变
科学报告(2022)
大规模微生物组数据集成可实现可靠的生物标志物识别
自然计算科学(2022)
只需添加数据:用于知识发现和特征选择的自动预测建模
npj精密肿瘤学(2022)
