文摘
我们旨在描述微生物群的胃粘膜肠类型的癌症进展。研究包括五个病人每个non-atrophic胃炎(唠叨),肠上皮化生(IM)和肠胃型胃癌(GC)。胃组织获得和DNA提取使用微阵列G3 PhyloChip微生物群分析。细菌多样性包括8 - 57和稳步下降从唠叨到IM GC (p = 0.004)。一个重要微生物群之间的差异观察唠叨和基于Unifrac-presence GC /没有和weighted-Unifrac-abundance指标283类群(p < 0.05)。HC-AN分析基于存在/缺乏238类群表明,GC和分组的唠叨,而我与两个重叠。协同分析基于weighted-Unifrac距离给出大量的44个分类单元显示的意义类别显示重要的微生物群唠叨和GC之间的分离。这项研究是第一个显示胃微生物群逐步转变文件从唠叨到IM GC。
介绍
幽门螺旋杆菌感染被认为是远端胃癌的主要危险因素(GC),尽管只有一小部分感染者(< 3%)开发GC,暗示其他因素也扮演了一定的角色1。病理GC的研究显示,这是一个inflammation-driven疾病和各种影响因素的黏膜免疫反应可能参与这多因子的模型2。因此,宿主基因、环境和幽门螺旋杆菌基因的作用。炎症相关的基因多态性IL-1β511 T, Interferon-γR1 -56 c / T,或已报告通常与GC3;减少食用富含抗氧化剂的食物(蔬菜、水果)、吸烟、酒精或肥胖也会增加风险4。大部分的风险因素可能有一个以上的角色在粘膜炎症和免疫反应,从而调节组织损伤的驱动力和胃癌的发展。
远端GC可能是两种类型,肠道和扩散,每个不同的发展路线。对于肠道类型,科雷亚州最初预测的发展模式胃炎症可能会失控,导致粘膜萎缩和hypochlorydria,反过来又增加了患肠上皮化生,发育不良,最后肠道类型的GC5。虽然对扩散GC的发展,它被接受幽门螺旋杆菌和炎症可能也扮演了一定的角色6。
胃微生物群也可以调节炎症和免疫反应的强度和类型在胃粘膜。研究微生物群胃的稀缺,一项研究发现,人类的胃是由一个复杂的微生物群包括殖民主要变形菌门、厚壁菌门、放线菌和梭菌属类群和显示明显差异与口腔微生物群描述和食道7。研究还表明,阳性的患者幽门螺旋杆菌文化显示出显著增加变形菌门的殖民和放线菌的减少。最初试图比较微生物群在GC和消化不良的患者报告在细菌社区没有显著差异,尽管作者观察到链球菌,乳酸菌,韦永氏球菌属和普氏菌主导GC患者8。
我们知道幽门螺旋杆菌感染通常不改变酸胃粘膜的屏障,除非语料库的不受管制的炎症反应,导致萎缩和hypochlorydria9。变更的酸度可能会导致一个更宽松的环境与其他细菌殖民化8。我们假设这种微生物群的转变增加了炎症反应已经到位,增加更多的萎缩和肠上皮化生的风险,从而增加风险开发GC。在这个工作我们旨在研究患者胃胃组织的微生物群的变化进步导致胃癌组织学阶段,从non-atrophic胃炎(唠叨),肠上皮化生(IM)和GC。
结果
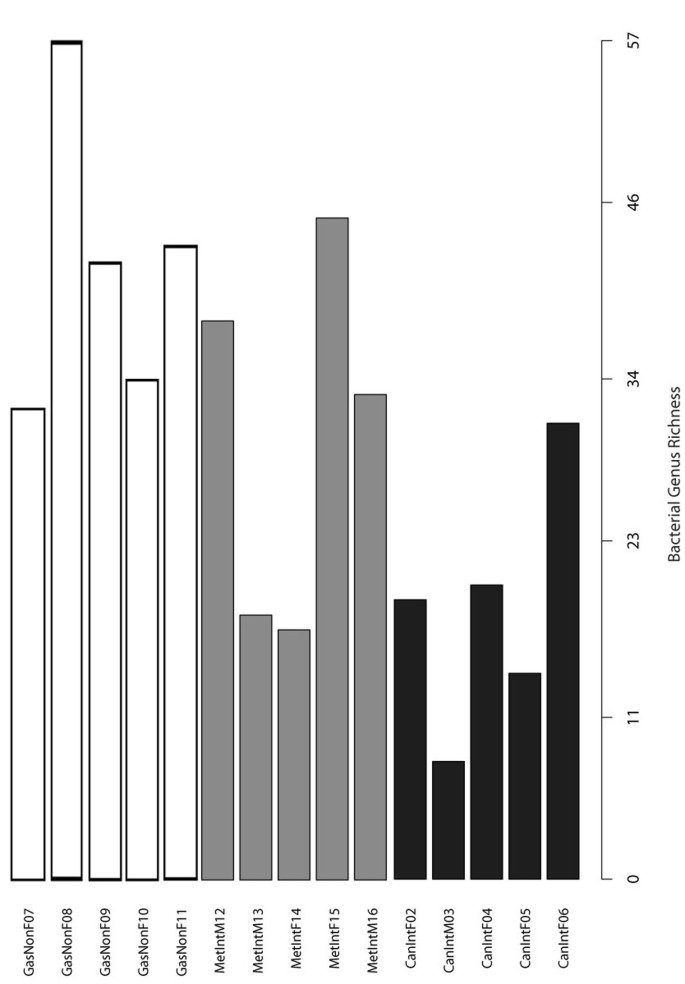
胃微生物群多样性较低,与9家庭代表> 50%的辣子鸡
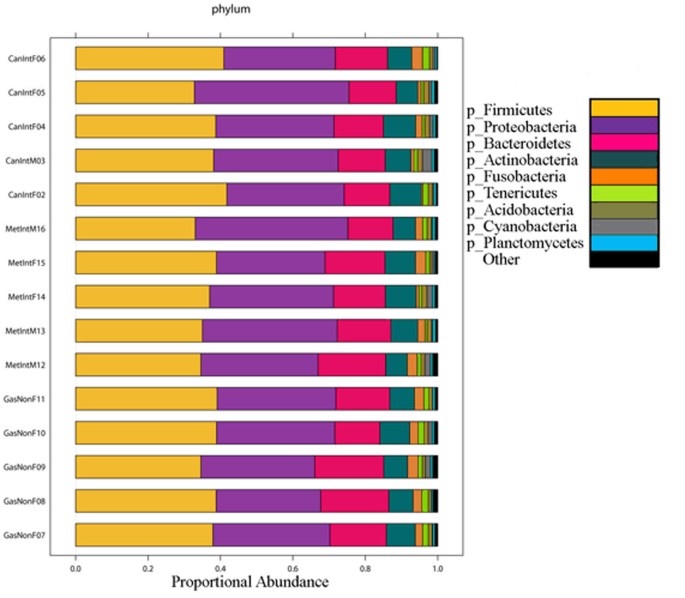
各样本细菌属多样性很低,范围从8日在GC M03病人,在唠叨病人F08 (57图1)。多样性显著不同的唠叨患者与患者之间GC(双尾heteroscadastic t检验,p = 0.004),但不唠叨和IM之间;然而,我们观察到多样性减少的趋势从唠叨到IM GC (图1)。辣子鸡的比例为每门中描述图2显示两个类群,厚壁菌门和变形菌门代表近70%的门在所有样本。我们还发现,9代表家庭平均高达55.6%的每个示例的辣子鸡,Lachnospiraceae和链球菌科代表超过20%的家庭来自疾病三个组的患者。
整个微生物群资料显示分离GC和唠叨,但不是IM和GC之间或IM和唠叨
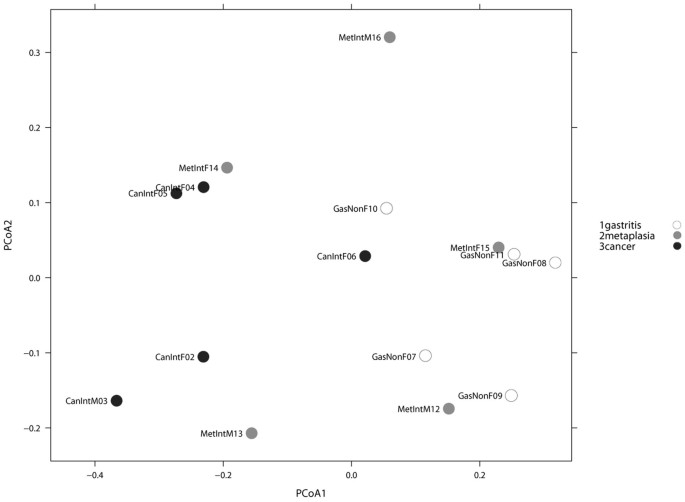
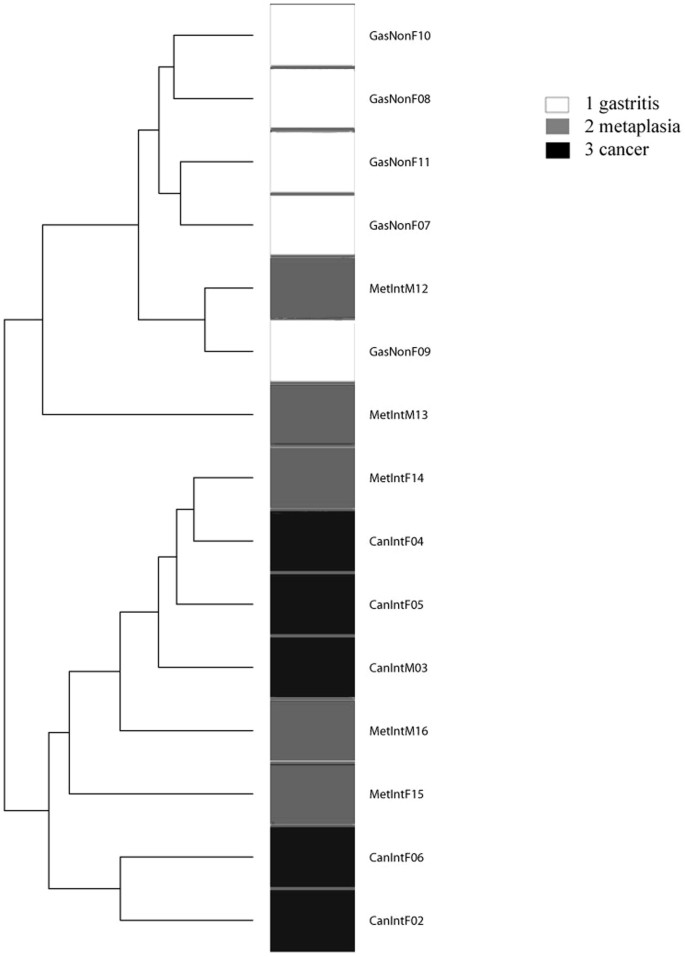
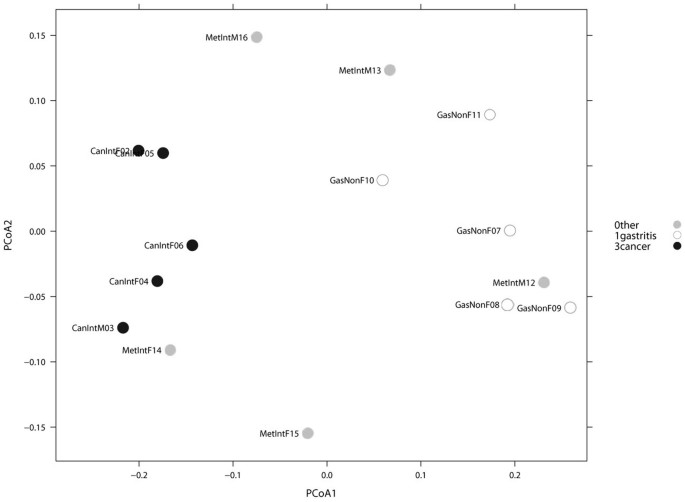
我们首先分析了可能的性别和年龄对微生物群组成的影响由于存在/ 283类群存在于至少一个样品。主坐标分析(PCoA)包括所有15例和基于未加权的Unifrac距离显示没有明显的微生物群基于年龄或性别差异(p值> 0.50,阿多尼斯测试)。类似PCoA分析比较唠叨和IM vs GC,鉴于存在/缺乏283类群存在于至少一个样品做了(图3)。阿多尼斯测试了假定值为0.026,表明一个重要的微生物群的区别疾病组的至少一个人。此外,二进制指标分析显示GC分离微生物群从唠叨,而微生物群的IM不是分开GC或唠叨组。进一步证实了这些发现,PCoA比较唠叨vs GC和阿多尼斯测试完成了假定值为0.005,表明一个重要的微生物群的区别这两个组。此外,对比我唠叨(p = 0.42)和IM和GC之间(p = 0.129)组没有区别。分层聚类分析(HC-AN)基于存在/缺乏283类群透露两个集群,一个只包含GC和IM,另一唠叨和IM,与一个GC可能异常(例CanIntF06)。
我们接下来做PCoA分析基于283年wUnifrac样本之间的距离得到大量的分类单元出现在至少一个样品。而丰度指标分析显示没有明显的分离微生物群根据疾病组,有/没有度量分析显示唠叨和GC之间的显著差异(p = 0.03,阿多尼斯测试)。同样的分析揭示了唠叨和IM之间没有显著差异和GC和IM之间;尽管GC和IM显示重要的微生物群的区别和NAG相比(p = 0.048,阿多尼斯测试)。
分析基于与重要类群丰度差异跨组织证实了微生物群分离GC和唠叨和重叠的IM
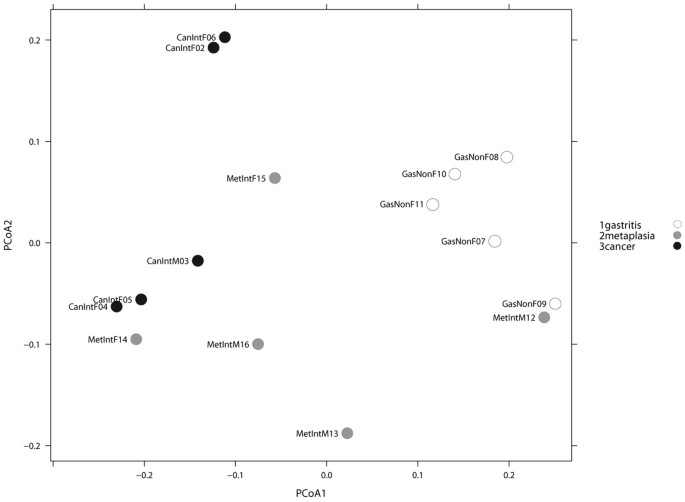
我们执行一个参数韦尔奇测试来识别辣子鸡,明显增加或减少在至少一种疾病组和发现27个类群有大量跨组的区别。PCoA加权距离Unifrac团体基于27个类群,显示所有三组之间没有明显的分离微生物群(NAG和IM vs GC)。然而,当考虑到只有唠叨和GC的病人,一个完整的分离的微生物群观察PCoA1轴(图4)。HC-AN分析基于同一类群27日表明,唠叨和GC集群分开,而我例重叠的两个唠叨和GC组(图5)。
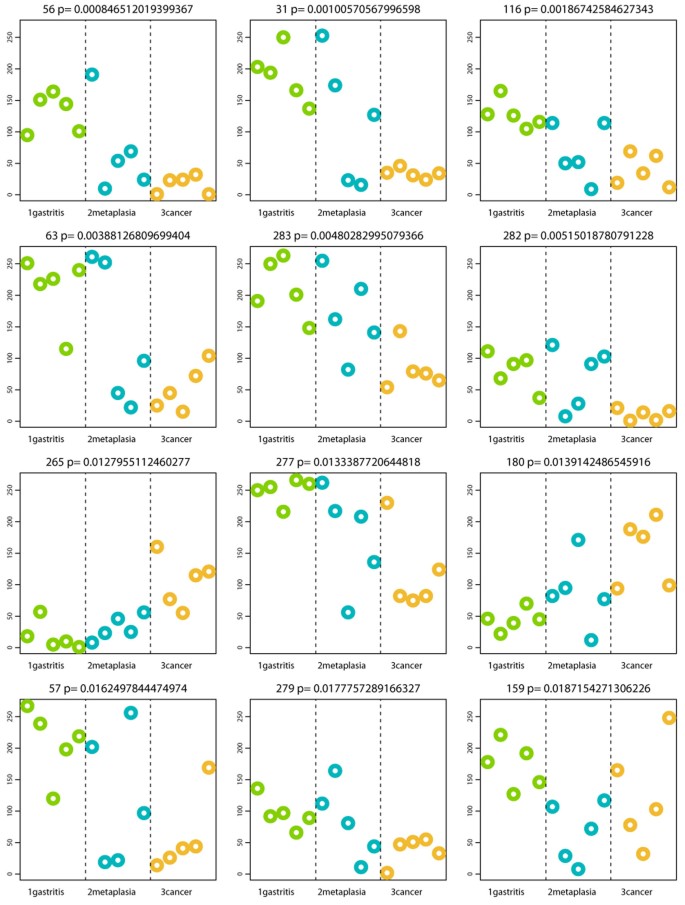
从27个类群大量不同,不同的团体,我们选择了12更重要的假定值(表1在每个疾病组)和策划丰富(图6)。结果表明,5这些前12显示下降趋势从唠叨我GC (2 TM7, 2Porphyromonas和1奈瑟氏菌属),而从唠叨2显著增加IM GC (乳酸菌coleohominis和Lachnospiraceae)。
分析基于与重要类群丰富唠叨和GC之间的差异确认这些团体的重要微生物群分离
进一步记录观察到的唠叨和GC之间的差异,韦尔奇参数测试是为了寻找辣子鸡,显著增加或减少这两组之间。测试确定了44个辣子鸡p值< 0.05,与这些PCoA加权Unifrac距离分析显示明显分离的微生物群的唠叨和GC (图7)。与上述44 HC-AN辣子鸡也显示单独的集群的唠叨和GC(结果未显示)。假定值最低的一个概要文件的前12显示9的12显示唠叨患者显著增加,而乳酸菌coleohominis和Lachnospiraceae在GC患者(更丰富表2)。
讨论
在这项研究中我们测试的假设患者胃粘膜的变化的微生物群唠叨IM患者和患者GC。为了测试这个我们使用代G3 PhyloChip™,增加敏感度低丰度的细菌10。这是特别相关的研究胃样品细菌殖民化的微环境的限制。事实上,我们发现低微生物群的多样性,共有12门和283辣子鸡检测到,而属多样性变化在所有患者从8到57。我们与以前的研究结果没有可比性,因为技术和数量的差异;不过,协议中存在低数量的发现门和厚壁菌门的优势,变形菌门、拟杆菌、放线菌在胃微生物群7,11,12。然而,使用代3 PhyloChip我们能够发现更多的辣子鸡和更高的多样性在属水平。因此,据报道,在胃粘膜最丰富的家庭是链球菌科和Prevotellaceae8,11,这与我们的结果Lachnospiraceae是最丰富的,代表近20%的微生物群在所有患者。Lachnospiraceae家族是由严格的厌氧菌,其中一个新的属最近所描述的嘴13。在小肠,这个家庭的成员已报告产生一种保护作用梭状芽孢杆菌殖民用抗生素微生物群后修改14。此外,有人建议Lachnospiraceae的家人可能会减少肠道致癌作用15。
我们还发现在几个病人的物种嗜血杆菌56和ratti韦永氏球菌属已被报道,以前只在尿道16,17和成员的家庭Desulfobacteraceae Acidobacteriaceae(数据未显示),已被描述为自由生活的细菌18,19。这些细菌丰度低的存在在一些情况下表明他们不是污染物及其识别可能是因为G3 PhyloChip的敏感性增强10。结果从这些和其他的研究表明,胃粘膜可能与细菌从没被殖民在胃里,甚至在人类身上7。
低多样性在这项研究中观察到与最近的一份报告中使用检测到的G2 PhyloChip更高的多样性与胃炎成年人20.,多达44门,其中许多都是在非常低的频率,尽管协议的主要类群。差异与我们的研究可能会解释说,部分原因在于人口的差异,因为他们从委内瑞拉亚马逊印第安人取样与特定血统和社会经济和卫生条件差。
关于微生物群的变化,我们发现的证据逐渐改变患者唠叨IM GC,细菌多样性显示趋势减少从唠叨我唠叨的GC和显著高于在GC的病人。这些结果表明,胃粘膜的变化作为移动preneoplasia和癌症患者呈现环境更加严格的细菌生长,相反别人猜测21。黏蛋白是由正常的上皮细胞在胃肠道和作为一个保护屏障和微生物群作为殖民的专业化22。一个健康的胃粘膜产生MUC5AC MUC6黏蛋白23胃微生物群,殖民;但当正常黏膜萎缩或肿瘤前期病变所取代的黏蛋白化生的肠道MUC2的表达变化24我们建议这使粘膜不适合殖民的正常微生物群。
当整个微生物群剖面进行了研究,分类分析的基础上,存在/缺失或丰富,显示明显分离的GC唠叨,而我没有分开唠叨或GC。集群分析还显示为唠叨两个独立的组,与GC和GC与我一起唠叨。这些研究结果建议改变从唠叨到GC,我作为一个中间群体。这个观察是有可能的,因为我们的研究包括IM病人,与以前的报告,GC只唠叨患者相比8。最近的研究在转基因hypergastrinemic小鼠模型发现感染幽门螺旋杆菌在肠道菌群引起的胃肠道上皮内肿瘤的发展25;甚至殖民与一个非常受限制的肠道菌群被发现足以诱发肿瘤26。这些观察表明,除了其他细菌的存在幽门螺旋杆菌胃癌的发展需要,尽管他们并没有真正帮助阐明是否增加或减少细菌多样性负责这佐剂效应。
特别感兴趣的是强调更丰富的类群之一GC比唠叨是属假单胞菌(数据未显示),这是相关的胃腺癌患者最近的一份报告发现特定的集成假单胞菌——DNA体细胞,5′utr和3′utr四个原癌基因的上调GC27。事实上,这是第一个研究记录bacterial-human体细胞横向基因转移的癌症患者的优势和我们的结果假单胞菌在GC会同意这一重要报告。
进一步选择最低的辣子鸡假定值显示6个类群明显减少趋势从唠叨到IM GC (2 TM7, 2Porphyromonas sp,奈瑟氏菌属sp和链球菌sinensis)和2有增加的趋势(乳酸菌coleohominis和Lachnospiraceae)。TM7是最近描述门出现在人类的口腔和肠道;和一小部分基因序列相似的基因中发现类的成员杆菌,梭状芽胞杆菌和Fusobacteria28。TM7编码IV型菌毛基因,被认为是一些病原菌的毒力因素,也可能在炎性疾病如克罗恩氏病中的作用29日和结肠炎30.。链球菌sinensis据报道在口腔吗31日和相关感染性心内膜炎32。另一方面,l . coleohominis是一个相对新颖的物种从阴道中恢复过来33和尿液的健康的女人34存在于任何疾病协会,但没有报告。Lachnospiracea家庭报道明显降低患者的炎性疾病35。因此,许多细菌群体中我们发现改变患者的胃粘膜GC已报告相关的炎症过程,可能可以想象在炎症反应的调控中发挥作用在胃壁与GC的发展有关。
一项研究报告没有区别胃GC之间的微生物群,消化不良的患者,尽管在某些种类的癌症患者链球菌,乳酸菌和韦永氏球菌属更普遍的8。本研究基于克隆、测序和限制片段长度多态性16 s rRNA,在敏感性和覆盖重要的局限性与最近开发的技术,包括G3 PhyloChip。不过,同意我们的结果,他们还占主导地位乳酸菌在GC。据我们所知,没有额外的报告描述微生物群患者的GC和研究是必要的学习更多关于胃微生物群在健康和疾病的作用。研究微生物群个体间在其他人体组织显示重要的多样性和不同人群之间的显著差异,即使在使用相同的样品处理技术研究平台36,37。我们需要解决的研究微生物群在胃里使用类似的方法,研究涉及到更大的群体和不同的人群。我们承认我们的研究的一个重要收回数量减少的病人,这允许我们发现疾病组之间的差异,但缺乏研究组织内其他变量的影响;例如,我们有少量的雄性的唠叨和GC组。这些问题应该通过学习解决更多的病例。
目前的研究是第一个显示胃癌患者胃微生物群的差异并提出逐步改变微生物群的轮廓从胃炎之间癌症病变。研究还表明,减少Porphyromonas,奈瑟氏菌属、TM7集团和美国的中国,以及增加l . coleohominis和Lachnospiraceae可能对胃癌的发展。这些类群的作用及其在胃癌发病机制仍有待研究。
方法
病人
我们包括肿瘤患者咨询和综合医院医疗中心SXXI, Instituto Mexicano del全民社会(IMSS)在墨西哥城。五个病人都有唠叨,IM和GC肠道类型选择的研究。,唠叨和IM病人我们选择从腔组织样本,而对于GC组与我们合作组织的病变,而4例扩展腔和语料库和一个眼底和语料库。这些患者的特征描述表3;病人的组织研究在身体质量指数没有显著差异或学校多年来,尽管有有限数量的年轻男性和唠叨患者比IM和GC。我们排除了免疫缺陷患者,糖尿病或其他慢性疾病和病人抗生素或质子泵抑制剂在过去三个月,或先前接受治疗的病人幽门螺旋杆菌根除。幽门螺旋杆菌诊断使用的酶联免疫吸附试验之前进行验证38。所有患者Mexican-mestizo中长期社会经济水平低。病人被告知研究和那些愿意参与签署了同意信。IMSS的IRB委员会批准了这项研究。
胃样品
唠叨和IM受到患者内镜活检腔和语料库,而患者GC受到肿瘤切除手术。样品的唠叨,IM或GC处理如下:一个活检或一小部分肿瘤病变组织学研究立即被放置在福尔马林和另一个组织部分放置在液态氮运输我们的实验室,活检和组织都存储在−70°C到测试。固定的组织与苏木精和伊红染色,染色和高碘酸修改。希夫/阿尔新蓝染色时上皮化生被怀疑。准备工作由一个有经验的病理学家检查和最终诊断使用之前报道的标准分配39,40,41。
DNA提取、PCR扩增16 s rRNA微阵列
15个患者的组织样本解冻使用QIAamp DNA和DNA提取简单的迷你包(试剂盒、希尔登,德国)。孤立冻干DNA被水化和量化使用PicroGreen工具包(生活技术,卡尔斯巴德,CA)。样品受到放大使用退化16 s rRNA基因引物27 f。1(5′-AGRGTTTGATCMTGGCTCAG-3′) and 1492R (5′-GGTTACCTTGTTACGACTT-3′) for bacteria and 4Fa and 1492R for archaea as described in Hazen et al.10。放大PCR产品集中,纯化,量化和500 ng放大产品的分散区间50 - 200个基点,然后生物素标记(Affymetrix WT双链DNA终端标签)杂交。
微阵列杂交
Phylochip一代3 (G3)使用微阵列杂交和样本运行第二个基因组,INC .(旧金山,CA)。这包括110万个DNA探针能够分类所有已知的细菌和古细菌的操作分类单元(辣子鸡)超过50000个分类单元使用59959的17个核苷酸探针147门,1123类,1219订单和1464的家庭,总共27938辣子鸡。根据制造商的指示进行了染色和扫描(Affymetrix)和之前描述的过程10。PhyloChip内置的控制微阵列杂交过程的所有步骤和探针强度的归一化值。荧光强度是编码为从0到65536的整数。杂交评分(HybScore)值在0到16000的范围和变化在1000年出的HybScore表明双重荧光强度增加。
减少数据和统计分析
类群过滤这些出现在至少一个的样品,或类群丰富的显著增加一个类别相比,交替类别。韦尔奇丰富滤波器的参数测试是用来计算假定值。此外,q值计算正确使用Benjamini-Hochberg过程假定值,控制错误发现率10。
类群被确认后纳入分析,使用的值为每个taxa-sample路口被定义在两个不同的方面。在第一种情况下,丰度指标是直接使用()。在第二种情况下,二进制创建指标(缺席,0;存在,1)(BT)。不成,heteroscadastic t假设不平等的方差是用来测试学生对微生物多样性的差异和层次总结两个样本的类别。
评估样本距离所有资料都inter-compared双向的方式来确定不同分数和存储在不同的距离矩阵。距离函数选择只允许类似的生物样品生产小不同的分数。Unifrac距离度量利用之间的系统发育辣子鸡之间的距离来确定不同的社区42。Unifrac用于存在/缺失,而加权Unifrac (WUnifrac)是用于大量数据。
二维任圣职和层次聚类样本的地图的形式创建系统树图以图形的方式总结inter-sample关系。创建系统树图,距离矩阵的样品都聚集分层次使用average-neighbor (HC-AN)方法。主坐标分析(PCoA)是一个方法用于可视化的二维配合策划样本之间的复杂关系。PCoA使用不同的值位置点相对于彼此。阿多尼斯的测试是用来评估统计上显著的差异。在这种随机化/蒙特卡洛排列测试,样品是随机重新分配不同的样本类别和between-category差异比较真实between-category差异。阿多尼斯直接利用样本距离矩阵。数据分析使用PhyCA-Stats™分析软件(第二基因组,旧金山,CA)。
引用
Bjorkholm B。福尔克,P。,Engstrand, L. & Nyren, O. Helicobacter pylori: resurrection of the cancer link. J Intern Med 253, 102–119, 1119 (2003).
艾莉森,c . c &费列罗,r . l .毒性的作用因素和宿主细胞识别信号的幽门螺杆菌和免疫反应的生成。未来Microbiol 5, 1233 - 1255, 10.2217 / fmb.10.84 (2010)。
Gianfagna F。,德Feo说,E。,van Duijn, C. M., Ricciardi, G. & Boccia, S. A systematic review of meta-analyses on gene polymorphisms and gastric cancer risk. Curr Genomics 9, 361–374, 10.2174/138920208785699544 (2008).
Tsugane, s & Sasazuki美国饮食和胃癌的风险:审查的流行病学证据。胃癌10,75 - 83,10.1007 / s10120 - 007 - 0420 - 0 (2007)。
科雷亚,p &霍顿,j .幽门螺杆菌的致癌作用。胃肠病学133年,659 - 672,10.1053 / j.gastro.2007.06.026 (2007)。
Uemura: et al。幽门螺杆菌感染和胃癌的发展。N地中海拉米345,784 - 789,10.1056 / NEJMoa001999 (2001)。
Bik, e . m . et al .细菌的分子分析微生物群在人类胃。103年《美国国家科学院刊,732 - 737,10.1073 / pnas上。0506655103 (2006)。
Dicksved, j . et al .胃的分子表征微生物群在胃癌患者和控制。J地中海Microbiol 58、509 - 516年的10.1099 / jmm.0.007302-0 (2009)。
Talley: J。,Ormand, J. E., Frie, C. A. & Zinsmeister, A. R. Stability of pH gradients in vivo across the stomach in Helicobacter pylori gastritis, dyspepsia and health. Am J Gastroenterol 87, 590–594 (1992).
海森,t . c . et al .深海石油羽丰富本土oil-degrading细菌。330年科学,204;10.1126 /科学。1195979 (2010)。
李x x et al .细菌微生物群分析在胃炎幽门螺杆菌感染或非甾体抗炎药物使用。e7985 PloS One 4日,10.1371 / journal.pone。0007985 (2009)。
安德森,a . f . et al。人类肠道微生物群的编码焦磷酸测序进行比较分析。e2836 PloS One 3日,10.1371 / journal.pone。0002836 (2008)。
Sizova, m . v . et al . Stomatobaculum longum将军11月,11月sp.,人类口腔的专性厌氧细菌。Int J系统另一个星球Microbiol 63年,1450 - 1456,10.1099 / ijs.0.042812-0 (2013)。
Reeves, a E。,Koenigsknecht, M. J., Bergin, I. L. & Young, V. B. Suppression of Clostridium difficile in the gastrointestinal tracts of germfree mice inoculated with a murine isolate from the family Lachnospiraceae. Infect Immun 80, 3786–3794, 10.1128/IAI.00647-12 (2012).
梅,V。,Colbert, L. H., Perkins, S. N., Schatzkin, A. & Hursting, S. D. Intestinal microbiota: a potential diet-responsive prevention target in ApcMin mice. Mol Carcinog 46, 42–48, 10.1002/mc.20233 (2007).
杜洛埃,e . B。,Denoyel, G. A., Boude, M. M., Boussant, G. & de Montclos, H. P. Distribution of Haemophilus influenzae and Haemophilus parainfluenzae biotypes isolated from the human genitourinary tract. Eur J Clin Microbiol Infect Dis 8, 951–955 (1989).
Marchandin h . et al。人类第一次分离的分子识别属于韦永氏球菌属ratti-Veillonella criceti集团基于16 s rDNA和dnaK基因测序。Res Microbiol 156年,603 - 607,10.1016 / j.resmic.2005.01.013 (2005)。
Kjeldsen k U。,Jakobsen, T. F., Glastrup, J. & Ingvorsen, K. Desulfosalsimonas propionicica gen. nov., sp. nov., a halophilic, sulfate-reducing member of the family Desulfobacteraceae isolated from a salt-lake sediment. Int J Syst Evol Microbiol 60, 1060–1065, 10.1099/ijs.0.014746-0 (2010).
杨。,Wen, X., Jin, H. & Wu, Q. Pyrosequencing investigation into the bacterial community in permafrost soils along the China-Russia Crude Oil Pipeline (CRCOP). PloS One 7, e52730, 10.1371/journal.pone.0052730 (2012).
Maldonado-Contreras, a . et al。人类胃细菌群落结构与幽门螺杆菌的地位。ISME J 5, 574 - 579, 10.1038 / ismej.2010.149 (2011)。
Engstrand l . &林德伯格,m .幽门螺杆菌和胃微生物群。39-45最好Pract Res反应27日,10.1016 / j.bpg.2013.03.016 (2013)。
Lievin-Le Moal诉& Servin a l .肠主机的前线防御不受欢迎的入侵的有害微生物:黏蛋白,抗菌肽和微生物群。Microbiol转速19,315 - 337,10.1128 / cmr.19.2.315 - 337.2006 (2006)。
菲利普森,m . et al .胃黏液层:成分和监管的积累。是杂志Gastrointest肝脏杂志295,g806 - 812, 10.1152 / ajpgi.90252.2008 (2008)。
霍林m a & Swanson, b . j .黏蛋白在癌症:细胞表面的保护和控制。Nat牧师癌症4,45 - 60,10.1038 / nrc1251 (2004)。
洛夫格伦,j·l . et al。幽门螺杆菌感染缺乏共生的菌群INS-GAS小鼠减少胃炎和延迟上皮内瘤变。胃肠病学140年,210 - 220,10.1053 / j.gastro.2010.09.048 (2011)。
Lertpiriyapong, k . et al .胃殖民限制共生的微生物群复制肿瘤病变的推广不同的肠道微生物群胃幽门螺杆菌INS-GAS小鼠模型的致癌作用。63年肠道,54 - 63,10.1136 / gutjnl - 2013 - 305178 (2014)。
Riley d . r . et al . Bacteria-Human体细胞横向基因转移是癌症样本丰富。e1003107 PLoS第一版杂志9日,10.1371 / journal.pcbi。1003107 (2013)。
马西,y . et al。剖析了单细胞生物“暗物质”的罕见遗传分析和不文明的TM7微生物从人类的嘴。《美国国家科学院刊年代104年,11889 - 11894,10.1073 / pnas上。0704662104 (2007)。
Kuehbacher, t . et al .肠道TM7细菌的发展史在活跃的炎症性肠病。J地中海Microbiol 57岁,1569 - 1576,10.1099 / jmm.0.47719-0 (2008)。
Elinav、大肠等。NLRP6 inflammasome调节对结肠炎结肠微生物生态学和风险。细胞145、745 - 757、10.1016 / j.cell.2011.04.022 (2011)。
哇,p . c . et al。口腔链球菌sinensis作为自然宿主。Microbiol感染14,1075 - 1079 (2008)。
f . et al .链球菌sinensis Faibis:一个新兴的感染性心内膜炎的代理。J地中海Microbiol 57岁,528 - 531,10.1099 / jmm.0.47528-0 (2008)。
Nikolaitchouk: et al .乳酸菌coleohominis sp. 11月,独立于人类的来源。Int J系统另一个星球Microbiol 51, 2081 - 2085 (2001)。
西迪基,H。,Nederbragt, A. J., Lagesen, K., Jeansson, S. L. & Jakobsen, K. S. Assessing diversity of the female urine microbiota by high throughput sequencing of 16S rDNA amplicons. BMC Microbiol 11, 244, 10.1186/1471-2180-11-244 (2011).
浆果,d &钟情w .肠道菌群:小说在炎性肠道疾病生物标记物的来源吗?47-58最好Pract反应》27日,10.1016 / j.bpg.2013.03.005 (2013)。
Eckburg, p . b . et al。人类肠道微生物菌群的多样性。科学308年、1635 - 1638年的10.1126 /科学。1110591 (2005)。
Arumugam, m . et al .人类肠道微生物菌群。大自然473年,174 - 180,10.1038 / nature09944 (2011)。
托雷斯,j . et al。在墨西哥社区seroepidemiologic幽门螺杆菌感染的研究。J感染说178,1089 - 1094 (1998)。
迪克逊,m F。,Genta, R. M., Yardley, J. H. & Correa, P. Classification and grading of gastritis. The updated Sydney System. International Workshop on the Histopathology of Gastritis, Houston 1994. The American journal of surgical pathology 20, 1161–1181 (1996).
劳伦,p .两种胃癌组织学主要类型:分散和所谓的肠胃型癌。一个尝试Histo-Clinical分类。64年学报分册Microbiol Scand 31-49 (1965)。
明,美国胃癌。pathobiological分类。癌症39、2475 - 2485 (1977)。
Lozupone C。,Hamady, M. & Knight, R. UniFrac--an online tool for comparing microbial community diversity in a phylogenetic context. BMC Bioinformatics 7, 371, 10.1186/1471-2105-7-371 (2006).
确认
这项工作是由CONACYT (2007 - c01 - 69450)和Coordinacion de Investigacion en Salud IMSS (Fis / G12/1102),墨西哥。j.t是收件人Fundacion IMSS的排他性奖学金,墨西哥。
作者信息
作者和联系
贡献
j.t设计研究;j.t和F.A.J.分析数据和写了主要的手稿;F.V.J.处理临床样品微生物群分析;R.M.G.负责病人的选择和临床诊断;点负责组织样本的选择和病理学研究。所有作者回顾了手稿。
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
权利和权限
这项工作是在Creative Commons许可Attribution-NonCommercial-NoDerivs 3.0 Unported许可证。查看本许可证的副本,访问http://creativecommons.org/licenses/by-nc-nd/3.0/
关于这篇文章
引用这篇文章
Aviles-Jimenez F。,Vazquez-Jimenez, F., Medrano-Guzman, R.et al。胃微生物群组成non-atrophic胃炎患者之间的变化和肠道类型的胃癌患者。Sci代表44202 (2014)。https://doi.org/10.1038/srep04202
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/srep04202
本文引用的
发现特殊的微生物群与使用RNA-sequencing胃癌的发生和发展
科学报告(2023)
分析细菌多样性和群落结构在晚期胃癌患者胃液
发现肿瘤(2023)
non-Helicobacter螺杆菌细菌的作用在胃肠疾病的发病机制
肠道病原体(2022)
物种组成和总体多样性舌苔之间显著相关,胃炎患者胃液体微生物
BMC医学基因组学(2022)
主机在胃癌miRNAs-microbiota交互
转化医学杂志》(2022)