摘要
由于遗传背景、饮食、文化习惯和社会经济地位等多种因素的影响,世界人口的肠道菌群差异很大。本文采用鸟枪宏基因组测序方法对110例健康蒙古成年人肠道菌群进行了特征分析,并对蒙古人、汉族和欧洲人的肠道菌群进行了比较。结果表明,肠道菌群的分类图谱显示放线菌属和放线菌属双歧杆菌属分别是蒙古人、汉族和欧洲人在门水平和属水平上差异的关键微生物。宏基因组种分析表明Faecalibacterium prausnitzii而且Coprococcus来它们可能分别通过抗炎特性和丁酸盐的产生对肠道健康有贡献。另一方面,丰富的属Collinsella、症状性动脉粥样硬化患者的生物标志物,可能与蒙古成人心脑血管疾病的高发病率有关。在功能水平上,蒙古人肠道中存在独特的微生物代谢途径分布,主要分布在氨基酸代谢、碳水化合物代谢、能量代谢、脂类代谢、聚糖生物合成和代谢。我们可以将蒙古人肠道微生物群的特殊特征归因于他们独特的基因型、饮食习惯和生活环境。
简介
肠道菌群(GM)被认为是人类共同进化的伙伴,促进营养获取和免疫调节,并帮助维持宿主的稳态,以应对深刻的生活方式变化1,2,3..通过下一代测序,研究人员已经勾画出世界不同人群肠道宏基因组的结构和功能配置4,5,6,7.功能性转基因布局可能反映了寄主的基因型特征,反映了生态系统对寄主饮食和文化习惯的适应性反应。
蒙古人的微生物群对研究人员特别有兴趣,因为蒙古包含了独特的广泛的环境条件和民族地理群组。此外,蒙古帝国是世界上最大的毗连帝国,在中世纪蒙古人与欧亚大陆的各种人口融合的过程中,对蒙古与欧洲之间的文化交流产生了重大影响。重要的是,据报道,世界上超过20%的人与蒙古人有血缘关系8.生活在草原上的蒙古人,如肯提省,保持着传统的游牧生活方式(图1一个),而在城市地区,如乌兰巴托(蒙古的首都)和TUW省(首都的郊区),由于快速的现代化和经济发展,已经采取了城市的生活方式。呼和浩特和锡林郭勒地区的中国蒙古人与汉族通婚,这可能在基因组水平上与北方同胞产生一些差异。然而,所有蒙古人的饮食都是相似的,包括传统的奶酪(图1 b)和红肉(图1 c)作为它们的主食。
在我们之前的研究中,我们利用454焦磷酸测序技术描述了来自蒙古和中国的蒙古人肠道菌群的概况。在属的层面上,普氏菌的厚壁菌门门是最丰富的属拟杆菌,粪杆菌,示子杆菌,玫瑰杆菌,梭状芽胞杆菌,粪球菌,瘤胃球菌,阿里杆菌,副杆菌,链状杆菌,下绒毛杆菌和真杆菌均超过1%,我们发现季节性对蒙古农村人群肠道菌群的影响更为明显9,10.在以往的研究中,我们发现蒙古人的肠道菌群具有一些独特的特征。然而,对蒙古人肠道菌群的功能基因和代谢途径了解甚少11,12.据报告,蒙古成年人的心脑血管疾病患病率明显高于其他民族,这些疾病已成为蒙古人死亡的主要原因。先前的一些研究将上述慢性代谢性疾病归因于蒙古人大量食用红肉和酒的饮食传统1.随着人类肠道菌群工程和下一代测序技术的发展,肠道菌群与人类各种代谢性疾病之间的关系已成为研究热点13,14.通过探索心脑血管疾病和肠道菌群之间的潜在联系,这种方法也可能产生重大的见解。
本文采用鸟枪宏基因组测序方法对110例健康蒙古成年人肠道菌群进行了特征分析,并对蒙古人、汉族和欧洲人的肠道菌群进行了比较。(我)的肠道菌群的分类概况的队列显示放线菌和双歧杆菌属分别是蒙古人、汉族和欧洲人在门水平和属水平上差异的关键微生物。(2我们构建了包含1491813个基因的蒙古人非冗余微生物目录,并将本目录与其他已发表的汉族和欧洲人健康肠道微生物目录进行了合并。最终合并的非冗余目录包含5127,164个基因,其中283,401个基因是所有个体共有的。(3)在功能水平上,蒙古人肠道中存在独特的微生物代谢途径谱,主要分布在氨基酸代谢、碳水化合物代谢、能量代谢、脂类代谢、糖基生物合成和代谢。我们可以将蒙古人肠道微生物群的特殊特征归因于他们独特的基因型、饮食习惯和生活环境。
结果
分类描述
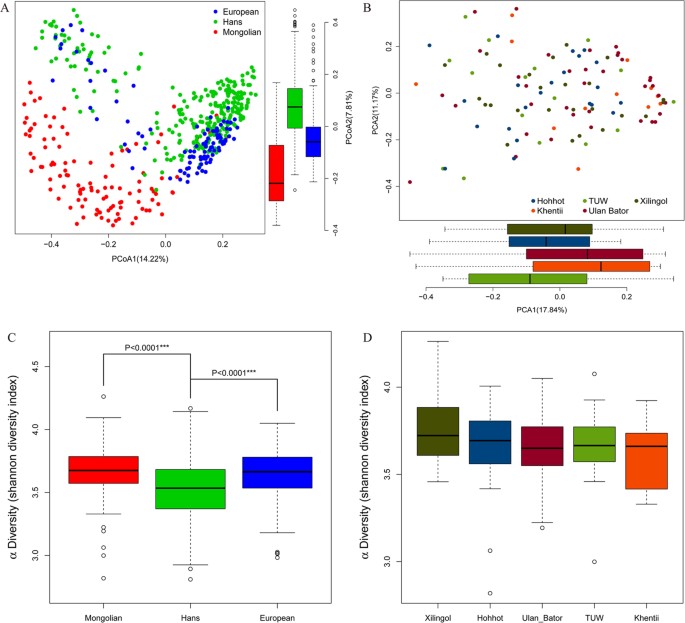
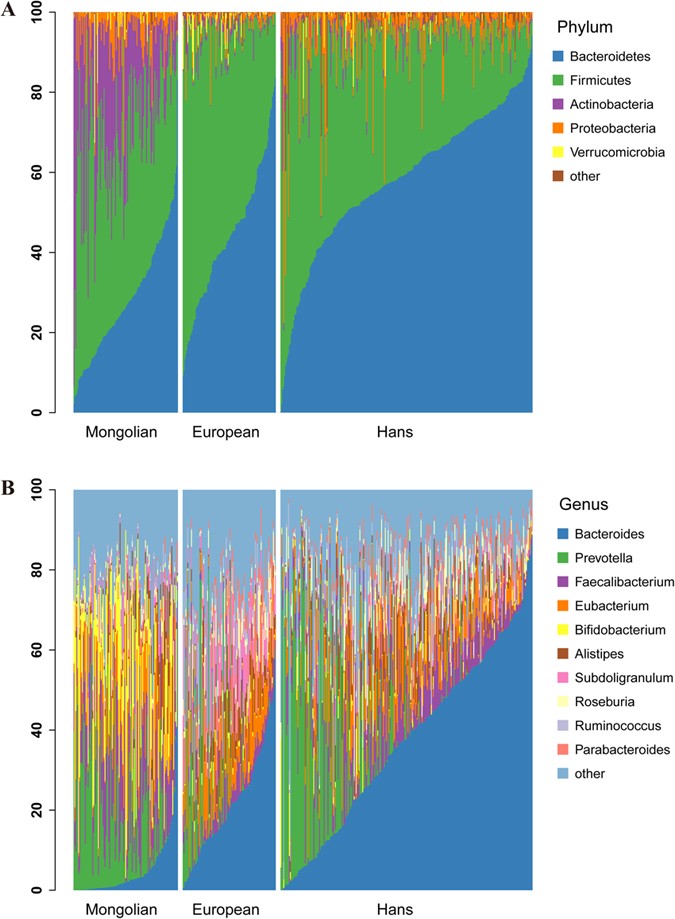
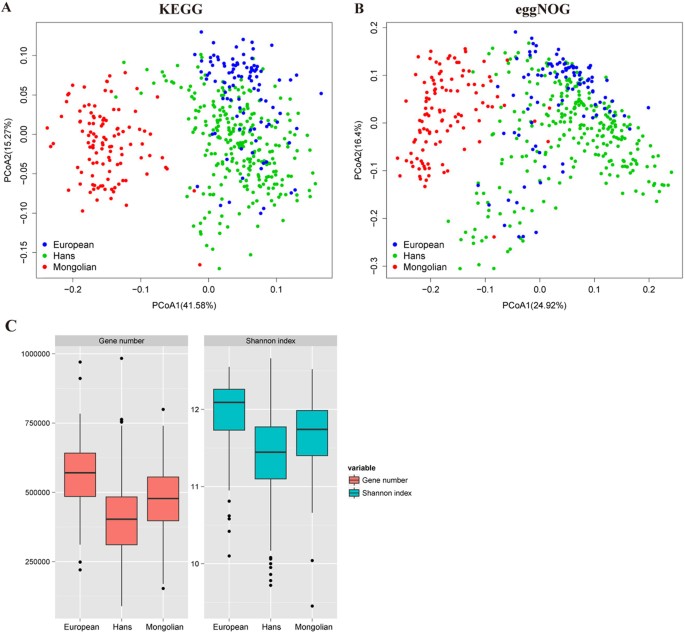
在本研究中,我们对110名蒙古人的粪便菌群进行了特征分析,并比较了呼和浩特、新陵果勒、TUW、乌兰巴托和Khentii人群的肠道菌群结构。抽样地点包括相当一部分人口相对密集的蒙古族聚居区,包括中国和蒙古地区。结果选取了蒙古乌兰巴托市(35例健康成人)、图乌省(16例健康成人)和肯提牧区(12例健康成人)、中国呼和浩特市(22例健康成人)和锡林郭勒牧区(25例健康成人)5个具有代表性的住区进行采样。基于物种丰度Bray-Curtis距离的主坐标分析(图2一个)揭示了汉族人和欧洲人的肠道菌群结构相似,但与蒙古人有显著差异。基于Shannon指数的细菌α多样性(图2 c)表明蒙古人和欧洲人的肠道菌群丰度显著高于汉族人。重要的是,我们观察到呼和浩特、新凌果勒、图瓦略、乌兰巴托和肯提地区的蒙古人在这些全局参数上没有显著差异(图2 d).
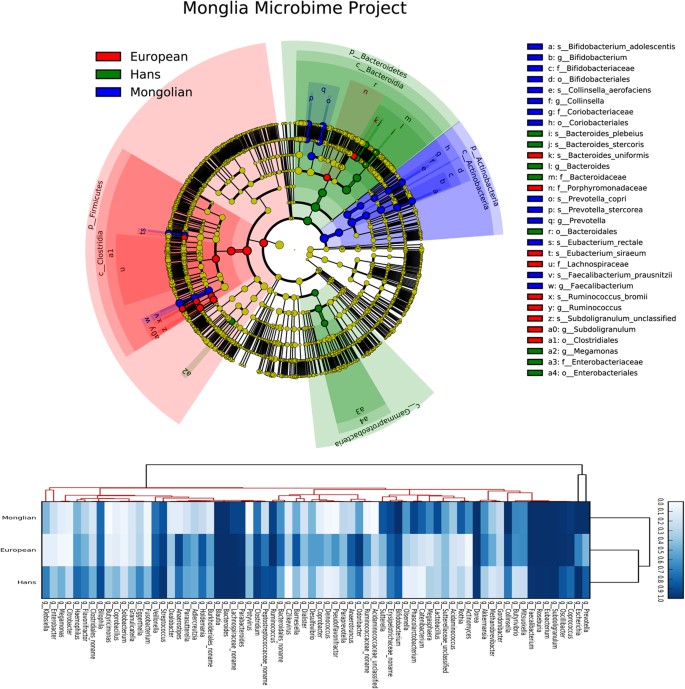
为了进一步了解蒙古人群肠道微生物群落特征,我们将结果与以往报道的样本进行了比较2,10,11,15利用Metaphlan2程序对宏基因组数据进行分类赋值。结果显示在系统发育树图3.对于所有这些蒙古小群体来说,放线菌,罗斯菌,连环菌,Phascolarctobacterium, Sutterella和乳杆菌属的含量显著增加。相反,Holdemania, peptostreptococcus, Potyvirus或Anaerostipes在所有蒙古样本中很少检测到(图3).人群肠道微生物群的分类概况(图4)也透露Actinobaceria而且双歧杆菌属分别是蒙古人、汉族和欧洲人在门水平和属水平上差异的关键组成部分。
蒙古国粪便微生物群项目与其他国家的比较。
利用Metaphlan2程序对欧洲、蒙古和汉族3个类群的所有样本数据进行了分类信息检测,分类图谱由LEfSe分析(http://huttenhower.org/galaxy/),热图为所有样品中微生物类群的检测频率(100%为深蓝色,0%为空白)。
与蒙古人有关的宏基因组种
为了探索蒙古人肠道菌群的特征,我们比较了110名蒙古人和268名汉人的基因丰度,从中我们发现了115,783个基因的显著丰度差异(Wilcoxon rank-sum, Benjamin-Hochberg)问-value < 0.001):汉族多78,345个,蒙古族多37,438个。蒙古人与欧洲人的相似性比较,共鉴定出127,540个基因,其中欧洲人富集66,030个,蒙古人富集61,510个。
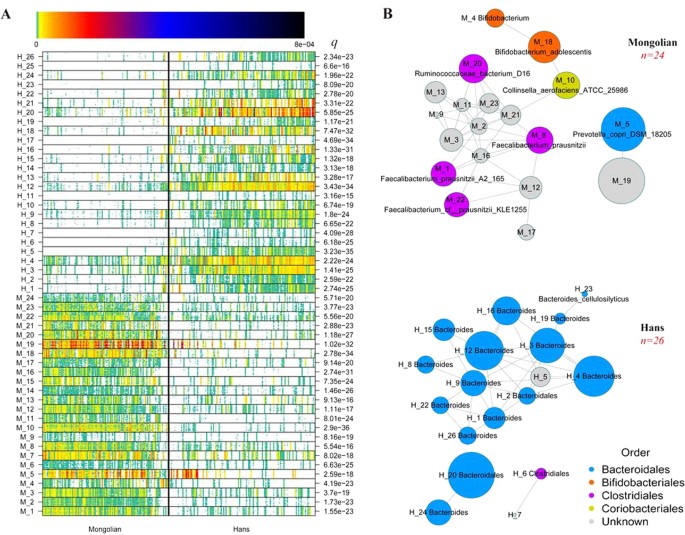
相关性高的差异丰度基因聚类为MGS16,17根据它们在所有样本中的含量。我们发现两组间差异有50个MGS,汉族个体和蒙古族个体分别有26和24个MGS, (图5而且补充表3).根据蒙古人显著富集的MGS的分类特征,我们发现了3种Faecalibacterium prausnitzii,其中两种在菌株水平和一种在物种水平上被发现,具有抗炎特性,并在先前的研究中发现与“健康”微生物组相关16,18.另一种富集于蒙古的MGS在种属水平上被鉴定为Coprococcus来ATCC 27758,它可能通过丁酸盐的产生促进肠道健康。有趣的是,Collinsella aerofaciens,在蒙古人体内富集,在有症状的动脉粥样硬化患者体内富集12.在蒙古人和欧洲人的比较中,我们发现欧洲人和蒙古人的MGS分别增加了29和16个(无花果S2。).值得注意的是,就像汉族和蒙古族之间的比较一样,普鲁氏粪杆菌,青少年双歧杆菌而且Collinsella aerofaciens在蒙古人身上得到了丰富。
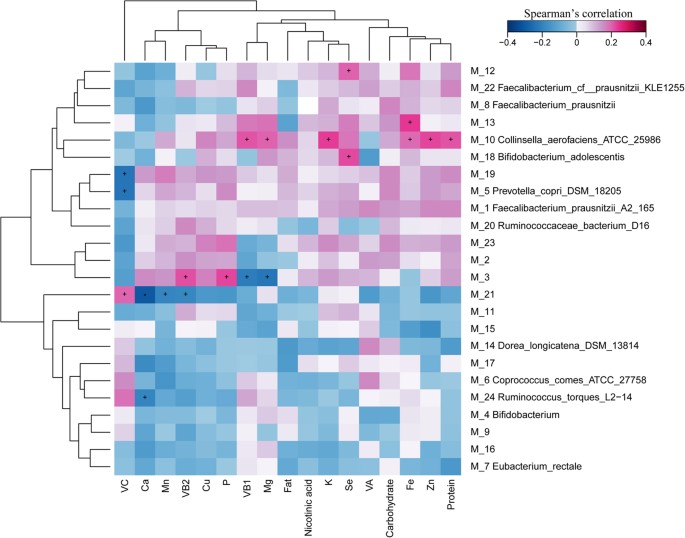
我们关注的另一个有趣的问题是蒙古人的饮食和他们的肠道菌群之间的相关性。通过将食物频率问卷转化为营养信息,计算了蒙古人富含MGS与膳食营养因子(图6而且S3无花果。).从图中可以看出,一个显著的正(P蛋白质、K、Zn、Fe、VB1与种属之间存在相关性(< 0.05)Collinsella aerofaciens.硒和镁元素呈显著阳性(P< 0.05)种间相关性双歧杆菌adolescentis.
肠道微生物基因目录
我们利用illumina平台对110个蒙古人的粪便宏基因组进行了测序,获得725 Gb的原始数据,平均每个样本6.54 Gb。经过质量控制,我们得到了平均每个6.33 Gb的数据,总共696 Gb的高质量数据,没有适配器和人类DNA污染。在此基础上,利用MetaHIT开发的方法,构建了蒙古5个地区(Khentii牧区、TUW省、乌兰巴托、呼和浩特市和锡林郭勒牧区)的蒙古基因目录。来自乌兰巴托的样本基因目录最丰富,包含768497个基因。Khentii集共包含400178个基因,样本最少。锡林郭勒、呼和浩特和TUW目录分别包含512,067,702,521和433,515个基因。合并后的非冗余目录包含1491813个基因。呼和浩特、肯提、TUW、乌兰巴托和锡林郭勒目录集分别包含137,496、99,760、110,428、277,758和242,637个独特基因。
此外,我们还将蒙古目录与深圳(T2D)、杭州(LC)、美国(HMP)、欧洲(MetaHIT)四种已出版的健康肠道微生物目录进行了比较。2,10,11,15.为了便于比较,我们使用相同的标准预测上述四个健康组的基因。蒙古基因目录包含1418578个基因,美国基因目录22243320个基因,欧洲基因目录1711862个基因,杭州基因目录1183368个基因,深圳基因目录2067885个基因。所有目录共有283,401个基因。蒙古、美国、欧洲、杭州和深圳样本分别包含599,370、1015、117、591,857、233,036和777,465个独特基因。最终合并的非冗余目录包含5,127,164个基因(无花果S1。).
功能描述
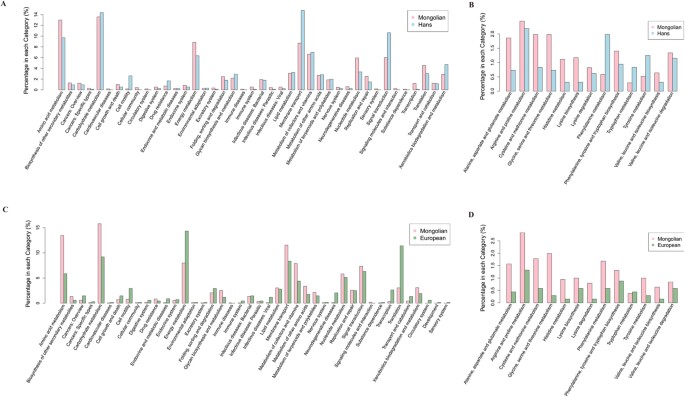
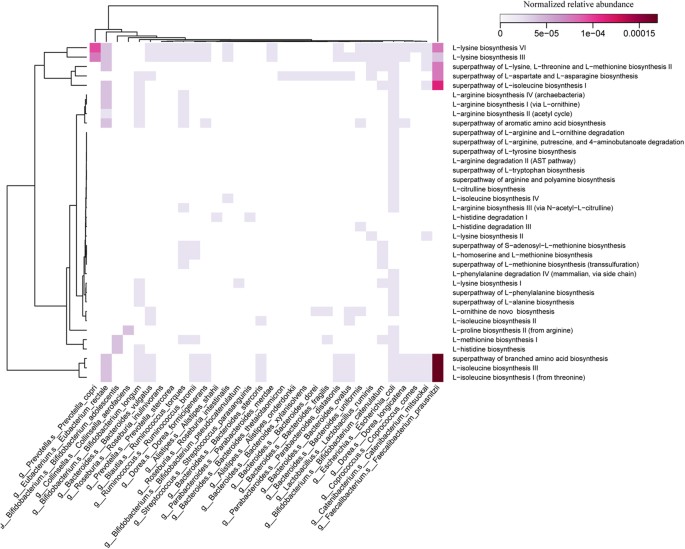
为了探索蒙古人肠道菌群的功能特征,我们用KEGG和eggNOG对基因目录进行了注释。在功能水平上,可以观察到蒙古人、汉族和欧洲人的肠道菌群功能结构存在显著差异(图7 b),因为蒙古人的肠道中存在一个独特的KEGG同源组(KO)谱。与分类多样性相似的是,蒙古人和欧洲人的肠道在功能水平上的多样性高于汉族(图7 c).在途径水平上,蒙古人富集的KOs主要分布在氨基酸代谢、碳水化合物代谢、能量代谢、脂质代谢、聚糖生物合成和代谢(图8 a, C).在氨基酸代谢中,富集途径主要有丙氨酸、天冬氨酸和谷氨酸代谢、半胱氨酸和蛋氨酸代谢、赖氨酸生物合成和赖氨酸降解(图8 b, D).结合各物种氨基酸代谢途径的丰度分析,普氏粪杆菌,柯氏普氏杆菌这似乎是氨基酸代谢的主要贡献者,特别是L-赖氨酸生物合成和L-异亮氨酸生物合成(图9).提示某些功能富集可能与某些特定物种的富集有关。丰富的氨基酸代谢和能量代谢功能与蒙古人较高的肉和发酵吸收有关(无花果S6。).此外,通过计算报告评分,我们发现蒙古人与汉族或欧洲人之间存在显著差异(报告评分> 1.6)的丰富代谢模式。在蒙古受试者中差异丰富的大多数模式与观察到的代谢途径的变化相吻合,主要是碳水化合物、氨基和核苷酸糖、磷酸转移酶和氨基酰基trna的代谢以及甲烷和硫化物的生物合成。无花果。S4).
种水平微生物氨基酸代谢途径热图。
热图显示了每个物种(蒙古人的中位数)每种路径的丰度。各物种各路径归一化相对丰度采用HUMAnN2 (http://huttenhower.sph.harvard.edu/humann2).
由于我们观察到蒙古人和汉族之间的碳水化合物代谢有显著差异。我们进一步探索了蒙古和汉族肠道宏基因组中复杂碳水化合物降解的潜力,并筛选了组装的contigs中的碳水化合物活性酶(CAZymes)。结果表明,蒙古品种的ca酶多样性高于汉族品种。P< 0.0001, Wilcoxon秩和检验;无花果S5。).这表明蒙古转基因具有较强的复杂碳水化合物代谢能力。
讨论
在本研究中,我们观察到来自蒙古和中国两个国家的蒙古族肠道菌群的几个明显特征。在分类学上,属Collinsella蒙古人的蛋白质含量显著增加,与蒙古人的蛋白质摄入量显著正相关(图6).据报道Collinsella是否被认为是症状性动脉粥样硬化患者的生物标志物19.在之前的研究中,我们比较了有症状的动脉粥样硬化患者和健康对照组的肠道菌群。在属水平上,表明其丰富程度Collinsella在有症状的动脉粥样硬化患者中增加,而Roseburia而且真细菌在健康对照组中增加了吗19.此外,Collinsella,肠杆菌科而且链球菌被认为是潜在的促炎GM成分在T2D患者中富集,导致肠道SCFA迅速下降20..据此,我们可以推断蒙古人饮食中蛋白质的高吸收与肠道的强健生长有关Collinsella,这可能会增加蒙古成年人心脑血管疾病的发病风险。
另一方面,乳酸菌而且双歧杆菌属通常被认为是益生菌的细菌在蒙古人的肠道中大量发现21,22.正如我们所知,乳酸菌而且双歧杆菌属是人类肠道中的主要有益微生物。它们表现出一系列促进健康的作用,包括调节肠道微生物内稳态19,抑制肠粘膜内的病原体23即局部和全身免疫反应的调节,维生素的产生24,以及许多膳食化合物转化为生物活性分子。不同种类的乳酸菌在酸奶发酵中起了关键作用。在之前的研究中,研究人员已经证明乳酸菌能够促进殖民双歧杆菌属23.因此,乳酸菌属和乳酸菌属广泛存在双歧杆菌属可能与蒙古人食用发酵奶的饮食习惯有关。此外,我们发现属双歧杆菌属与饲粮硒呈显著正相关。Se与属的相互作用双歧杆菌属研究发现MRS ager中补充Se能促进小鼠的生长双歧杆菌属25.
蒙古人肠道菌群中碳水化合物、氨基酸和核苷酸糖、磷酸转移酶和氨基酰基trna途径丰度的增加表明蒙古人肠道中微生物的生长和代谢阶段较汉人更为旺盛。氨基酰基trna能够与相应的动物酸结合,并将动物酸转移到核糖体上26.在本研究中,我们发现蒙古人肠道中氨基酰trna的生物合成增强,表明微生物基因表达水平整体增强。此外,磷酸化转移酶代谢途径的上调为微生物的增殖提供了能量来源27.一方面,蒙古人肠道微生物的旺盛代谢能够促进肠道对食物的消化和吸收;另一方面,由于肠道微生物的新陈代谢更强,会产生更多的代谢产物,包括对人体健康有更大影响的有益或有害因素。
据此,我们发现蒙古人肠道菌群中甲烷和硫化物的生物合成显著高于其他人群。这两种化合物被认为是人类肠道中典型的炎症因子。以往的研究已经证实了T2D肠道中甲烷和硫化物的危害性28IBD患者29,30..因此,很容易推测蒙古成年人心脑血管疾病的高发病率与他们肠道菌群中甲烷和硫化物的较强生物合成有关。
在蒙古族饮食中,肉类(包括羊肉和牛肉)是蛋白质的主要来源,蒙古人消耗的动物蛋白量明显高于汉族人。肠道菌群在动物蛋白质代谢中起着关键作用。在这个菌群中,属梭状芽胞杆菌而且拟杆菌,在蒙古人肠道中广泛检测到,主要与动物蛋白质、多种氨基酸和饱和脂肪酸的代谢有关16,17,18.因此,我们注意到蒙古人肠道菌群中蛋白质的降解途径和氨基酸的生物合成途径较为丰富。
材料与方法
样品信息
本研究共招募了110名蒙古人进行抽样。其中蒙古人63人,中国内蒙古人47人。来自蒙古的参与者中,35名志愿者生活在蒙古首都乌兰巴托的典型现代生活方式,16名志愿者生活在覆盖乌兰巴托郊区的TUW省,12名志愿者生活在典型的蒙古草原Khentii牧区。来自中国内蒙古的47名志愿者中,22名志愿者生活在内蒙古首府呼和浩特,过着城市生活方式。其余的牧民生活在锡林郭勒牧区,是典型的蒙古草原。在获得书面知情同意后,我们使用食物频率问卷从所有参与者收集习惯性长期饮食信息,并将其转化为营养信息(补充表1).该研究方案得到了内蒙古农业大学(呼和浩特,中国)伦理委员会的批准。在材料和方法中描述的采样和所有后续步骤已按照批准的指南进行。
DNA测序
如前所述,使用QIAampDNA粪便MiniKit从粪便样本中提取DNA10.根据Illumina TruSeq DNA Sample Prep v2指南,用2 μg基因组DNA构建宏基因组DNA文库,平均插入大小为500 bp。使用安捷伦生物分析仪和DNA LabChip 1000试剂盒评估所有文库的质量。测序软件为Illumina Hiseq2500。
用于比较的宏基因组数据
本研究使用的公共肠道微生物宏基因组数据包括:(i) 83份健康的汉斯粪便样本(杭州),从EBI下载,登录号为ERP00586014;(ii) 185个健康的汉斯粪便样本(深圳),从NCBI下载,登录号为SRA04564614;来自MetaHIT项目的99个欧洲健康粪便样本,从EBI下载,登录号为ERA0001162.
读取的质量控制
采用以下标准对原始的成对端读取进行质量控制:(1)去除带适配器的读取;(2)从3 '端使用质量阈值30对读进行修剪;(3)去除含有50%以上碱基的低质量reads (Q30);(4)小于70bp的Reads被删除;(5)删除了映射到人类基因组的reads。对于对齐,使用SOAPaligner 2.21 27,参数为' -m 100 -x 1000 ',这是“报告所有重复点击,最小插入大小为100 bp,最大插入大小为1000”。有关上述每个选项含义的详细信息,请参阅http://soap.genomics.org.cn/soapaligner.html.得到的高质量读数用于进一步的分析。
德新生装配与基因目录构建
使用SOAPdenovo 28(2.04版本)使用参数' - 3m -u -L 100 -d 1 -F -k 53 '组装高质量读取,这是“mergeLevel为3,在脚手架前取消高/低覆盖率的遮蔽contigs, minContigLen为100,KmerFreqCutoff为1,填补脚手架的空白,k-mer长度为53”。有关上述每个选项含义的详细信息,请参阅http://soap.genomics.org.cn/soapdenovo.html.得到的支架在模糊Ns处被切割成contigs,大于500 bp的contigs被保存。所有这些contigs都被用于基因预测。
我们使用MetaGeneMark31(3.26版本)从每个样本的contigs中识别orf,使用长度阈值为100 bp (补充表2).然后将所有预测的orf与CD-HIT进行两两比较,构建非冗余基因目录32(4.5.7版本),使用0.95的序列识别截点去除冗余基因,对齐长度覆盖较短序列的90%以上。最终蒙古非冗余基因目录包含1491813个微生物基因,平均长度为808 bp。
鉴于有限的测序数据中没有检测到一些低丰度的微生物,我们结合之前构建的基因目录,包括MetaHIT基因目录2, HMP基因目录3., T2D基因目录14和LC基因目录13建立非冗余基因目录供进一步分析。利用MetaGeneMark对上述研究的健康个体的基因组orf进行预测,并与蒙古基因目录合并为最终的orf,其标准为短基因覆盖率90%,一致性95%。最终,我们得到了一个非冗余的人类肠道基因目录,包含4,998,380个基因,平均长度为763 bp。
分类学和基因图谱
我们使用MetaPhlAn233使用默认参数生成生物体丰度分析,这依赖于从约17000个参考基因组中识别的约100万个独特的进化支特异性标记基因。共有1036种微生物被定位到标记基因上,其中古生菌9种,细菌963种,真核菌18种,病毒46种。
用秦N的方法测定了基因的相对丰度等.大自然2014年13.在计算基因丰度时,使用SOAPalign 2.21将每个样本的高质量reads与基因目录进行比对15参数为' -r 2 -m 100 -x 1000 '时,只接受能映射到同一基因的两端reads。对于对齐,使用SOAPaligner 2.21,参数为' -m 100 -x 1000 ',这表示“报告所有重复命中,最小插入大小为100 bp,最大插入大小为1000”。有关上述每个选项含义的详细信息,请参阅http://soap.genomics.org.cn/soapaligner.html.
机动式分析
为了比较蒙古人和汉人的粪便微生物群,鉴定了具有差异丰度的基因标记(Benjamin-Hochberg)问-value < 0.001)。蒙古人和欧洲人的比较也是用同样的方法进行的。为了将基因聚类为宏基因组物种(MGS),我们采用了Le Chatelier所描述的方法33和尼尔森15.我们将Spearman相关系数(rho) >0.8的基因采用单连锁聚类方法进行聚类,然后将超过25个Spearman相关系数(rho) >0.8的基因进行聚类融合。
MGS的分类注释如前所述13.当>90%的基因对同一系统发育组有最佳命中时,用blast与>95%的一致性和>90%的重叠查询,将MGS划分为从品系到超级王国级别的分类级别。
为了构建MGS共现网络,我们利用MGS的丰度计算其间的Spearman相关系数,并根据Spearman相关系数对MGS进行聚类。用Cytoscape3.0.2软件对MGS共现网络进行可视化分析。
KEGG, EggNOG和CAZy分析
预测基因的氨基酸序列与eggNOG 3.0数据库中的蛋白质进行比对34和KEGG数据库35(发布2014-12-09)使用BLAST。每个基因被分配到一个或多个OG(s)或EC(s),通过包含至少一个得分超过60位的HSP的最高注释得分命中(s)。为了构建eggNOG中OGs和KEGG数据库中ECs的图谱,我们采用了《秦J》中介绍的方法,对来自同一OG或EC的基因进行相对丰度的积累。等.大自然2010年2.为了确定差异富集的KO模块,我们从单个KO的z分数中计算它们的报告分数36.报告评分为Z > 1.6的模块定义为差异富集模块。
从氨基酸序列预测CAZymes(碳水酶活性酶),对照dbCAN数据库中CAZymes的家族特异性HMM37使用HMMER 3.0包中的Hmmscan程序38.
所有样本的原始测序数据已存入Sequence Read Archive (http://www.ncbi.nlm.nih.gov/sra/)根据SRP080787条款。
额外的信息
如何引用这篇文章:刘,W。等.宏基因组分析揭示了蒙古族肠道微生物群的独特特征。科学。代表。6, 34826;Doi: 10.1038/srep34826(2016)。
改变历史
2017年1月04日
本文的HTML和PDF版本都附有更正。这个错误在文中已经改正了。
参考文献
莱,R. E.,特恩堡,P. J.,克莱因,S.和戈登,J.微生物生态学:与肥胖相关的人类肠道微生物。Nature 444, 1022-1023, doi: 10.1038/4441022a(2006)。
秦杰等。通过宏基因组测序建立人类肠道微生物基因目录。Nature 464, 59-65, doi: 10.1038/nature08821(2010)。
大卫,洛杉矶等。饮食可以快速和重复地改变人体肠道微生物群。自然学报(自然科学版),2015,(1)。
Yatsunenko, T.等。跨越年龄和地域的人类肠道微生物组。Nature 486, 222-227, doi: 10.1038/nature11053(2012)。
马丁内斯,我,等人。巴布亚新几内亚农村的肠道菌群:组成、多样性模式和生态过程。Cell Rep 11, 527-538, doi: 10.1016/j.celrep.2015.03.049(2015)。
Rampelli, S.等。哈扎狩猎采集者肠道菌群的宏基因组测序。中华生物学报25,1682-1693,doi: 10.1016/j.cu .2015.04.055(2015)。
Clemente, J. C.等。未接触的美洲印第安人的微生物群。科学通报1,doi: 10.1126/sciadv.1500183(2015)。
佩德森,N.,赫塞尔,A. E.,巴塔尔比勒格,N.,安丘凯提斯,K. J.和迪·科斯莫,N.大雨,干旱,蒙古帝国,和现代蒙古。中国科学院学报(自然科学版)111,4375-4379,doi: 10.1073/pnas.1318677111(2014)。
张杰等。内蒙古蒙古族肠道菌群多样性研究。Benef Microbes 4,319 - 328, doi: 10.3920/BM2013.0028(2013)。
张杰等。蒙古人核心肠道菌群及其与季节性饮食变化的相关性科学通报,2015,(3):344 - 344。
蒙古人颈动脉粥样硬化的决定因素。种族与疾病20,257-260(2010年)。
徐杰等。在中国蒙古男性中基于人群的前瞻性队列研究中腰高比对缺血性中风的预测价值。《公共科学图书馆》9,e110245, doi: 10.1371/journal.pone.0110245(2014)。
秦,N.等。肝硬化患者肠道菌群的改变。《自然》杂志,2015,(1)。
秦杰等。2型糖尿病患者肠道菌群的宏基因组相关性研究。《自然》490,55-60,doi: 10.1038/nature11450(2012)。
李锐,李宇春,李宇春,李天森,王杰。SOAP:短寡核苷酸对准程序。生物信息学24,713-714,doi: 10.1093/生物信息学/btn025(2008)。
Voreades, N., Kozil, A. & Weir, T. L.饮食与人类肠道微生物群的发展。微生物学前缘5,494,doi: 10.3389/fmicb.2014.00494(2014)。
吴国栋等。将长期饮食模式与肠道微生物肠道型联系起来。科学334,105-108,doi: 10.1126/ Science .1208344(2011)。
骑士,d等人。反思“欠佳”。细胞宿主微生物16,433-437,doi: 10.1016/j.c om.2014.09.013(2014)。
Alexander, m.d.等。颅内动脉粥样硬化性疾病支架植入术的临床症状差异和前处理无症状间隔影响疗效。AJNR。中华神经放射学杂志35,1157-1162,doi: 10.3174/ajnr。A3836(2014)。
Candela, M.等人。长寿ma - pi2饮食对2型糖尿病患者肠道菌群失调的调节作用英国营养学杂志116,80-93,doi: 10.1017/S0007114516001045(2016)。
Oda, Y.等。采食网眼柳树提取物改善人体免疫功能与肠道菌群变化:一项随机安慰剂对照试验科学通报10,e0142909, doi: 10.1371/journal.pone.0142909(2015)。
陈晓明,陈晓明,陈晓明,等。消化系统疾病中益生菌的研究进展[j] .中国生物医学工程学报(自然科学版)。
Inturri, R., Stivala, A. & Blandino, G.联合使用益生菌B. longum BB536和L.鼠李糖乳杆菌HN001的微生物学特性。中华医学会胃肠病学杂志61,191-197(2015)。
Hevia, Delgado, S, Sanchez, B.和Margolles, A.参与有益细菌和免疫系统相互作用的分子参与者。微生物学前缘6,1285,doi: 10.3389/fmicb.2015.01285(2015)。
Pusztahelyi, T., Kovacs, S., Pocsi, I. & Prokisch, J.亚硒酸盐胁迫选择用于硒源生产的益生菌突变株。中华微量元素医学生物学杂志30,96-101,doi: 10.1016/j.jtemb.2014.11.003(2015)。
洪洪杰等。氨基酰基trna合成酶相互作用多功能蛋白1通过负向调节髓源性抑制细胞功能抑制乳腺癌小鼠肿瘤生长。《癌症免疫学》杂志,2016,doi: 10.1007/s00262-015-1777-2。
转座子介导的大肠埃希菌磷酸烯醇丙酮酸定向突变的控制:糖磷酸转移酶系统。中国生物医学工程学报,2015,29(3):359 - 361。
Karlsson, F. H.等。欧洲葡萄糖控制正常、受损和糖尿病妇女的肠道宏基因组。Nature 498, 99-103, doi: 10.1038/nature12198(2013)。
冯,Q.等。肠道微生物群沿结直肠腺瘤-癌序列发展。Nat comm 6, 6528, doi: 10.1038/ncomms7528(2015)。
巴菲,c.g.等。精确的微生物组重建恢复了胆汁酸介导的艰难梭菌耐药性。Nature 517, 205-208, doi: 10.1038/nature13828(2015)。
野口,H. Park, J. & Takagi, T. MetaGene:从环境基因组鸟枪序列中发现原核基因。核酸Res 34, 5623-5630, doi: 10.1093/nar/gkl723(2006)。
Cd-hit:用于聚类和比较大量蛋白质或核苷酸序列的快速程序。生物信息学22,1658-1659,doi: 10.1093/生物信息学/btl158(2006)。
Segata, N.等。使用独特的进化枝特异性标记基因的宏基因组微生物群落分析。Nat方法9,811 - 814,doi: 10.1038/ nmem .2066(2012)。
Powell, S. et al. eggNOG v3.0:在41个不同的分类学范围涵盖1133个生物的同源类群。核酸Res 40, D284-D289, doi: 10.1093/nar/gkr1060(2012)。
Kanehisa, M., Goto, S., Kawashima, S., Okuno, Y. & Hattori, M.解码基因组的KEGG资源。核酸Res 32, D277-D280, doi: 10.1093/nar/gkh063(2004)。
利用代谢网络拓扑结构揭示代谢的转录调控。美国科学院学报102,2685-2689,doi: 10.1073/pnas.0406811102(2005)。
dbCAN:一个自动标注碳水化合物活性酶的网络资源。核酸Res 40, W445-W451, doi: 10.1093/nar/gks479(2012)。
基于概率推理的新一代同源搜索工具。Genome Inform 23, 205-211(2009)。
确认
我们衷心感谢所有蒙古志愿者的参与。国家自然科学基金(NO.)资助。国家自然科学基金资助项目(31430066);浙江省自然科学基金资助项目(81301475);LR15H030002)。
作者信息
作者和隶属关系
贡献
H.Z领导了这个项目,H.Z, N.Q.和W.L.构思和设计了这个项目,J.Z, X.X, W.H.和q.h收集了样品。b.z进行了DNA提取实验。B.Z.和C.J.进行文库构建和测序。nq设计了这项分析。C.W, C.J, j.z和z.l分析了数据。n.q和S.C.进行了功能注释分析。J.Z.和C.W.撰写了最初的手稿,s.c.、N.Q.和C.J.做出了重要贡献,其他所有作者都提出了批判性意见。
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
权利与权限
本作品遵循创作共用署名4.0国际许可协议。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在信用额度中另有说明;如果该材料不在创作共用许可之下,用户将需要获得许可持有人的许可才能复制该材料。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/
关于本文
引用本文
刘伟,张建军,吴聪,张建军。et al。宏基因组分析揭示了蒙古族肠道微生物群的独特特征。Sci代表6, 34826(2016)。https://doi.org/10.1038/srep34826
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/srep34826
进一步的阅读
慢性腹泻恒河猴(Macaca mulatta)肠道菌群和抗生素耐药菌群及其与人类肠道菌群的相似性
微生物组(2022)
利用深度学习从人类肠道菌群中鉴定抗菌肽
自然生物技术(2022)
一种蛋白水解激活的抗菌毒素编码在拟杆菌的移动质粒诱导保护性反应
自然通讯(2022)
人类肠道中的普雷沃氏菌属主要由copri普雷沃氏菌属、stercorea普雷沃氏菌属及相关谱系组成
科学报告(2022)
粪便微生物群移植后的细菌菌株的精确定量描述了长期移植并解释了结果
微生物学性质(2021)