- 软件gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
GSVA:微阵列和RNA-Seq数据的基因集变异分析gydF4y2Ba
BMC生物信息学gydF4y2Ba体积gydF4y2Ba14gydF4y2Ba,文章编号:gydF4y2Ba7gydF4y2Ba(gydF4y2Ba2013gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
基因集富集(GSE)分析是一种流行的框架,用于将基因表达谱中的信息浓缩成路径或签名摘要。与单基因分析相比,这种方法的优势包括噪声和降维,以及更强的生物学解释性。随着分子谱分析实验超越了简单的病例对照研究,我们需要稳健而灵活的GSE方法来在高度异构的数据集中建模途径活性。gydF4y2Ba
结果gydF4y2Ba
为了应对这一挑战,我们引入了基因集变异分析(gsa),这是一种GSE方法,以无监督的方式估计样本种群中通路活性的变化。我们通过与目前最先进的样品富集方法的比较,证明了GSVA的鲁棒性。进一步,我们提供了它在差异通路活性和生存分析中的效用的例子。最后,我们展示了gsa如何与微阵列和RNA-seq实验数据进行类比。gydF4y2Ba
结论gydF4y2Ba
与相应的方法相比,gsa在检测样本群体中微妙的通路活性变化方面提供了更强的能力。GSE方法通常被视为生物信息学分析的终点,而GSVA则构成了构建生物学通路中心模型的起点。此外,gsa有助于目前对RNA-seq数据的GSE方法的需求。gsa是R的一个开源软件包,它是Bioconductor项目的一部分,可以在以下网站下载gydF4y2Bahttp://www.bioconductor.orggydF4y2Ba.gydF4y2Ba
背景gydF4y2Ba
在基因组尺度上测量mRNA丰度的能力导致了许多对生物过程中潜在的不同分子模式进行编目的努力。为了方便解释和组织从微阵列实验中得到的长序列基因,引入了基因集富集(GSE)方法。他们系统地测量和注释分子图谱,这些图谱本身就有噪声且难以解释。GSE分析首先获得一个排序的基因列表,通常来自研究两组间基因表达变化的微阵列实验。然后将这些基因映射到预定义的基因集,并将它们的基因表达统计结果汇总为每个基因集的单个富集评分。这些基于通路的方法的一个显著好处是可解释性:基因功能是集体发挥的,可能会因环境刺激、基因修饰或疾病状态而变化。因此,将基因组织成基因集为评估生物活性提供了一个更直观和稳定的背景。gydF4y2Ba
GSE方法的许多方法变体已经被提出[gydF4y2Ba1gydF4y2Ba-gydF4y2Ba6gydF4y2Ba],包括非参数富集统计量[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba7gydF4y2Ba],电池测试[gydF4y2Ba8gydF4y2Ba-gydF4y2Ba10gydF4y2Ba,以及集中的基因集检测[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba].电池测试方法旨在从大量注释路径和基因签名中识别出突出的基因集。重点基因集检测方法试图仔细评估与所分析实验相关的少数基因集[gydF4y2Ba12gydF4y2Ba].GSE方法已成功应用于许多实验条件,用于解释包括癌症在内的生物状态的通路架构[gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,代谢性疾病[gydF4y2Ba15gydF4y2Ba]和发展[gydF4y2Ba16gydF4y2Ba].有关GSE方法的最新综述,读者可参考[gydF4y2Ba17gydF4y2Ba].gydF4y2Ba
许多GSE方法之间的一个重要区别是所检验的零假设的定义[gydF4y2Ba18gydF4y2Ba].竞争检验的零假设声明基因集内外的基因之间没有差异(例如,[gydF4y2Ba4gydF4y2Ba])。一个自给自足的检验只根据被测基因集中的基因来定义其零假设(例如,[gydF4y2Ba1gydF4y2Ba])。更具体地说,对于一个基因集的自成体系的检测,仅其中一个基因的差异表达就可以拒绝该基因集无差异表达的原假设。由此可见,在检测基因表达的细微变化方面,自成体系的检测比竞争性检测提供了更高的能力。但是,由于报告的结果数量可能很大,因此在电池测试环境中选出几个基因集可能并不有用。gydF4y2Ba
最后,许多GSE方法假设两个类(例如case/control),并在此上下文中评估富集[gydF4y2Ba19gydF4y2Ba-gydF4y2Ba22gydF4y2Ba].随着大型基因组研究的兴起,如癌症基因组图谱计划(TCGA -),这种假设所施加的限制变得明显起来gydF4y2Bahttp://cancergenome.nih.govgydF4y2Ba),这是一个雄心勃勃的项目,目标是识别多种癌症类型的分子决定因素。与小样本量的病例对照研究相比,TCGA项目有大量具有多种表型的患者队列,由分层、多类和截尾数据构成。因此,需要GSE方法来评估具有复杂表型性状的大型异质群体的途径变异。gydF4y2Ba
为了解决这些挑战,我们提出了一种非参数的、无监督的方法,称为基因集变异分析(gsa)。gsa计算样本的基因集富集分数作为基因集内外基因的函数,类似于竞争基因集测试。此外,它估计的变异基因集富集的样本独立于任何类别标签。从概念上讲,这种方法可以理解为基因表达数据坐标系统的变化,从gydF4y2Ba基因gydF4y2Ba来gydF4y2Ba基因集gydF4y2Ba.这种转换有助于事后构建以通路为中心的模型,如差异通路活性识别或存活预测。此外,我们通过将gsa应用于RNA-seq数据来证明它的灵活性。gydF4y2Ba
实现gydF4y2Ba
gsa方法的概览如图所示gydF4y2Ba1gydF4y2Ba,其中显示了两个主要的必需输入:一个矩阵gydF4y2BaXgydF4y2Ba= {gydF4y2BaxgydF4y2Ba我gydF4y2BajgydF4y2Ba}gydF4y2BapgydF4y2Ba×gydF4y2BangydF4y2Ba的规范化表达式值(有关预处理步骤的详细信息,请参见方法)gydF4y2BapgydF4y2Ba基因的gydF4y2BangydF4y2Ba样本,其中通常gydF4y2BapgydF4y2Ba≫gydF4y2BangydF4y2Ba以及一系列的基因集gydF4y2Ba.我们将用gydF4y2BaxgydF4y2Ba我gydF4y2Ba的表达式配置文件gydF4y2Ba我gydF4y2Ba-th基因,由gydF4y2BaxgydF4y2Ba我gydF4y2BajgydF4y2Ba的特定表达式值gydF4y2Ba我gydF4y2Ba-基因中的gydF4y2BajgydF4y2Ba-th样本,和bygydF4y2BaγgydF4y2BakgydF4y2Ba中的行索引的子集gydF4y2BaXgydF4y2Ba这样gydF4y2BaγgydF4y2BakgydF4y2Ba⊂gydF4y2Ba{1,……gydF4y2BapgydF4y2Ba}定义了一组形成通路或其他功能单元的基因。让|gydF4y2BaγgydF4y2BakgydF4y2Ba|是基因的数量gydF4y2BaγgydF4y2BakgydF4y2Ba.gydF4y2Ba
GSVA方法大纲。gydF4y2Bagsa算法的输入是log2微阵列表达值或RNA-seq计数形式的基因表达矩阵和基因集数据库。1.累积密度函数(kcdf)的核估计。这两个图显示了两个模拟的表达谱,模拟了来自微阵列和RNA-seq数据的6个样本。的gydF4y2BaxgydF4y2Ba-轴对应表达值,其中每个基因在4个低值样本中低表达,而在其他两个样本中高表达。kcdf的比例尺在左边gydF4y2BaygydF4y2Ba-轴和高斯核和泊松核的比例在右边gydF4y2BaygydF4y2Ba设在。2.对每个样本,表达级统计是按顺序排列的。3.对于每个基因集,计算Kolmogorov-Smirnov-like排序统计量。这幅图展示了一个由3个基因组成的基因集,总共有10个基因,并对该基因集内外的基因进行了采样计算。4.gsa浓缩分数要么是与零的最大偏差(上),要么是两个和之间的差(下)。这两个图显示了在基因表达没有变化的原假设下的两个模拟结果分数(见正文)。算法的输出是一个矩阵,包含每个基因集和样本的路径富集分数。gydF4y2Ba
gsa首先评估一个基因是否gydF4y2Ba我gydF4y2Ba在样品中是高表达还是低表达gydF4y2BajgydF4y2Ba在样本总体分布的背景下。探针效应可以改变微阵列数据中的杂交强度,使得两个非表达基因之间的表达值相差很大[gydF4y2Ba23gydF4y2Ba].类似的基因特异性偏差,如GC含量或基因长度已在RNA-seq数据中描述过[gydF4y2Ba24gydF4y2Ba].为了使不同的表达式配置文件具有相同的规模,表达式级别的统计信息计算如下。每个基因表达谱gydF4y2Ba,对其累积密度函数进行非参数核估计。在微阵列数据的情况下,高斯核([gydF4y2Ba25gydF4y2Ba],第148页)使用:gydF4y2Ba
在哪里gydF4y2BahgydF4y2Ba我gydF4y2Ba是否设置了控制内核估计分辨率的特定于基因的带宽参数gydF4y2BahgydF4y2Ba我gydF4y2Ba=gydF4y2Ba年代gydF4y2Ba我gydF4y2Ba/ 4,gydF4y2Ba年代gydF4y2Ba我gydF4y2Ba是样品的标准差吗gydF4y2Ba我gydF4y2Ba-th基因(图gydF4y2Ba1gydF4y2Ba,步骤1)。对于RNA-seq数据,离散泊松核[gydF4y2Ba26gydF4y2Ba受雇:gydF4y2Ba
在哪里gydF4y2BargydF4y2Ba=0.5,以设置泊松核的模gydF4y2BaxgydF4y2Ba我gydF4y2BakgydF4y2Ba,因为均值为整数的泊松分布的众数gydF4y2BaλgydF4y2Ba发生在gydF4y2BaλgydF4y2Ba而且gydF4y2BaλgydF4y2Ba−1,且取小于gydF4y2BaλgydF4y2Ba当gydF4y2BaλgydF4y2Ba是连续的。gydF4y2Ba
让gydF4y2BazgydF4y2Ba我gydF4y2BajgydF4y2Ba表示以前的表达式级统计信息gydF4y2Ba,或gydF4y2Ba,取决于是否gydF4y2BaxgydF4y2Ba我gydF4y2BajgydF4y2Ba分别为连续微阵列,或离散计数RNA-seq值。下面的步骤通过计算样本富集分数将表达水平的统计数据压缩到基因集中。为了减少潜在异常值的影响,我们首先进行转换gydF4y2BazgydF4y2Ba我gydF4y2BajgydF4y2Ba来排名gydF4y2BazgydF4y2Ba(gydF4y2Ba我gydF4y2Ba)gydF4y2BajgydF4y2Ba对于每个样本gydF4y2BajgydF4y2Ba进一步正常化gydF4y2BargydF4y2Ba我gydF4y2BajgydF4y2Ba= |gydF4y2BapgydF4y2Ba/ 2−gydF4y2BazgydF4y2Ba(gydF4y2Ba我gydF4y2Ba)gydF4y2BajgydF4y2Ba|使秩在零周围对称(图gydF4y2Ba1gydF4y2Ba,步骤2)。这样做是为了在计算最终富集分数时增加排名分布的两个尾部的权重。gydF4y2Ba
我们评估富集分数类似于GSEA和评估方法[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba27gydF4y2Ba]使用Kolmogorov-Smirnov (KS)类似随机漫步统计量(图gydF4y2Ba1gydF4y2Ba,步骤3):gydF4y2Ba
在哪里gydF4y2BaτgydF4y2Ba是一个参数,描述在随机游走(默认gydF4y2BaτgydF4y2Ba= 1),gydF4y2BaγgydF4y2BakgydF4y2Ba是gydF4y2BakgydF4y2Ba-第th基因集,gydF4y2Ba我gydF4y2Ba(gydF4y2BaggydF4y2Ba(gydF4y2Ba我gydF4y2Ba)gydF4y2Ba∈gydF4y2BaγgydF4y2BakgydF4y2Ba)为是否gydF4y2Ba我gydF4y2Ba-th基因(与…对应的基因gydF4y2Ba我gydF4y2Ba排在第3位的表达水平统计量)属于基因集gydF4y2BaγgydF4y2BakgydF4y2Ba、|gydF4y2BaγgydF4y2BakgydF4y2Ba|是基因的数量gydF4y2BakgydF4y2Ba-基因集,和gydF4y2BapgydF4y2Ba是数据集中的基因数量。从概念上讲,情商。gydF4y2Ba3.gydF4y2Ba产生一个基因的分布,以评估基因集中的基因是否更有可能被发现在排名分布的两端(见[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba27gydF4y2Ba]以获取更详细的说明)。gydF4y2Ba
我们提供了两种方法将类似于KS的随机漫步统计量转化为充实统计量(ES)(也称为GSVA评分),即经典的最大偏差法[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba和一个标准化的ES。第一个ES是随机游走离零的最大偏差gydF4y2BajgydF4y2Ba-th个样本gydF4y2BakgydF4y2Ba-th基因集:gydF4y2Ba
对于每个基因集gydF4y2BakgydF4y2Ba,这种方法产生了富集分数的双峰分布(图gydF4y2Ba1gydF4y2Ba步骤4,顶部面板,附加文件gydF4y2Ba1gydF4y2Ba:图S1)。这是KS的一个内在属性,就像随机游走一样,在零分布下产生非零的最大偏差。在GSEA中[gydF4y2Ba4gydF4y2Ba]还观察到,通过排列样本标签得到的经验零分布是双峰的,因此,显著性是使用零分布的正负两边独立确定的。在我们的案例中,我们希望在整个样本群体中通路活性没有变化的零假设下,提供富集分数的标准高斯分布。为此,我们提出了第二个备选分数,它产生了一个接近这一要求的ES分布(图gydF4y2Ba1gydF4y2Ba步骤4,底部面板,附加文件gydF4y2Ba1gydF4y2Ba图S1):gydF4y2Ba
在哪里gydF4y2Ba而且gydF4y2Ba样本的最大正随机游走偏差和最大负随机游走偏差是否分别为零gydF4y2BajgydF4y2Ba还有基因集gydF4y2BakgydF4y2Ba.此统计量可与柯伊伯检验统计量比较[gydF4y2Ba29gydF4y2Ba],将最大和最小偏差相加,使检验统计量在尾部更加敏感。相比之下,我们的测试统计数据惩罚两个尾部都较大的偏差,并通过减去潜在噪声提供富集分数的“归一化”。对于这一统计数据有一个明确的生物学解释,它强调的是仅在一个方向上协调激活的通路中的基因,相对于总体人群,要么是过表达,要么是过表达。对于含有双向作用基因的通路,偏差会相互抵消,很少或没有富集。因为这个统计量是单峰的和近似正态的(通过模拟观察,见下文),因此下游分析可能对数据施加分布假设。在某些情况下,这个统计数据的特征可能是不需要的,特别是如果相关的基因集没有明确地分为“向上”和“向下”行为(如MSigDB对许多基因集提供的那样)。在这种情况下,由Eq定义的统计量。gydF4y2Ba4gydF4y2Ba应该使用。gydF4y2Ba
数字gydF4y2Ba1gydF4y2Ba,步骤4和附加文件gydF4y2Ba1gydF4y2Ba:图S1显示了一个简单的模拟,其中标准高斯偏差是独立采样的gydF4y2BapgydF4y2Ba=2万个基因和gydF4y2BangydF4y2Ba=30个样本,从而模拟基因表达无变化的空分布。100个基因集被均匀随机地从gydF4y2BapgydF4y2Ba大小从10到100个基因不等的基因。利用这两个输入,我们计算最大偏差ES和归一化ES。结果的分布如图所示gydF4y2Ba1gydF4y2Ba,步骤4和附加文件gydF4y2Ba1gydF4y2Ba:图S1。gydF4y2Ba
虽然GSVA算法本身并不评估基因集富集的统计显著性,但使用传统统计模型可以很容易地评估表型的显著性。同样,可以通过排列样本标签(方法)来估计错误发现率。对于显著性或错误发现的阈值,我们没有通用的处方,因为这些选择高度依赖于上下文,并且可能根据每个实验而变化。以下部分将提供这些技术的示例。gydF4y2Ba
结果gydF4y2Ba
其他方法的回顾gydF4y2Ba
基因集富集的方法一般可以按照有监督与无监督、群体与单样本评估的标准进行划分。大多数GSE方法,如GSEA [gydF4y2Ba4gydF4y2Ba],是有监督的和基于群体的,因为他们计算每个基因集的富集分数来描述整个数据集,以表型为模型(离散的,如病例对照或连续)。田先生描述了这种类型中最简单的一种gydF4y2Baet al。gydF4y2Ba[gydF4y2Ba6gydF4y2Ba,gydF4y2Ba19gydF4y2Ba],评估为一组基因的平均差异表达(例如病例vs对照),与那些不在该基因集中的基因进行比较。该方法的主要缺点之一是没有考虑到基因相关性,这可能导致GSEA假阳性基因集数量增加[gydF4y2Ba30.gydF4y2Ba].许多其他受监督的、基于人群的方法也被描述过[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba-gydF4y2Ba34gydF4y2Ba].gydF4y2Ba
在evaluate方法中引入了一种受监督的、基于单样本的方法[gydF4y2Ba27gydF4y2Ba].在基于表型类对样本进行二分类后,evaluate方法计算每个基因/类的密度估计值,然后评估每个样本/基因集的富集分数。这种方法非常适合于评估跨二分类表型的基因集变异。gsa也利用密度估计来评估样本富集,但通过省略表型信息,它可以进行更一般的下游分析,因此应用范围更广。gydF4y2Ba
已经开发了三种无监督的单样本富集方法,基因表达途径水平分析(PLAGE)、单样本GSEA (ssGSEA)和联合z评分[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba35gydF4y2Ba].这些方法计算每个基因集和单个样本的富集分数。PLAGE对样本上的每个基因表达谱进行标准化,然后将每个基因集的途径活性谱估计为基因集奇异值分解的第一个右奇异向量的系数([gydF4y2Ba35gydF4y2Ba], pg. 9).联合z得分法[gydF4y2Ba22gydF4y2Ba如PLAGE,首先将每个基因表达谱标准化为z分数,然后通过组合每个样本的单个基因z分数获得途径活性谱([gydF4y2Ba22gydF4y2Ba,图一)。PLAGE和组合z评分都是参数,并假设基因表达谱共同呈正态分布。合并的z分数还假设每个基因集中的基因独立作用。芭比的ssGSEA方法gydF4y2Baet al。gydF4y2Ba[gydF4y2Ba5gydF4y2Ba]使用基因集内外基因表达序列的经验累积分布函数的差异来计算每个样本的富集统计量,该统计量通过在所有基因集和样本中取的值的范围进一步归一化。gydF4y2Ba
模拟数据的方法比较gydF4y2Ba
GSVA是无监督的,产生单一样品富集分数。因此,我们可以直接将GSVA的性能与组合z-score、单样本GSEA和PLAGE进行比较[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba35gydF4y2Ba].然而,与其他方法相比,gsa首先用ECDF在样本上的核估计计算表达统计量,这应该有助于保护方法不受系统基因特异性效应(如探针效应)的影响,从而提高其灵敏度。为了验证这一假设,我们进行了以下三个模拟研究。gydF4y2Ba
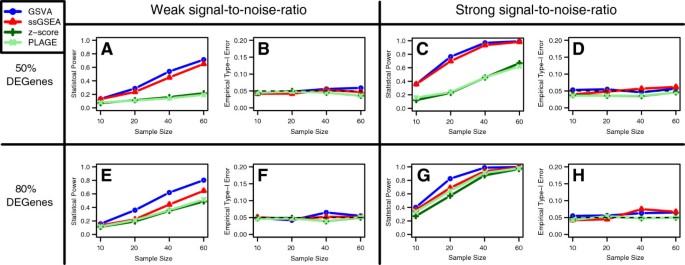
在第一项研究中,我们从线性相加模型模拟微阵列数据与样本和探针效应gydF4y2BapgydF4y2Ba= 1000个基因和两组样本(见方法)。利用该模型,我们生成了样本量不断增加的数据集,并定义了两个由30个基因组成的基因集,其中一个基因集差异表达(DE),另一个基因集没有差异表达(DE)。对于DE基因集,我们考虑了强信噪比和弱信噪比以及DE基因的两个不同部分(50%和80%),从而产生了四种不同的模拟场景。利用每个场景的模拟数据,我们用四种样本GSE方法(GSVA、ssGSEA、PLAGE和组合z评分)计算了通路活性剖面,并应用了agydF4y2BatgydF4y2Ba-检测两组样品DE和非DE基因集。利用DE基因集和显著性阈值gydF4y2BaαgydF4y2Ba=0.05时,我们估计了每种方法的统计威力作为样本量的函数。在同样的数据上,但使用非de基因集,我们估计了经验i型错误率在gydF4y2BaαgydF4y2Ba= 0.05。仿真结果如图所示gydF4y2Ba2gydF4y2Ba表明gsa在四个模拟场景中都比其他三种方法获得了更高的统计能力,同时对i型错误率提供了相似的控制。gydF4y2Ba
gsa、PLAGE、单样本GSEA (ssGSEA)和组合z评分(zscore)的统计效力和i型错误率比较gydF4y2Ba1000次模拟的平均结果被描述为样本大小的函数gydF4y2BaxgydF4y2Ba-轴,对于每一个GSE方法。在gydF4y2BaygydF4y2Ba-axis表示统计幂(A, C, E, G)或经验型i型错误率(B, D, F, H)。的线性相加模型模拟数据,模型具有样本和探针效应(见方法)gydF4y2BapgydF4y2Ba= 1000个基因。用每种方法计算两个基因集的GSE评分,其中一个是差异表达(DE),另一个没有。统计功率和经验型i型错误率估计通过执行gydF4y2BatgydF4y2Ba-检验在DE和非DE基因集上的显著性水平分别为gydF4y2BaαgydF4y2Ba= 0.05。这些模拟是在DE基因集的以下四种不同场景下进行的:(gydF4y2Ba一个gydF4y2Ba,gydF4y2BaBgydF4y2Ba)信噪比较弱,DE基因集中DE基因占50%;(gydF4y2BaCgydF4y2Ba,gydF4y2BaDgydF4y2Ba)信噪比强,DE基因集中50%的DE基因;(gydF4y2BaEgydF4y2Ba,gydF4y2BaFgydF4y2Ba)信噪比弱,DE基因集中80%的DE基因;(gydF4y2BaGgydF4y2Ba,gydF4y2BaHgydF4y2Ba)强信噪比,DE基因集中80%的DE基因。gydF4y2Ba
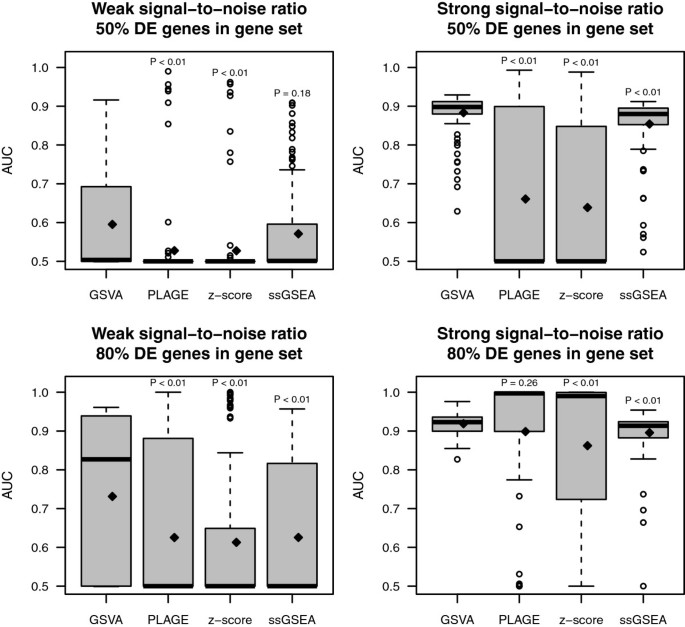
在第二个模拟研究中,我们通过调用DE基因集来比较每种GSE方法识别差异通路活性的准确性。为此,我们使用了前面定义的四种模拟场景以及固定样本量的线性相加模型gydF4y2BangydF4y2Ba= 60,gydF4y2BapgydF4y2Ba=10,000个基因,以模拟更真实维度的数据。我们将前2000个基因设为DE,并模拟1000个基因集,其中定义500个为DE(参见方法)。对于每个模拟的基因表达数据集,计算GSE得分和两个样本gydF4y2BatgydF4y2Ba-test在5% FDR时调用DE基因集。每种GSE方法的性能通过100个独立模拟的ROC曲线下面积(AUC)来衡量(见方法)。从DE调用的二元向量中计算AUC值,以比较每种方法在全基因组显著性水平上识别DE基因集的能力。结果如图所示gydF4y2Ba3.gydF4y2Ba.该图显示,gsa获得的平均AUC值明显高于其他GSE方法(gydF4y2BaPgydF4y2Ba12组成对比较中,除2组外,其余均<0.05)。在更严格的FDR截断值为1%的情况下,也可以观察到gsa比其他方法的性能改善gydF4y2Ba1gydF4y2Ba:图S2)。gydF4y2Ba
gsa、PLAGE、单样本GSEA (ssGSEA)和联合z评分(zscore)差异途径活性鉴定的比较gydF4y2Ba每个面板显示的面积下的ROC曲线(AUC)gydF4y2BaygydF4y2Ba在100次模拟中,每种方法在5% FDR下预测的差异表达基因的-轴(见方法)。在每个箱线图的顶部是gydF4y2BatgydF4y2Ba-test报告GSVA与相应方法的均值无差异。上面的两个面板对应于模拟,其中DE基因集中50%的基因是DE,而底部的两个面板包含DE基因集中80%的DE基因。左边的两个面板对应DE量级中的弱信噪比,而右边的两个面板对应强信噪比。菱形表示箱线图中的平均值。gydF4y2Ba
最后,我们在生存分析的背景下进行了第三次仿真研究。我们再次使用以前的线性相加模型来模拟微阵列数据gydF4y2BapgydF4y2Ba= 1000个基因和两组样本。然而,这一次,我们进行了一项交叉验证研究,以评估使用50个基因集的预测能力,每个基因集包含10个基因。两个样本组之间设置一个基因集为DE,其余49个基因集为非DE,在其他990个基因中均匀随机抽样形成。我们对差异表达的大小(强)和DE基因在DE基因集中的比例(50%)使用了固定的配置。类似于Bair和Tibshirani的生存模拟[gydF4y2Ba36gydF4y2Ba],我们为每组样本用不同的参数生成每个观察的生存时间和审查状态(见方法)。该设置生成两次,以获得独立的训练和测试数据集。gydF4y2Ba
GSE分数分别根据训练和测试数据计算。对训练数据中的每个GSE评分剖面拟合Cox比例风险模型(Cox PHM)。用Wald检验提供的p值最低的模型对试验数据进行风险预测。作为基线比较,我们还为训练数据上的每个基因表达谱拟合了Cox PHM,并选择了10个基因,对应于所有基因集的基因集大小,Wald检验给出的最低p值也可以预测测试数据上的风险。gydF4y2Ba
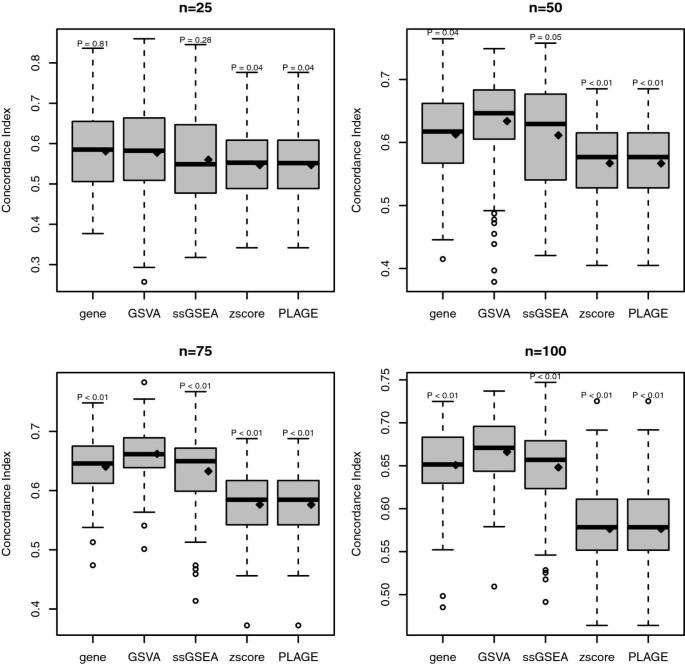
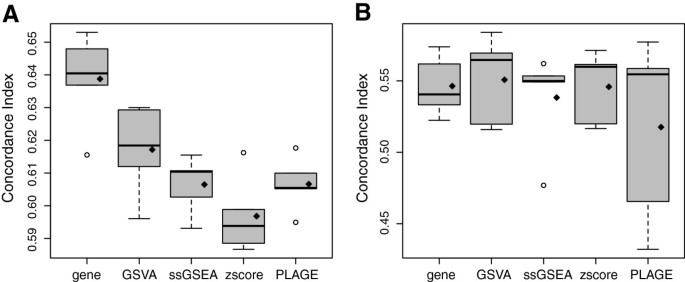
用一致性指数评价各基因集和基因级模型(使用10个基因)对试验数据的性能。这个模拟重复了100次,在增加样本量的情况下执行了4次完整的运行gydF4y2BangydF4y2Ba={25,50,75,100}的模拟数据。在图gydF4y2Ba4gydF4y2Ba对每种方法和样本量分别报告一致性指数值的分布。在四个样本量中,GSVA比其他方法提供了更高的均值和中位数一致性指数值,且均值差异显著(gydF4y2BaPgydF4y2Ba< 0.05)当gydF4y2BangydF4y2Ba≥50岁。gydF4y2Ba
淋巴母细胞白血病:ALL vs MLLgydF4y2Ba
通路中心方法的一个典型应用是研究通路或基因集的变化如何揭示与给定表型相关的潜在生物结构。这类分析的一个例子是Verhaak,gydF4y2Ba等gydF4y2Ba[gydF4y2Ba28gydF4y2Ba在那里,他们展示了老鼠来源的神经元基因集如何在一个大的人类队列中显示出胶质母细胞瘤亚型的对应结构。为了评估gsa检测与真实数据中感兴趣的表现型相关的差异表达基因集的更高能力,我们使用了人类白血病数据集。数据集包含37个不同的白血病个体,其中20个对应于急性淋巴细胞白血病(ALL), 17个对应于混合血统白血病(MLL) [gydF4y2Ba37gydF4y2Ba].我们评估了四种样本GSE方法的性能,通过评估它们在不同表达变化幅度的情况下产生表型ALL与MLL的签名的能力。gydF4y2Ba
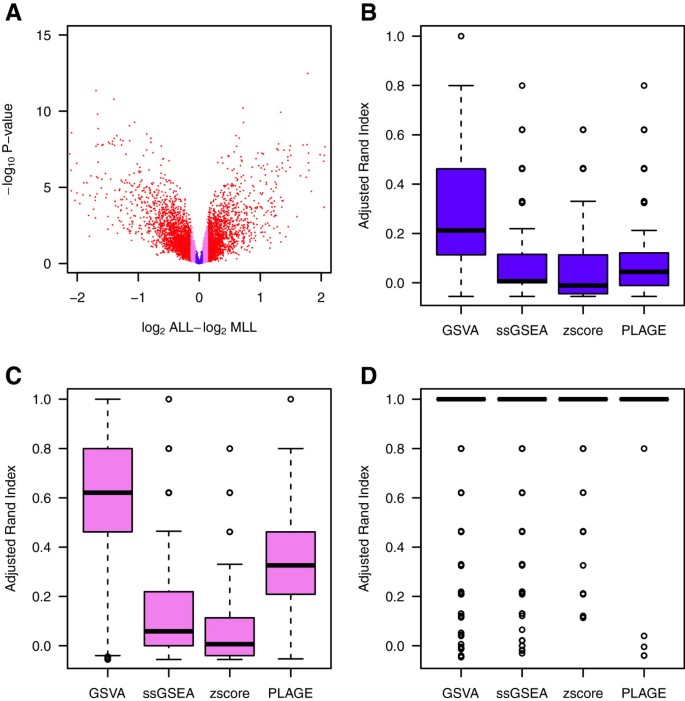
我们一开始根据折叠变化对所有基因进行排序。然后,我们将这一排名划分为三个大小相同的分数,分别用红色、紫色和蓝色表示gydF4y2Ba5gydF4y2Ba我们使用每一倍变化增加的基因,并从每个类中引导10个样本1000次。我们将四种GSE方法应用于引导数据以及标准的Broad C2基因集[gydF4y2Ba4gydF4y2Ba].随后,我们使用limma [gydF4y2Ba38gydF4y2Ba].从每个调整后的p值排序中,我们选择了前5个基因集,并使用它们的富集分数对样本进行分层聚类。最后,我们利用层次结构的两个主要分支将样本分成两组,并计算调整后的兰特指数(ARI) [gydF4y2Ba39gydF4y2Ba],以评估聚类的鲁棒性。gydF4y2Ba
白血病数据集上gsa、PLAGE、单样本GSEA (ssGSEA)和联合z评分(zscore)差异通路活性鉴定的比较gydF4y2Ba(gydF4y2Ba一个gydF4y2Ba)白血病数据集中基因表达变化的火山图。红色标记的基因形成了绝对折叠变化最大的第一个毛胞,紫色表示第二个毛胞,蓝色表示第三个毛胞。(gydF4y2Ba罪犯gydF4y2Ba)调整兰德指数(ARI),表示通过对前5个差异激活基因集上各比较方法产生的富集分数进行分层聚类对两组样本进行分类的准确性。ARI值的分布是通过从每个样本组中引导1000乘以10个样本形成的。颜色与(A)火山图中给出的基因的键相匹配,表明如预期的那样,折叠变化越大的基因导致ARI值越大。然而,当折叠变化很小时(gydF4y2BacgydF4y2Ba)和潜在的特征变得非常微妙,gsa产生富集分数,导致差异激活的基因集,这比使用ssGSEA, zscore或PLAGE更好地分类两个样本组。gydF4y2Ba
如图gydF4y2Ba5gydF4y2Ba由图可知,ARI值取决于考虑的折叠变化幅度的量级。除了具有最大倍数变化的三倍体基因(图D)外,gsa产生的富集分数导致显著较高的ARI值(gydF4y2BatgydF4y2Ba-检验均值p值<2的差异gydF4y2BaegydF4y2Ba−16)优于ssGSEA、PLAGE或联合z评分方法,表明gsa在产生能够检测微妙基因表达变化的签名方面具有更大的能力。样本富集分数很容易将这种分析扩展到具有三个或更多样本组的更复杂的表型。这样一个使用肾上腺皮质癌数据的例子可以在附加文件中找到gydF4y2Ba1gydF4y2Ba:图S3和表S1。gydF4y2Ba
卵巢癌的生存分析gydF4y2Ba
我们接下来检查了预测卵巢浆液性囊腺癌(OV)患者生存的途径模型。我们使用了一个大型基因表达实验(gydF4y2BangydF4y2Ba=588)从TCGA [gydF4y2Ba40gydF4y2Ba]获取MSigDB中每个典型基因集(C2)的途径富集评分,并比较四种GSE方法。我们使用四种比较方法分别对数据的每个训练和测试分区进行了五次交叉验证,并分别计算GSE得分。我们还考虑了原始的表达数据为一个简单的基因级模型。在每个训练数据集上,我们在基因级模型中为每个基因集和每个基因拟合一个Cox PHM。然后,我们选择了这5个基因集,或基因水平模型中的基因,Wald检验的p值最低,对生存没有影响。使用选定的基因集,我们再次在训练数据上拟合Cox PHM,并使用它预测GSE得分训练和测试数据集的风险。我们在基因层面的模型中重复了这一过程。最后,我们通过计算预测风险的一致性指数来评估这些模型的预测性能,每个模型代表不同的方法。如图gydF4y2Ba6gydF4y2Ba表明,除了使用基因级模型的训练数据集外,在训练和测试数据集中,gsa均比其他方法获得了更高的平均和中位数一致性指数值。gydF4y2Ba
通路中心方法的主要好处之一是它们在理解疾病机制方面提供了可解释性。在表gydF4y2Ba1gydF4y2Ba,我们列出了由gsa识别的与生存相关的顶级基因集(完整的列表可在附加文件中获得)gydF4y2Ba1gydF4y2Ba:表S2)。通过100次随机排序样本标签(患者生存时间),使用基于排列的方法重新估计错误发现率(FDR),结果对p值阈值为10的FDR估计为0.05和0.2gydF4y2Ba−4gydF4y2Ba和5·10gydF4y2Ba−3gydF4y2Ba,分别。排名第一和第二的基因集分别表明了两种重要的生存机制:DNA修复和先天免疫和适应性免疫的调节。进一步检查顶部显著基因集(gydF4y2BaPgydF4y2Ba<10gydF4y2Ba−3gydF4y2Ba)表明它们中的许多参与了伤口和免疫反应。有趣的是,排在第3和第13位的基因集来自对维甲酸治疗的反应特征,维甲酸是一种全反式维甲酸药物,已被证明可以抑制卵巢癌细胞系的生长[gydF4y2Ba41gydF4y2Ba]gydF4y2Ba,gydF4y2Ba[gydF4y2Ba42gydF4y2Ba].最后,在前20个基因集中,我们注意到一些EGF和RAS相关通路的存在。虽然EGFR和RAS突变在卵巢癌中并不常见[gydF4y2Ba43gydF4y2Ba],这些被充分研究的癌基因的激活可能仍然在卵巢癌的进展和生存中发挥重要作用。gydF4y2Ba
RNA-seq数据的gsagydF4y2Ba
应用高通量测序来测定生物样品中的RNA浓度,通常称为RNA-seq,正稳步成为基因表达谱分析的首选技术[gydF4y2Ba44gydF4y2Ba].由此产生的基于序列的测量采用离散计数数据的形式,产生比微阵列技术更大的动态范围和无偏功率来调查整个转录组的细胞状态。然而,这些数据的性质往往需要特定的统计模型和生物信息学方法来进行分析,如差异表达分析[gydF4y2Ba45gydF4y2Ba].这也是许多为微阵列数据开发的GSE方法的情况,这些方法的分布假设排除了它们直接应用于RNA-seq计数数据的可能性[gydF4y2Ba1gydF4y2Ba]gydF4y2Ba,gydF4y2Ba[gydF4y2Ba46gydF4y2Ba].gydF4y2Ba
据我们所知,还没有人尝试将基因级别的RNA-seq表达谱压缩到基因集中,以捕捉基因表达的微妙变化。存在的GSE方法,要么处理差异表达基因的封闭列表(如topGO [gydF4y2Ba47gydF4y2Ba, GOseq [gydF4y2Ba48gydF4y2Ba]),或对某些差异表达统计数据进行排序,如GSEA [gydF4y2Ba4gydF4y2Ba]和平均秩基因集富集法[gydF4y2Ba49gydF4y2Ba].GOseq [gydF4y2Ba48gydF4y2Ba专门设计用于解决从RNA-seq数据中获得的差异表达基因列表中的基因长度偏差。但是GOseq忽略了那些不被认为是差异表达的基因,并将它们从分析中删除,因此忽略了有细微变化的基因。此外,基于排名的方法忽略了通路中基因的相对变化,从而导致对基因的平等处理,尽管它们可能有不同的折叠变化[gydF4y2Ba50gydF4y2Ba].因此,这些方法可能不足以检测通路活性的细微变化。gydF4y2Ba
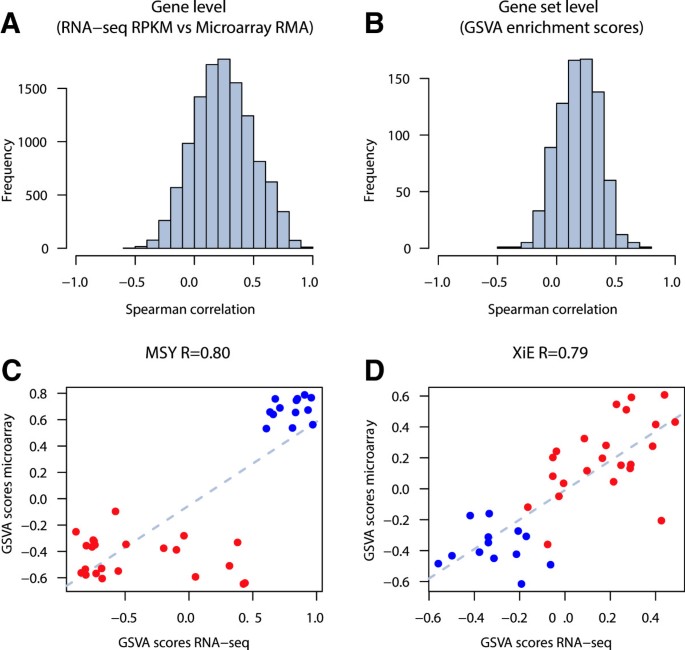
在这里,我们展示了如何将gsa应用到RNA-seq数据。我们通过使用HapMap个体的淋巴母细胞系(LCL)样本,提供了类似于从微阵列数据中获得的途径活性概况,这些样本使用了两种技术[gydF4y2Ba51gydF4y2Ba]gydF4y2Ba,gydF4y2Ba[gydF4y2Ba52gydF4y2Ba].对Microarray和RNA-seq数据进行处理,得到匹配基因和样本标识符的基因表达数据矩阵(方法)。RNA-seq数据包括两个计数表,这些计数来自于两个不同的测序中心,分别由Argonne和Yale表示;参见[gydF4y2Ba52gydF4y2Ba].我们计算了来自两种技术的所有基因和基因集的斯皮尔曼相关性。得到的相关值分布如图所示gydF4y2Ba7gydF4y2Ba,面板A和B,使用Argonne RNA-seq数据(见附加文件gydF4y2Ba1gydF4y2Ba:图S4为Yale RNA-seq数据的类似结果)。我们发现GSVA富集分数与两种分析技术产生的基因表达水平相似。gydF4y2Ba
RNA-seq的gsa (Argonne)。gydF4y2Ba一个。gydF4y2BaRNA-seq基因表达谱与微阵列数据之间的Spearman相关值分布。gydF4y2BaB。gydF4y2BaRNA-seq计算的基因集gsa富集分数与微阵列数据之间的Spearman相关值分布。gydF4y2BaCgydF4y2Ba而且gydF4y2BaD。gydF4y2Ba从微阵列和RNA-seq数据中获得的两个包含性别特异性表达基因的基因集的gsa富集分数的比较:由Y染色体男性特异性区域的基因形成的MSY(男性特异性),和由女性中x失活的基因形成的XiE(女性特异性)。红色和蓝色的点分别代表女性和男性样本。在这两种情况下,GSVA分数显示两种谱分析技术之间的高度相关性,其中女性样本在女性特异性基因集中表现出更高的富集分数,而男性样本在男性特异性基因集中表现出更高的富集分数。gydF4y2Ba
我们还详细检查了两个包含性别特异性基因的基因集:在女性样本中逃避x -失活的基因[gydF4y2Ba53gydF4y2Ba和位于Y染色体男性专用区域的基因[gydF4y2Ba54gydF4y2Ba].数字gydF4y2Ba7gydF4y2Ba说明微阵列和RNA-seq富集分数在这些基因集中非常相关,与gydF4y2BaρgydF4y2Ba男性特异性基因集=0.82gydF4y2BaρgydF4y2Ba女性特异性基因组=0.78。男性和女性样本在相应的基因集中表现出更高的GSVA富集分数。这证明了GSVA的灵活性,可以在来自微阵列和RNA-seq技术的数据中实现类似的无监督和单样本GSE分析。gydF4y2Ba
方法gydF4y2Ba
模拟gydF4y2Ba
仿真研究使用以下线性相加模型模拟归一化微阵列数据gydF4y2BapgydF4y2Ba基因和gydF4y2BangydF4y2Ba样本分为两组,代表病例对照情况:gydF4y2Ba
在哪里gydF4y2Ba基因特异性效应,如探针效应,是否具有gydF4y2Ba,gydF4y2Ba样品效果与gydF4y2BajgydF4y2Ba= 1, 2,gydF4y2Ba对应于随机噪声。gydF4y2Ba
在评估统计能力和图中的i型错误时gydF4y2Ba2gydF4y2Ba,我们设定gydF4y2BapgydF4y2Ba= 1000个基因,其中前30个被认为是DE基因集,后30个是非DE基因集。我们考虑了四种不同的样本量gydF4y2BangydF4y2Ba={10,20,40,60}和两个不同的条件导致了四个不同的模拟场景:基因集中差异表达基因的比例(50%和80%)和信噪比表示为样本组中一个DE基因的平均样本效应的大小(弱信噪比和强信噪比)。对于非de基因gydF4y2BaμgydF4y2Ba1gydF4y2Ba=gydF4y2BaμgydF4y2Ba2gydF4y2Ba= 0和gydF4y2BaσgydF4y2Ba1gydF4y2Ba=gydF4y2BaσgydF4y2Ba2gydF4y2Ba=1,为DE基因gydF4y2BaμgydF4y2Ba2gydF4y2Ba=0.5表示效果弱,gydF4y2BaμgydF4y2Ba2gydF4y2Ba=1表示强烈的效果和gydF4y2BaσgydF4y2Ba2gydF4y2Ba= 0.5。利用Eq.(中的模型gydF4y2Ba6gydF4y2Ba)使用这些参数,我们模拟了1000个独立的数据集。对于四种GSE方法的每一种,我们都得到了两个基因集(DE和非DE)的GSE评分矩阵gydF4y2BangydF4y2Ba样本。在每个GSE评分矩阵上,我们执行了两个样本gydF4y2BatgydF4y2Ba-对两组样本的两组基因集的平均值差异的检验(gydF4y2BaHgydF4y2Ba0gydF4y2Ba:gydF4y2BaμgydF4y2Ba1gydF4y2Ba−gydF4y2BaμgydF4y2Ba2gydF4y2Ba=0)具有显著性gydF4y2BaαgydF4y2Ba= 0.05。然后估计统计功率为1减去DE基因集的非排斥比例,经验型i型误差估计为1000次模拟中非DE基因集的排斥比例。gydF4y2Ba
在第二个模拟研究中,我们考虑gydF4y2BapgydF4y2Ba= 10000个基因,其中2000个设置为DE, 1000个构建基因集,其中500个为DE。使用前面描述的参数和模拟场景对DE基因和基因集进行模拟。非de基因集通过采样模拟gydF4y2BapgydF4y2Ba= 10000个基因均匀随机,DE基因集通过在DE和非DE基因中按相应场景定义的比例(50%或80% DE基因)进行抽样模拟。对于每个场景,我们以这种方式对数据进行100次采样,并对每个结果数据集使用四种GSE方法计算GSE得分。使用这些GSE评分,我们进行了两个样本gydF4y2BatgydF4y2Ba-对每个基因集进行测试,并将满足5%和1% FDR截断值的称为DE。通过使用R包计算ROC曲线和AUC值来评估性能gydF4y2BaROCRgydF4y2Ba[gydF4y2Ba55gydF4y2Ba].gydF4y2Ba
模拟研究评估GSE评分对生存率的预测能力如图所示gydF4y2Ba4gydF4y2Ba方程中采用线性相加模型进行求解。gydF4y2Ba6gydF4y2Ba,在那里gydF4y2BaμgydF4y2Ba2gydF4y2Ba其中一个样本组DE基因为=1。存活时间由两个正态分布为每个样本组生成gydF4y2Ba而且gydF4y2Ba.审查时间由正态分布产生gydF4y2Ba.当审查时间小于存活时间时,样本被认为被审查。gydF4y2Ba
数据gydF4y2Ba
差异表达分析的数据来自以下来源:gydF4y2Ba37gydF4y2Ba) (gydF4y2Bahttp://www.broadinstitute.orggydF4y2Ba)及肾上腺皮质癌[gydF4y2Ba56gydF4y2Ba) (gydF4y2Bahttp://www.ncbi.nlm.nih.gov/geogydF4y2Ba;GSE10927)。卵巢分析数据于2011年4月从TCGA下载。在分析时,389个样本有临床资料、基因表达(Affy U133A)和CNV (Affy SNP 6.0)。所有病例均使用TCGA 3级数据。基因表达数据使用ComBat批量校正[gydF4y2Ba57gydF4y2Ba].RNA-seq数据对应于HapMap [gydF4y2Ba58gydF4y2Ba约鲁巴人的淋巴母细胞系[gydF4y2Ba52gydF4y2Ba]及已处理的计数表下载于gydF4y2Bahttp://eqtl.uchicago.edu/RNA_Seq_data/resultsgydF4y2Ba.匹配微阵列样本是黄和同事更大研究的一部分[gydF4y2Ba51gydF4y2Ba) (gydF4y2Bahttp://www.ncbi.nlm.nih.gov/geogydF4y2Ba;GSE7792)。gydF4y2Ba
微阵列数据处理gydF4y2Ba
数据分析使用R [gydF4y2Ba59gydF4y2Ba]和Bioconductor [gydF4y2Ba60gydF4y2Ba)软件。我们使用affyPLM选择通过质量控制的芯片[gydF4y2Ba61gydF4y2Ba].AffyPLM在探针集水平上拟合模型,以识别质量较低的芯片。计算相对对数表达式(RLE)值(将每个阵列上的探针表达式与所有阵列上的中值表达式进行比较)和归一化无标度标准误差(NUSE)(为每个基因获得的标准误差估计并跨阵列进行标准化),并应用截断去除低质量样本。gydF4y2Ba
处理批次与感兴趣结果混淆的芯片不考虑在分析中。使用鲁棒多阵列平均(RMA)算法对每个剩余的Affymetrix芯片进行背景调整、归一化和log2转换[gydF4y2Ba62gydF4y2Ba].gydF4y2Ba
如果基因的表达没有超过微阵列的检测水平,或者其表达值在样本中具有有限的可变性,则不能提供鉴别力,并可能影响后续分析的统计力。因此,除LCL微阵列数据外,我们删除了50%样本中通过四分位范围(IQR)测量的变异性较低的基因。gydF4y2Ba
RNA-seq数据处理gydF4y2Ba
皮克勒尔的rna序列数据gydF4y2Baet al。gydF4y2Ba(2010) (gydF4y2Ba52gydF4y2Ba]是在Argonne和Yale两个测序中心制作的,并由作者预处理成两个独立的表,分别由80和81个样本计数41466个集合基因。我们使用这两个计数表,并向读者推荐有关读取映射和基因级计数摘要的详细信息,请参阅出版物的方法[gydF4y2Ba52gydF4y2Ba].一些样本(11个来自阿贡,12个来自耶鲁)在每个测序中心进行了两次测序。在这些情况下,我们保留了较深覆盖的样本,在每个表上获得了69个样本的最终数量。我们进一步过滤了低表达的基因,丢弃了那些以log2尺度计算的平均每百万计数少于0.5个的基因,结果69个样本的表中有17607个基因(Argonne)和17843个基因(Yale),我们保留了两个表中的基因(17324)。接下来,我们将这两个计数表归一化,调整基因长度和G+C含量使用Bioconductor包cqn [gydF4y2Ba24gydF4y2Ba].从下载计数表的同一站点的数据中提取相应的基因长度和G+C含量信息。gydF4y2Ba
为了进一步比较微阵列和RNA-seq数据之间的GSVA富集分数,我们进一步过滤了这两个归一化计数表,以便与黄和同事的LCL微阵列数据处理后得到的基因和样本相匹配[gydF4y2Ba51gydF4y2Ba].这一步首先需要将integrl基因标识符转换为Entrez基因标识符,然后在微阵列和RNA-seq数据之间匹配基因和样本标识符。经过这两个步骤,我们得到了本文分析的两张最终计数表,共有36个样本11,508个Entrez基因,其中23个属于女性,13个属于男性。gydF4y2Ba
基因库gydF4y2Ba
在所有的实验中,我们使用的基因集数据库来自分子特征数据库版本3 (MSigDB) C2集合(策展路径)[gydF4y2Ba4gydF4y2Ba]包含833个典型途径和2392个化学和基因干扰,除非另有说明。在将实验中的基因映射到基因集数据库后,我们会忽略所有小于10个或大于500个基因的基因集。gydF4y2Ba
FDR和多重假设修正gydF4y2Ba
在大多数实验中,我们使用置换方法在指定的p值阈值处估计经验FDR。在一些案例中,我们报告了基于Benjamini-Hochberg (B.H.)方法的多重假设修正[gydF4y2Ba63gydF4y2Ba,以获得校正后的p值。一般来说,对基因集的多重假设修正是有问题的,因为许多基因集高度重叠,因此不仅相关,而且本质上是重复的。我们使用B.H.可能是对罗斯福的保守估计,因此主要用作统计力量的演示。gydF4y2Ba
讨论gydF4y2Ba
对模拟和真实数据的分析表明,在鉴别差异通路活性和存活分析的背景下,GSVA在跨样本的通路变异建模方面优于竞争方法。然而,鉴于生物信息界已经发表和可用的大量GSE方法,gsa可能不是每个表达数据集的最佳工具。我们推荐gsa作为一种中间通用工具,为更开放的生物学分析提供途径活性的摘要。对于特定的应用,为解决领域特定问题而优化的高度专门化算法可能优于gsa。用户还应该意识到,gsa算法中的非参数密度估计需要足够多的观测数据,根据我们在图中的统计能力分析gydF4y2Ba2gydF4y2Ba,应该大于gydF4y2BangydF4y2Ba= 10。gydF4y2Ba
在高通量实验中对基因进行非特异性筛选已被证明可以增加检测基因表达水平显著变化的统计能力[gydF4y2Ba64gydF4y2Ba这一观察结果很可能在基因集水平上成立。我们使用了一种简单的非特异性过滤策略,即在基因表达数据和基因集之间匹配基因标识符后,对基因集的大小进行最小和最大截断。然而,其他基于生物相关基因集预期特征的策略可能更有帮助。例如,属于同一基因集或通路的基因更有可能协调表达,并有望表现出某种程度的相关性。包含相关基因的基因集比不相关、不相关的基因集更具一致性,并提供更高的生物信号[gydF4y2Ba65gydF4y2Ba].因此,去除功能不一致的通路可以构成一种具有吸引力的非特异性过滤策略,以提高基因集水平的检测能力。gydF4y2Ba
结论gydF4y2Ba
我们提出了一种方法,以测定基因集富集的变异在一个样本群体。该方法可作为R的生物导体包,在GSVA的名称下免费获得gydF4y2Bahttp://www.bioconductor.orggydF4y2Ba.具有多种检测方法和复杂表型的大型数据集的日益增加的可用性激励了我们的工作,因为在通路背景下对这些数据集的研究对理解它们至关重要。gsa方法是非参数和无监督的,并且绕过了在富集评分算法中显式建模表型的传统方法。我们还展示了GSVA可以很容易地适应于RNA-seq数据的分析,产生类似于其微阵列对应的结果。在附加文件中gydF4y2Ba1gydF4y2Ba, gsa应用的另外两个例子可以在多类肾上腺皮质癌数据集中发现,包括差异途径分析(附加文件gydF4y2Ba1gydF4y2Ba:图S3和表S1),卵巢癌的通路和拷贝数改变的相关性分析(附文献)gydF4y2Ba1gydF4y2Ba:图S5)。gydF4y2Ba
对于未来的方向,我们认为gsa可能被用于类似于eQTL映射的遗传基因组学策略中,我们可以称之为路径- qtl,以识别影响路径活性的DNA多态性[gydF4y2Ba66gydF4y2Ba].这可以进一步扩展以支持因果推理[gydF4y2Ba67gydF4y2Ba,其中通路取代了基因,建立了基因型→基因表达→表型的因果链。gydF4y2Ba
可用性和需求gydF4y2Ba
项目名称:gydF4y2BaGSVAgydF4y2Ba
项目主页:gydF4y2Bahttp://www.bioconductor.org/packages/release/bioc/html/GSVA.htmlgydF4y2Ba
操作系统:gydF4y2Ba平台无关的gydF4y2Ba
编程语言:gydF4y2BaR、CgydF4y2Ba
其他要求:gydF4y2BaR (>= 2.15.0), R包方法,Bioconductor包GSEABase (>= 1.18.0)gydF4y2Ba
许可:gydF4y2BaGPL (>= 2)gydF4y2Ba
非学术人士使用的限制:gydF4y2Ba没有限制gydF4y2Ba
参考文献gydF4y2Ba
Goeman JJ, Geer SAvd, Kort Fd, Houwelingen HCv:一种基因组的全局检测:检测与临床结果的相关性。生物信息学,2004,20:93-99。[gydF4y2Bahttp://bioinformatics.oxfordjournals.org/content/20/1/93.abstractgydF4y2Ba]gydF4y2Ba
Mootha VK、Lindgren CM、Eriksson KF、Subramanian A、Sihag S、Lehar J、Puigserver P、Carlsson E、Ridderstråle M、Laurila E、Houstis N、Daly MJ、Patterson N、Mesirov JP、Golub TR、Tamayo P、Spiegelman B、Lander ES、Hirschhorn JN、Altshuler D、Groop LC:参与氧化磷酸化的pgc -1 α响应基因在人类糖尿病中协调下调。自然与生物学报,2003,34(3):267-273。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/12808457gydF4y2Ba]gydF4y2Ba
Sweet-Cordero A, Mukherjee S, Subramanian A, You H, Roix JJ, ladda - acosta C, Mesirov J, Golub TR, Jacks T:跨物种基因表达分析鉴定的KRAS2致癌表达特征。自然科学,2005,37:48-55。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/15608639gydF4y2Ba]gydF4y2Ba
Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA, Paulovich A, Pomeroy SL, Golub TR, Lander ES, Mesirov JP:基因集富集分析:一种基于知识的解释全基因组表达谱的方法。美国国家科学研究院。中国科学:d辑,2005,38(4):359 - 359。[gydF4y2Bahttp://www.pnas.org/content/102/43/15545.abstractgydF4y2Ba]gydF4y2Ba
Barbie DA、Tamayo P、Boehm JS、Kim SY、Moody SE、Chan EM、Schinzel AC、Sandy P、Meylan E、Scholl C、Fröhling S、Chan EM、Sos ML、Michel K、Mermel C、Silver SJ、Weir BA、Reiling JH、Sheng Q、Gupta PB、Wadlow RC、Le H、Hoersch S、Wittner BS、Ramaswamy S、Livingston DM、Sabatini DM、Meyerson M、Thomas RK、Lander ES、Mesirov JP、Root DE、Gilliland DG、Jacks T、Hahn WC:系统RNA干扰揭示kras驱动的致癌癌症需要TBK1。自然学报,2009,462(7269):108-112。[gydF4y2Bahttp://www.nature.com/nature/journal/v462/n7269/abs/nature08460.htmlgydF4y2Ba]gydF4y2Ba
田玲,Greenberg SA,孔文伟,Altschuler J, Kohane IS, Park PJ:表达谱研究中发现具有统计学意义的通路。美国国家科学研究院。中国科学:d辑,2005,38(4):359 - 359。[gydF4y2Bahttp://www.pnas.org/content/102/38/13544gydF4y2Ba]gydF4y2Ba
Barry WT, Nobel AB, Wright FA:基因表达研究中功能类别的显著性分析:结构化排列方法。生物信息学,2005,21(9):1943-1949。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/15647293gydF4y2Ba]gydF4y2Ba
Efron B, Tibshirani R:关于测试基因组的意义。应用统计,2006,1(1):107-129。[gydF4y2Bahttp://arxiv.org/abs/math/0610667gydF4y2Ba]gydF4y2Ba
Dørum G, Snipen L, Solheim M, Sæbø S:用于小型直接比较实验的基因集富集分析中的旋转检测。Stat Apps Gen Mol Bio. 2009, 8: [gydF4y2Bahttp://www.bepress.com/sagmb/vol8/iss1/art34gydF4y2Ba]gydF4y2Ba
Irizarry RA,王超,周燕,Speed TP:简化基因集富集分析。统计方法医学杂志2009,18(6):565-575。[gydF4y2Bahttp://smm.sagepub.com/content/18/6/565.abstractgydF4y2Ba]gydF4y2Ba
蒋铮,R先生:基因集富集的延伸。生物信息学,2007,23(3):306-313。[gydF4y2Bahttp://bioinformatics.oxfordjournals.org/content/23/3/306.abstractgydF4y2Ba]gydF4y2Ba
Wu D, Lim E, Vaillant F, Asselin-Labat ML, Visvader JE, Smyth GK: ROAST:用于复杂微阵列实验的旋转基因集测试。生物信息学(牛津,英国)。2010, 26(17): 2176-2182。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/20610611gydF4y2Ba].(PMID: 20610611)gydF4y2Ba
Lamb J、Ramaswamy S、Ford HL、Contreras B、Martinez RV、Kittrell FS、Zahnow CA、Patterson N、Golub TR、Ewen ME:细胞周期蛋白D1在人类癌症基因表达模式中的作用机制中国生物医学工程学报,2004,23(3):323-334。[gydF4y2Bahttp://www.cell.com/abstract/s0092 - 8674 (03) 00570 - 1gydF4y2Ba]gydF4y2Ba
Shepard JL, Amatruda JF, Stern HM, Subramanian A, Finkelstein D, Ziai J, Finley KR, Pfaff KL, Hersey C, Zhou Y, Barut B, Freedman M, Lee C, Spitsbergen J, Neuberg D, Weber G, Golub TR, Glickman JN, Kutok JL, Aster JC, Zon LI:斑马鱼bmyb突变导致基因组不稳定和癌症易感增加。美国国家科学研究院。中国科学:地球科学,2015,38(4):359 - 359。[gydF4y2Bahttp://www.pnas.org/content/102/37/13194.abstractgydF4y2Ba]gydF4y2Ba
Segrè AV, grop L, Mootha VK, Daly MJ, Altshuler D, Consortium D:线粒体基因中常见的遗传变异与2型糖尿病或相关血糖特征的关系不丰富。科学通报,2010,6 (8):e1001058-[gydF4y2Bahttp://dx.doi.org/10.1371/journal.pgen.1001058gydF4y2Ba]gydF4y2Ba
Pece S, Tosoni D, Confalonieri S, Mazzarol G, Vecchi M, Ronzoni S, Bernard L, Viale G, Pelicci PG, Fiore PPD:乳腺癌的生物学和分子异质性与其癌症干细胞含量相关。中国生物医学工程学报,2010,30(4):447 - 447。[gydF4y2Bahttp://www.sciencedirect.com/science/article/B6WSN-4Y3TDSF-D/2/9fd74fc1accc422d7a6e6d935b45975cgydF4y2Ba]gydF4y2Ba
洪建华,杨浩,胡铮,翁智,DeLisi C:基因集富集分析:性能评价和使用指南。生物信息学,2012,13(3):281-291。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/21900207gydF4y2Ba].(PMID: 21900207)gydF4y2Ba
Goeman JJ, Bühlmann P:从基因集的角度分析基因表达数据:方法问题。生物信息学(牛津,英国)。2007,23(8): 980-987。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/17303618gydF4y2Ba].(PMID: 17303618)gydF4y2Ba
PAGE:基因集富集的参数分析。生物信息学,2005,6:144-[PMID: 15941488 PMCID: 1183189]gydF4y2Ba
Tenenbaum JD, Walker MG, Utz PJ, Butte AJ:基于表达的通路特征分析(EPSA):挖掘公开可用的微阵列数据,以洞察人类疾病。医学基因组学杂志,2008,1:51-[gydF4y2Bahttp://www.biomedcentral.com/1755-8794/1/51gydF4y2Ba]gydF4y2Ba
Creighton CJ:在人类前列腺肿瘤中,多种致癌途径信号显示协同表达模式。科学通报,2008,3 (3):e1816-[gydF4y2Bahttp://dx.doi.org/10.1371/journal.pone.0001816gydF4y2Ba]gydF4y2Ba
李娥,庄海海,金jw, Ideker T, Lee D:推断疾病精确分类的途径活性。科学通报,2008,4 (11):e1000217-[gydF4y2Bahttp://dx.doi.org/10.1371/journal.pcbi.1000217gydF4y2Ba]gydF4y2Ba
Zilliox MJ, Irizarry RA:用于微阵列数据的基因表达条形码。化学学报,2007,4(11):911-913。[gydF4y2Bahttp://dx.doi.org/10.1038/nmeth1102gydF4y2Ba]gydF4y2Ba
吴铮,刘志强,刘志强,吴志强:用条件分位数归一化方法去除RNA-seq数据中的技术变异。生物统计学,2012,[gydF4y2Bahttp://biostatistics.oxfordjournals.org/content/early/2012/01/24/biostatistics.kxr054.abstractgydF4y2Ba]gydF4y2Ba
Silverman BW:统计和数据分析的密度估计。1986,伦敦:查普曼和霍尔,[gydF4y2Bahttp://www.crcpress.com/product/isbn/9780412246203gydF4y2Ba].(ISBN 9780412246203)gydF4y2Ba
Canale A, Dunson DB:贝叶斯核混合计数。中国科学(d辑),2011,38(4):361 - 361。gydF4y2Ba
Edelman E, Porrello A, Guinney J, Balakumaran B, Bild A, Febbo PG, Mukherjee S:样本集富集评分分析:在全基因组表达谱中分析单个样本的基因集富集。生物信息学,2006,22 (14):e108-e116。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/16873460gydF4y2Ba]gydF4y2Ba
王Verhaak RGW Hoadley KA, Purdom E, V,气Y,威尔克森博士米勒CR,丁L, Golub T, Mesirov JP, Alexe G,劳伦斯•米凯利M, Tamayo P,堰英航,加布里埃尔年代,Winckler W,古普塔年代,Jakkula L, Feiler HS,霍奇森詹,詹姆斯CD, Sarkaria约,布伦南C,卡恩,首位PT,威尔逊RK,速度TP,灰色JW, Meyerson M,斯坦利·G, Perou厘米,海耶斯DN:整合基因组分析确定临床相关亚型PDGFRA胶质母细胞瘤表现为异常的IDH1,表皮生长因子受体,NF1。中华癌症杂志,2010,17:98-110。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/20129251gydF4y2Ba]gydF4y2Ba
皮尔逊E:对直线上各点随机性检验的比较。生物识别技术,2003,19(5):557 - 557。gydF4y2Ba
Tamayo P, Steinhardt G, Liberzon A, Mesirov JP:基因集富集分析正确。arXiv: 1110.4128。2011年,(gydF4y2Bahttp://arxiv.org/abs/1110.4128gydF4y2Ba]gydF4y2Ba
基因表达数据的本体论分析:现有工具、局限性和开放问题。生物信息学,2005,21(18):3587-3595。[gydF4y2Bahttp://bioinformatics.oxfordjournals.org/content/21/18/3587gydF4y2Ba]gydF4y2Ba
Nam D, Kim SY:表达模式分析的基因集方法。生物信息学,2008,9(3):189-197。[gydF4y2Bahttp://bib.oxfordjournals.org/content/9/3/189gydF4y2Ba]gydF4y2Ba
黄德伟,Sherman BT, Lempicki RA:生物信息学丰富工具:通往大型基因表综合功能分析的路径。核酸学报,2009,37:1-13。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pmc/articles/PMC2615629/gydF4y2Ba].[pmid: 19033363 pmcid: pmc2615629]gydF4y2Ba
Jung K, Becker B, Brunner E, Beißbarth T:两组设计中功能基因集全局检测的比较及潜在效应基因的选择。生物信息学,2011,27(10):1377-1383。[gydF4y2Bahttp://bioinformatics.oxfordjournals.org/content/27/10/1377gydF4y2Ba]gydF4y2Ba
Tomfohr J, Lu J, Kepler TB:基于奇异值分解的基因表达路径水平分析。生物信息学,2005,6:225-[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pmc/articles/PMC1261155/gydF4y2Ba].[pmid: 16156896 pmcid: pmc1261155]gydF4y2Ba
Bair E, Tibshirani R:通过基因表达数据预测患者生存期的半监督方法。科学通报,2004,2 (4):[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pmc/articles/PMC387275/gydF4y2Ba].[pmid: 15094809 pmcid: pmc387275]gydF4y2Ba
Armstrong SA, Staunton JE, Silverman LB, Pieters R, Boer MLd, Minden MD, salan SE, Lander ES, Golub TR, Korsmeyer SJ: MLL易位指定了一种独特的基因表达谱,可以区分一种独特的白血病。自然科学,2002,30:41-47。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/11731795gydF4y2Ba]gydF4y2Ba
在微阵列实验中评估差异表达的线性模型和经验贝叶斯方法。中华分子生物学杂志,2004,3:[gydF4y2Bahttp://www.bepress.com/sagmb/vol3/iss1/art3gydF4y2Ba]gydF4y2Ba
Hubert L, Arabie P:比较分区。分类学报,1985,2:193-218。[gydF4y2Bahttp://www.springerlink.com/content/x64124718341j1j0/abstract/gydF4y2Ba]gydF4y2Ba
网络TCGAR:卵巢癌的综合基因组分析。自然学报,2011,474(7353):609-615。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/21720365gydF4y2Ba].(PMID: 21720365)gydF4y2Ba
Soprano KJ, Purev E, Vuocolo S, Soprano DR: Rb2/p130和蛋白磷酸酶2A:全反式维甲酸抑制卵巢癌细胞生长的关键介质。癌基因研究。2006,25(38):5315-5325。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/16936753gydF4y2Ba].(PMID: 16936753)gydF4y2Ba
Um SJ, Lee SY, Kim EJ, Han HS, Koh YM, Hong KJ, Sin HS, Park JS:类视黄酮衍生物在卵巢癌细胞中的抗增殖机制。中华癌症杂志,2001,(2):127-134。[gydF4y2Bahttp://www.sciencedirect.com/science/article/pii/S0304383501006978gydF4y2Ba]gydF4y2Ba
Forbes SA, Bindal N, Bamford S, Cole C, Kok CY, Beare D, Jia M, Shepherd R, Leung K, Menzies A, Teague JW, Campbell PJ, Stratton MR, Futreal PA: COSMIC:在癌症体细胞突变目录中挖掘完整的癌症基因组。核酸学报,2010,39(数据库):D945-D950。[gydF4y2Bahttp://nar.oxfordjournals.org/content/39/suppl_1/D945.longgydF4y2Ba]gydF4y2Ba
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B:利用RNA-Seq绘制和量化哺乳动物转录组。化学学报,2008,5(7):621-628。[gydF4y2Bahttp://dx.doi.org/10.1038/nmeth.1226gydF4y2Ba]gydF4y2Ba
Robinson MD, McCarthy DJ, Smyth GK: edgeR:用于数字基因表达数据的差异表达分析的Bioconductor包。生物信息学,2010,26:139-140。[gydF4y2Bahttp://bioinformatics.oxfordjournals.org/content/26/1/139.shortgydF4y2Ba]gydF4y2Ba
Wu D, Lim E, Vaillant F, Asselin-Labat ML, Visvader JE, Smyth GK: ROAST:用于复杂微阵列实验的旋转基因集测试。生物信息学(牛津,英国)。2010, 26(17): 2176-2182。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/20610611gydF4y2Ba].(PMID: 20610611)gydF4y2Ba
Alexa A, Rahnenführer J, Lengauer T:通过去关联GO图结构改进基因表达数据中功能基团的评分。生物信息学,2006,22(13):1600-1607。[gydF4y2Bahttp://bioinformatics.oxfordjournals.org/content/22/13/1600.abstractgydF4y2Ba]gydF4y2Ba
Young MD, Wakefield MJ, Smyth GK, Oshlack A: RNA-seq的基因本体论分析:考虑选择偏差。中国生物医学工程学报,2010,11 (2):R14-[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/20132535gydF4y2Ba].(PMID: 20132535)gydF4y2Ba
Michaud J、Simpson KM、Escher R、Buchet-Poyau K、Beissbarth T、Carmichael C、Ritchie ME、Schütz F、Cannon P、Liu M、Shen X、Ito Y、Raskind WH、Horwitz MS、Osato M、Turner DR、Speed TP、Kavallaris M、Smyth GK、Scott HS: RUNX1下游通路和靶基因的综合分析。中国生物医学工程学报,2008,29 (3):363-[gydF4y2Bahttp://www.biomedcentral.com/1471-2164/9/363/abstractgydF4y2Ba]gydF4y2Ba
Khatri P, Sirota M, Butte AJ:十年路径分析:当前方法和突出挑战。中国生物医学工程学报,2012,38 (2):e1002375-[gydF4y2Bahttp://dx.doi.org/10.1371/journal.pcbi.1002375gydF4y2Ba]gydF4y2Ba
黄景生,段硕,Bleibel WK, Kistner EO, Zhang W, Clark TA, Chen TX, Schweitzer AC, Blume JE, Cox NJ, Dolan ME:一种全基因组方法识别对乙托苷诱导细胞毒性有贡献的遗传变异。美国国家科学研究院。2007, 104(23): 9758-9763。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/17537913gydF4y2Ba].(PMID: 17537913)gydF4y2Ba
Pickrell JK, Marioni JC, Pai AA, Degner JF, Engelhardt BE, Nkadori E, Veyrieras JB, Stephens M, Gilad Y, Pritchard JK:通过RNA测序了解人类基因表达变化的机制。自然学报,2010,42(6):768-772。[gydF4y2Bahttp://dx.doi.org/10.1038/nature08872gydF4y2Ba]gydF4y2Ba
Carrel L, Willard HF: x失活谱显示女性x连锁基因表达的广泛变异性。自然学报,2005,434(7031):400-404。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/15772666gydF4y2Ba].(PMID: 15772666)gydF4y2Ba
Skaletsky H, Kuroda-Kawaguchi T,风骚女子PJ, Cordum HS,希利尔L,棕色的LG,代表年代,Pyntikova T,阿里•J Bieri T, Chinwalla, Delehaunty, Delehaunty K, Du H,周期G,富尔顿L,富尔顿R,坟墓T,侯科幻,Latrielle P,伦纳德,狂欢节E, Maupin R,矿工T,纳什W,阮C, Ozersky P, Pepin K,摇滚年代,Rohlfing T,舒尔茨B, C,斯科特•K Tin-Wollam,杨SP,沃森RH,威尔逊RK Rozen年代,页面,麦克弗森珍:人类Y染色体的男性特异性区域是一个离散序列类的马赛克。自然学报,2003,38(4):359 - 359。[gydF4y2Bahttp://dx.doi.org/10.1038/nature01722gydF4y2Ba]gydF4y2Ba
王晓燕,王晓燕,王晓燕,等。基于ROCR算法的生物信息学研究。中国生物工程学报,2012,33(6):669 - 672。gydF4y2Ba
Giordano TJ, Kuick R, Else T, Gauger PG, Vinco M, Bauersfeld J, Sanders D, Thomas DG, Doherty G, Hammer G:用转录组谱分析肾上腺皮质肿瘤的分子分类和预测。临床癌症研究:美国癌症学会官方杂志,2009,15(2):668-676。[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/19147773gydF4y2Ba].(PMID: 19147773)gydF4y2Ba
李晨,李晓燕,李晓燕。基于经验贝叶斯方法的微阵列表达数据的批处理调节。生物统计学,2007,8:118-127。gydF4y2Ba
Frazer KA, Ballinger DG, Cox DR, Hinds DA, Stuve LL, Gibbs RA, Belmont JW, Boudreau A, Hardenbol P, Leal SM, Pasternak S, Wheeler DA, Willis TD, Yu F,杨红,曾超,高勇,胡红,胡玮,李超,林玮,刘姝,潘宏,唐欣,王杰,王玮,余杰,张斌,张强,赵宏:一份包含310万snp的第二代人类单倍型图谱。自然学报,2007,42(4):361 - 361。[gydF4y2Bahttp://www.nature.com/nature/journal/v449/n7164/abs/nature06258.htmlgydF4y2Ba]gydF4y2Ba
RDC团队:R:统计计算语言和环境。2010,维也纳:R统计计算基金会,[gydF4y2Bahttp://www.R-project.orggydF4y2Ba].(ISBN 3-900051-07-0)gydF4y2Ba
mr RC, Carey VJ, Bates DM, Bolstad B, Dettling M, Dudoit S, Ellis B, Gautier L, Ge Y, Gentry J, Hornik K, Hothorn T, Huber W, Iacus S, Irizarry R, Leisch F, Li C, Maechler M, Rossini AJ, Sawitzki G, Smith C, Smyth G, Tierney L, Yang JYH, Zhang J: Bioconductor:用于计算生物学和生物信息学的开放式软件开发。中国生物医学工程学报,2004,5 (10):R80-[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/15461798gydF4y2Ba].(PMID: 15461798)gydF4y2Ba
Bolstad BM:高密度寡核苷酸阵列数据的低水平分析:背景、归一化和总结。博士论文。怀卡托大学2004年。[gydF4y2Bahttp://bmbolstad.com/Dissertation/Bolstad_2004_Dissertation.pdfgydF4y2Ba]gydF4y2Ba
Irizarry RA, Bolstad BM, Collin F, Cope LM, Hobbs B, Speed TP: Affymetrix基因芯片探针级数据摘要。核酸学报,2003,31 (4):e15gydF4y2Ba
Benjamini Y, Hochberg Y:控制错误发现率:一种实用而强大的多重测试方法。中国科学(d辑),1995,38(4):369 - 371。gydF4y2Ba
Bourgon R, Gentleman R, Huber W:独立滤波提高了高通量实验的检测能力。中国生物医学工程学报,2010,29(5):576 - 576。[gydF4y2Bahttp://www.pnas.org/content/107/21/9546gydF4y2Ba]gydF4y2Ba
中国生物医学工程学报,2006,7 (10):R93-[gydF4y2Bahttp://www.ncbi.nlm.nih.gov/pubmed/17044931gydF4y2Ba].(PMID: 17044931)gydF4y2Ba
L, Stegle O, Winn J, Durbin R:基因表达数据与推测细胞表型的联合遗传分析。科学通报,2011,7:e1001276-[gydF4y2Bahttp://dx.doi.org/10.1371/journal.pgen.1001276gydF4y2Ba]gydF4y2Ba
Schadt EE, Lamb J, Yang X, Zhu J, Edwards S, Guhathakurta D, Sieberts SK, Monks S, Reitman M, Zhang C, Lum PY, Leonardson A, Thieringer R, Metzger JM, Yang L, Castle J, Zhu H, Kash SF, Drake TA, Sachs A, Lusis AJ:一种推断基因表达与疾病之间因果关系的综合基因组学方法。自然科学学报,2005,37:710-717。gydF4y2Ba
确认gydF4y2Ba
我们感谢以下个人提供的有益意见:Ingo Vogt, Alba Jene, Gunes Gundem, Sonja Althammer和Josh Millstein。S.H.和R.C.感谢ISCIII COMBIOMED赠款[RD07/0067/0001]和西班牙MINECO赠款[TIN2011-22826]的支持。国家癌症研究所综合癌症生物学项目对J.G.项目提供部分支持,资助项目为U54CA149237。gydF4y2Ba
作者信息gydF4y2Ba
作者和隶属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益冲突。gydF4y2Ba
作者的贡献gydF4y2Ba
JG提出并设计了gsa算法。软件由JG和RC实现。SH和JG构思并设计了gsa的应用。SH, RC和JG对数据进行了分析并撰写了论文。所有作者阅读并批准了最终稿件。gydF4y2Ba
电子补充材料gydF4y2Ba
作者提交的图片原始文件gydF4y2Ba
下面是作者提交的原始图片文件的链接。gydF4y2Ba
权利与权限gydF4y2Ba
开放获取gydF4y2Ba本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(gydF4y2Bahttps://creativecommons.org/licenses/by/2.0gydF4y2Ba),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Hänzelmann, S., Castelo, R. & Guinney, J. gsa:微阵列和RNA-Seq数据的基因集变异分析。gydF4y2BaBMC生物信息学gydF4y2Ba14gydF4y2Ba7(2013)。https://doi.org/10.1186/1471-2105-14-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/1471-2105-14-7gydF4y2Ba
关键字gydF4y2Ba
- 不同的表达gydF4y2Ba
- 差异表达基因gydF4y2Ba
- 浓缩的分数gydF4y2Ba
- 调整后的兰特指数gydF4y2Ba
- 线性相加模型gydF4y2Ba