文摘
高通量测序分析,如RNA-Seq ChIP-Seq或条形码计数提供定量的形式读数计数数据。推断出微分信号等数据正确和具有良好的统计能力,估计数据可变性在整个动态范围和一个合适的误差模型是必需的。我们提出一个方法基于负二项分布,由当地回归和方差和均值与目前的一个实现,DESeq,作为一个R / Bioconductor包。
背景
高通量测序的DNA片段中使用一系列的定量分析。这些化验之间的一个共同特征是,他们大量的DNA序列片段反映,例如,一个生物系统的曲目RNA分子(RNA-Seq [1,2])或核苷酸的DNA或RNA相互作用区域绑定分子(ChIP-Seq [3],HITS-CLIP [4])。通常,这些读取被分配到一个类基于他们映射到一个共同的目标基因组区域,每一个类代表一个目标记录,对于RNA-Seq,或有约束力的地区,对于ChIP-Seq。一个重要的汇总统计是读入一个类的数量;对于RNA-Seq,这阅读数已经发现(好的近似)线性相关目标的丰富成绩单(2]。兴趣在于读计数比较不同生物之间的条件。在最简单的情况下,比较是单独完成的,类的类。我们将使用这个词基因也上课,即使一个类也可能指的是,例如,转录因子结合位点,甚至一个条形码(5]。
我们想使用统计测试来决定,对于一个给定的基因,一个读计数观察区别是重要的,也就是说,它是否大于预计将刚才由于自然随机变化。
如果读独立抽样从人口,固定的基因,分数读计数会遵循一个多项分布,可以用泊松分布近似。
因此,泊松分布已被用于测试微分表达式(6,7]。泊松分布的一个参数,这是唯一由它的意思;其方差和所有其他属性遵循;特别是,方差等于的意思。然而,它一直指出[1,8),泊松分布的假设过于严格的:它预测变化小于下图所显示的数据。因此,由此产生的统计测试不控制i型错误(错误发现的概率)的广告。我们显示实例之后,在讨论。
为了解决这个所谓的overdispersion问题,已经提出了模型计算数据与负二项分布(NB) (9,这种方法的使用刨边机方案分析,鼠尾草和RNA-Seq [8,10]。NB分布参数,是由独特的意思μ和方差σ2。然而,在感兴趣的数据集复制的数量通常是太小,估计这两个参数,均值和方差,可靠地为每一个基因。为刨边机,罗宾逊和史密斯认为11均值和方差是相关的σ2=μ+αμ2一个比例常数α在相同的实验,可以估计的数据。因此,只有一个参数需要估计为每一个基因,允许应用程序与少量复制实验。
在本文中,我们扩展这个模型通过允许更一般的,数据驱动的方差和均值的关系,提供一个有效的数据拟合模型的算法,和显示,它提供了更好的适合(部分模型)。结果,更加平衡的选择差异表达基因在整个动态范围的数据可以获得(部分测试微分表达式)。我们将演示方法通过应用这四个数据集(部分应用程序),并讨论如何比较替代方法(部分结论)。
结果与讨论
模型
描述
我们假设读入样本的数量j分配给基因我可以建模的负二项分布(NB),
这有两个参数,意味着什么μij和方差。读取数Kij非负整数。的概率分布给出了补充注意a(补充笔记都在附加的文件1)。通常用于NB分布模型计算数据当overdispersion存在(12]。
在实践中,我们不知道参数μij和,我们需要估计的数据。通常,复制的数量很小,并进一步建模假设需要为了获得有用的估计。在本文中,我们开发一种方法,是基于以下三个假设。
首先,意味着参数μij的预期值,观察到的基因我在示例j的产品,是condition-dependent每个基因值问我,ρ(j)(ρ(j是样品的实验条件j)和一个大小的因素年代j,
问我,ρ(j)真正的期望价值成正比(但未知)从基因片段的浓度我条件下ρ(j)。大小的因素年代j代表了保险,或采样深度、图书馆j,我们将使用术语常见的规模量,如问我,ρ(j)除以,调整的范围年代j。
第二,方差的总和散粒噪声项和一个原始方差项,
第三,我们假设每个基因原始方差参数v我,ρ是一个光滑函数的问我,ρ,
这个假设是必要的,因为复制的数量通常是太低,得到一个精确的估计方差的基因我从数据可用于这种基因。这种假设允许我们从基因池数据类似的表达强度方差估计的目的。
方差的分解方程(3)是出于以下层次模型:我们假设从基因片段的实际浓度我在示例j一个随机变量成正比吗Rij,这样的速度从基因片段我排序是年代jrij。对于每一个基因我和所有的样品j的条件ρ,Riji.i.d.的意思问iρ和方差viρ。因此,计算值Kij,条件Rij=rij,泊松分布率年代jrij。的边缘分布Kij——当允许变化Rij——平均μij和总方差(依法)方差给出方程(3)。此外,如果的时刻就越高Rij根据伽马分布建模,边缘分布的KijNB(见,例如,124.2.2节)。
拟合
我们现在描述模型可以安装数据。数据是一个n×米表的数量,kij,在那里我= 1,…,n索引的基因和j= 1,…,米索引的样品。这个模型有三个组参数:
(我)米大小的因素年代j;从样本数量的预期值j是成正比的年代j。
(2)为每个实验条件ρ,n表达强度参数问iρ;它们反映了预期的片段基因的丰度我条件下ρ,预期值的基因我是成正比的问iρ。
(3)光滑函数vρ:ℝ+→ℝ+;为每一个条件ρ,vρ模型的依赖原始方差viρ在预期的意思问iρ。
规模因素的目的年代j是呈现数量来自不同样本,这可能是不同的深度测序,具有可比性。因此,比率(Kij)/ (Kij”)的预期数量相同的基因我在不同的样品j和j '应该等于比大小吗年代j/年代j '如果基因我不是差异表达或样品吗j和j '是复制。读取的总数,Σ我kij,似乎是一个不错的测序深度的测量,因此一个合理的选择年代j。经验与真实数据,然而,这并非总是如此,因为几个高度和差异表达基因可能读总数有强烈影响,导致总比读计数不是个好估计预期数量的比率。
因此,估计规模因素,取中值的观测数的比率。泛化的过程只是概述了超过两个样本的情况下,我们使用:
分母的这个表达式可以被视为一种pseudo-reference样品在样品通过几何平均获得。因此,每个大小因素的估计计算的值的比率jth pseudo-reference的样本的数量。(注意:虽然这手稿被审查,罗宾逊和Oshlack13提出了一个相似的方法。)
估计问iρ,我们使用样品的数量的平均值j相应的条件ρ,转化为共同的规模:
在哪里米ρ是复制的数量的条件ρ并对这些复制和运行。的函数vρ,我们首先计算样本方差的共同尺度
和定义
在补充报告中B在附加文件中1我们表明,wiρ- - - - - -ziρ是原始的无偏估计量方差参数viρ方程(3)。
然而,对于小数量的复制,米ρ,通常是在应用程序中,价值观wiρ高度可变,wiρ- - - - - -ziρ不会是一个有用的方差估计量的统计推断。相反,我们使用本地回归(14图上的)获得一个光滑函数wρ(问),
作为我们的原始方差估计。
一些需要注意避免在当地回归估计偏差。wiρ是一个随机变量的平方之和,残差吗有欠公平。以下引用(15),第八章和149.1.2],部分,我们使用伽马家族的广义线性模型为当地回归,使用的实现locfit包(16]。
测试微分表达式
假设我们有米一个复制生物条件和样品米B为每个基因样本条件下b我,我们要权衡证据的数据微分表达式之间的基因两个条件。特别是,我们希望测试零假设问iA=问iB,在那里问iA是表达强度参数样本的条件,和qiB对于条件。为此,我们定义,作为检验统计量,总数量在每一个条件,
和他们的总和K是=KiA+KiB。从上一节描述的误差模型,下面我们将展示,在零假设下,我们可以计算的概率事件KiA=一个和KiB=b对于任何两个数字一个和b。我们表示这个概率p(一个,b)。的P一双观察计数的价值金额(kiA,kiB)然后概率的总和等于更少p(kiA,kiB),考虑到整体的总和k是:
的变量一个和b在上面的金额值0,…k我年代。提出的方法到目前为止,罗宾逊和史密斯11),类似于采取其他条件测试,如确切概率法。(见文献[17),第三章讨论的优点调节测试。)
计算p(一个,b)。首先,假设,在零假设下,从不同的样本是独立的计数。然后,p(一个,b)=公关(K我一个=一个)公关(K我B=b)。这样的问题是计算的概率事件K我一个=一个类似地,K我B=b。随机变量K我一个的总和米一个
NB-distributed随机变量。我们近似分布由NB的参数我们获得的Kij。为此,我们首先计算池意味着估计两种情况下的计算,
占的零假设规定问我一个=问我B。条件的总结均值和方差
补充注意C的额外文件1描述了NB的分布参数K我一个可以确定的和。(为了避免偏见,我们直接不匹配的时刻,而是匹配不同的分布统计信息。)的参数K我B类似地。
补充注意D在附加的文件1在方程(解释我们如何评估总结11)。
应用程序
数据集
我们现在结果基于以下数据:
RNA-Seq在飞胚胎
b . Wilczynski中州。刘:Delhomme和大肠弗隆进行了RNA-Seq飞胚胎实验,请提前与我们共享他们的数据。在每个样本数据集,一个基因被改造的过度,我们比较两个生物复制每一两个这样的条件,在下面标记为a和B。
Tag-Seq神经干细胞
Engstromet al。(18]Tag-Seq执行[19)组织培养的神经细胞,其中包括四名从glioblastoma-derived神经干细胞(GNS)和两个解释:神经干细胞(NS)细胞。因为每个组织文化是来自一个不同的学科有不同的基因型,这些数据显示高可变性。
RNA-Seq的酵母
Nagalakshmiet al。(1RNA-Seq执行复制的酿酒酵母文化。他们测试了两个图书馆准备协议,dT和RH为每个协议,获得三个测序运行,每个协议的首次运行,他们有一个进一步的技术复制(相同的文化,图书馆准备复制)和另一个生物复制(不同的文化)。
ChIP-Seq人类基因组单体型图样本
Kasowskiet al。(20.)相比,蛋白质之间的DNA区域占领ChIP-Seq十个人。他们列出了区域聚合酶II和NF-κB和清点,对于每一个样本,读取映射到每个地区的数量。这项研究的目的是调查地区的占领多少个体之间的不同。
方差的估计
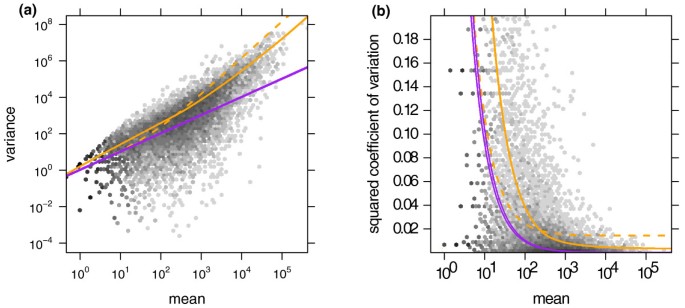
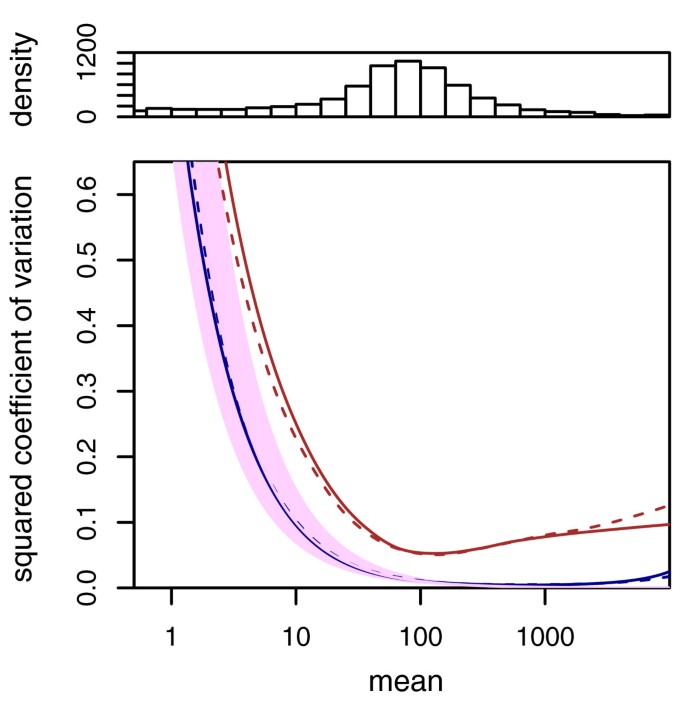
我们开始通过展示方差估计。图1显示了样本方差wiρ(方程(7)策划反对的意思(方程(6))的条件一个在飞RNA-Seq数据。也显示当地回归健康wρ(问)和散粒噪声。在图1 b,我们策划方变异系数(SCV),这是方差的比值均方。在这个情节,橙色和紫色的线之间的距离的SCV噪音是由于生物采样(cf。方程(3))。
许多数据点在图1 b,远高于上橙色曲线可能让当地的健康回归显得可怜。然而,强烈的剩余倾斜分布是可以预料的。看到补充注意E在附加的文件1详情和讨论诊断适用于验证。
测试
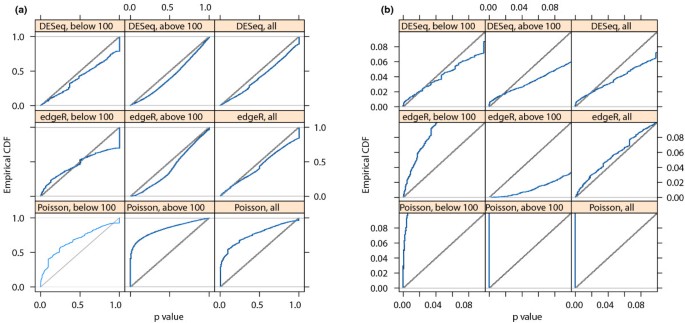
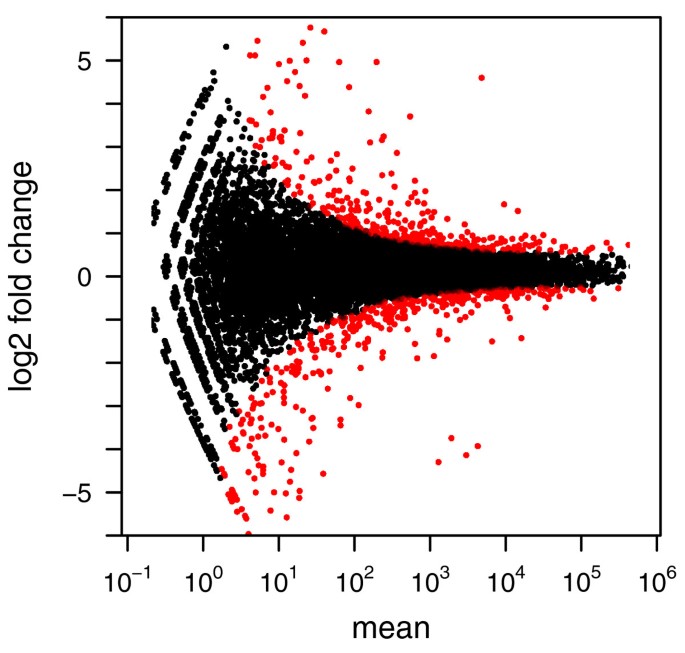
为了验证DESeq保持控制的i型错误,我们对比一个复制的条件一个在动态数据与另一个,使用两个样本方差函数的估计两个复制。图2显示的是经验累积分布函数(ECDFs)P从这种比较获得的值。控制i型错误,的比例P值低于一个阈值α必须≤α,即ECDF曲线(蓝色线)不应该高于对角线(灰线)。图显示,是由i型错误刨边机和DESeq,但不是Poisson-basedχ2测试。后者低估了变化的数据,从而使许多假阳性的拒绝。除了这对真实数据评估,我们也证实DESeq的i型误差控制模拟数据生成上述误差模型;看到补充注意G在附加的文件1。接下来,我们对比了两个一个对两个样本B样本。使用前一节中描述的过程,我们计算P为每个基因值。图3显示了褶皱的变化和获得P值。12%的P值低于5%。调整过程的多个测试Benjamini和业务21)取得了重要的微分表达式错误发现率(罗斯福)10%的864个基因(17605)。这些都是用红色标注的图中。图3演示了如何检测微分表达式的能力取决于总体数量。具体来说,强大的散粒噪声低计数使测试过程调用只有非常高的褶皱变化显著。也可以看出,计数低于约100,即使是很小的数浓度的增加减少了叠化散粒噪声的影响,因此要求,在高计数,当散粒噪声变得不重要(cf图1 b),叠化截止只有弱取决于数水平。这些情节是有用的指导实验设计:弱表达基因,在该地区,散粒噪声很重要,权力可以增加深度测序,而对于higher-count政权,权力只能通过进一步增加生物复制。
i型错误控制。面板显示经验累积分布函数(ECDFs)P值从一个复制的比较情况一个与另一个的飞RNA-Seq数据。没有真正的差异表达基因,和ECDF曲线(蓝色)应保持低于对角线(灰色)。面板(一):上面一行对应DESeq,中间行刨边机和底部行Poisson-basedχ2测试。正确的列显示了所有基因的分布,左边和中间列显示基因分别低于和高于平均为100。面板(b)显示相同的数据,但显示的范围小P值。故事情节表明刨边机和DESeq控制错误(实际上略低于)名义汇率,而Poisson-basedχ2测试失败。刨边机有过多的小P值低计数:上面的蓝线位于对角线。然而,这种过度补偿的方法更为保守的高数。所有方法都显示一个质点p= 1,这是由于数据的离散性,以低计数的效果尤为明显。
测试条件之间的微分表达式一个和B:日志的散点图2比(褶皱变化)和意思。红色标记的基因发现差异表达10%错误发现率当Benjamini-Hochberg多个测试调整。上下情节的符号边界表明基因非常大或无限日志褶皱变化。相应的火山情节补充图所示S8额外的文件2。
与磨边机
我们还分析了数据刨边机(版本1.6.0;(8,10,11])。我们跑刨边机通过四个不同的设置,即在common-dispersion和tagwise-dispersion模式,要么使用估计的大小的因素DESeq或测序读的总数。没有在很大程度上取决于这些选择的结果,在这里,我们报告的结果tag-wise分散模式DESeq估计大小的因素。(所需的R代码复制所有分析数据和数字在本文中提供额外的文件2;此外,该补充提供其他设置的结果刨边机。原始数据可以在附加的文件3)。
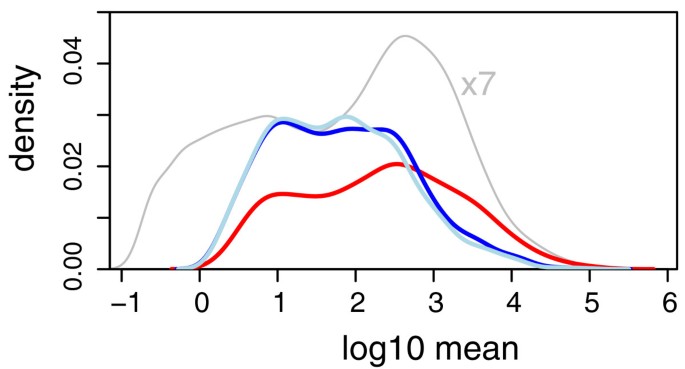
回到图1我们可以看到,刨边机的单一分散的估计方差比低DESeq为弱表达基因和更高的强烈表达基因。因此,正如我们所看到的图2刨边机anti-conservative低表达基因。然而,它弥补了这个被更为保守的强烈表达基因,因此,平均而言,i型误差控制。
然而,在一个测试在不同的条件下,这种行为会导致偏见的列表中发现;目前数据,如图4显示,弱表达基因似乎过多,而很少平均水平高的基因被称为差异表达刨边机。而整体的敏感性两种方法似乎比较(DESeq报告了864支安打,刨边机127支),DESeq产生的结果是更加平衡的动态范围。
与神经干细胞相似的结果数据,数据集有不同的生物学背景和不同的噪声特征(见补充注意F在附加的文件1)。方差估计方案的灵活性提出了工作似乎提供真正优于现有方法在一系列应用程序。
工作没有复制
DESeq允许的分析实验,没有生物复制一个,甚至两个条件。当一个可能不希望从这样的分析得出一个明确的结论,它可能仍然是有用的探索和假说的一代。
如果只提供复制的一个条件,可以选择一个假设variance-mean依赖数据的估计条件持有unreplicated一。
如果条件都没有复制,仍然可以进行分析的基础上,假设对于大多数基因,没有真正的微分表达式,一个有效的均值-方差的关系可以从治疗两个样本估计就像复制。少数不同丰富的基因将作为异常值;然而,他们不会产生严重影响gamma-family GLM适合作为低伽马分布的形状参数值有一个沉重的右手尾。一些过高的估计方差可能预期,这将使这种方法保守。
我们进行了这样的分析飞RNA-Seq和神经细胞Tag-Seq数据,通过限制这两个数据集只有两个样本,分别来自条件。神经细胞的数据,估计方差函数,正如预期的那样,有些上面两个函数的估计GNS和NS复制。
用它来测试微分表达式仍然发现269支安打在罗斯福= 10%,其中202是612的点击率与所有可用的更可靠的分析样本。飞RNA-Seq数据,然而,只有90 862的点击量(11%)恢复(两个新的打击)。解释这些观察神经细胞中的事实数据,复制之间的可变性之间并不是远小于条件,使得前者后者一个可用的代理。另一方面,对于动态数据,复制之间的差异远远小于之间的条件,表明复制提供了重要的信息,否则无法获得实验数据的变化(参见下一节)。
Variance-stabilizing转换
给定一个variance-mean依赖,variance-stabilizing转换(威仕特)是一个单调映射为转换后的值,方差(大约)独立的意思。使用variance-mean依赖w(问)估计DESeq威仕特是由
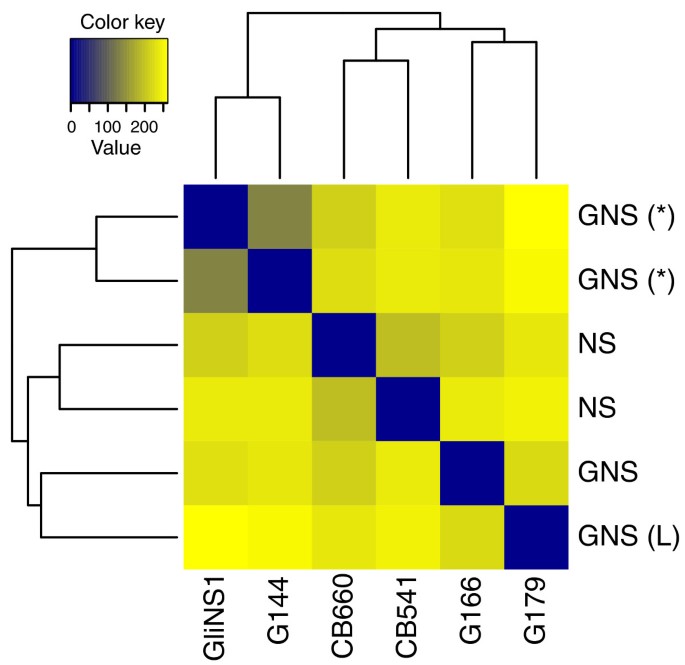
应用转换τ相同等级的统计数据,kij/年代j,收益率值的方差在整个动态范围大约是相同的。威仕特的一个应用程序是示例集群,如图5;这种方法比,更直接的说,定义一个合适的距离度量untransformed计数数据,不明显的选择,可能不容易结合聚类或分类算法(这往往是专为有相似的变量分配属性)。
示例集群的神经细胞数据Kasowski et al。(18]。常见的方差函数估计为所有样本和使用应用variance-stabilizing转换。热图显示一个错误的颜色表示的欧氏距离矩阵(从零距离深蓝色到橙色大距离),和系统树图代表一个层次聚类。两个GNS样本来自相同的病人(标有“(*)”)并显示最高程度的相似性。另外两个GNS样品(包括一个非典型的大细胞,标记(L))一样不同的前两个NS样本。
ChIP-Seq
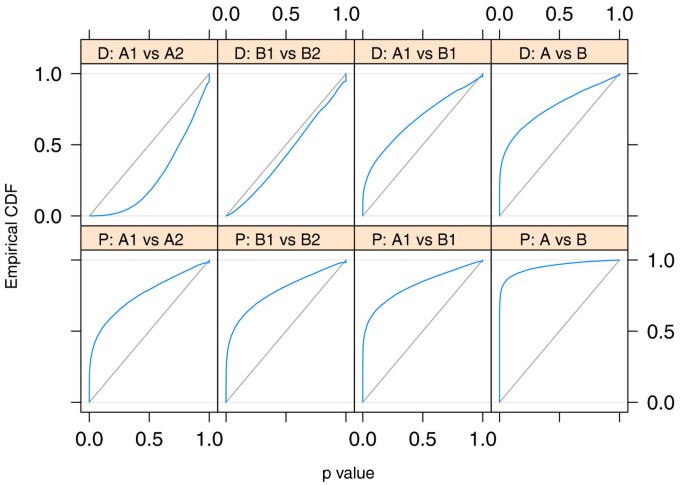
DESeq也可以用来分析比较ChIP-Seq化验。Kasowskiet al。(20.]分析了人类基因组单体型图个人和转录因子结合计算每个样本多少读取区域映射到预先确定的绑定。从他们的数据集,我们考虑两个个体人类基因组单体型图IDs GM12878 GM12891,至少在这两个四个复制已经完成,测试和微分占领的地区。图的左上角两个面板6显示比较在同一个体,表明i型控制是错误DESeq。没有显著的地区使用Benjamini-Hochberg罗斯福调整10%。微分职业被发现,然而,当对比的两个人,有4460 19028个地区重要的只有两个复制使用和8442年四个复制使用(右上角两个面板)。
应用ChIP-Seq数据。显示ECDF曲线P聚合酶ii的价值观造成比较ChIP-Seq之间的数据复制相同的个体(第一和第二列)和两个不同的个体之间(第三和第四列)。上面的一行对应一个分析DESeq(' D '),较低的行到一个基于泊松的漠视(“P”)。如果不存在真正的微分占领(即当比较复制)的ECDF(蓝色)应保持低于对角线(灰色),这对应于制服P值。在第一列,两个复制从人类基因组单体型图个人GM12878 (A1)比较两个进一步的复制相同的个人(A2)。同样,在第二列,两个从个人GM12891复制(B1)比较两个进一步的复制相同的个人(B2)。为DESeq没有多余的低P如预期值被认为,当比较复制。相比之下,泊松GLM分析产生强烈的小充实P值;overdispersion的这是一个反射数据,即数据的方差大于漠视泊松假设(参见部分选择分布)。从个人GM12878第三列比较两个复制(A1从其他个人()对两个B1)。真正的职业差异预计,这两种方法都导致浓缩的小P值。第四列显示了四个GM12878复制的比较(A1结合A2)对四个复制GM12891 (B1,B2);增加样本容量导致更高的检测能力,因此小P值。
使用另一种方法,Kasowski等。拟合广义线性模型(glm)泊松家族的。这(低排图6)导致一个浓缩的小P值甚至比较在同一个体,表明方差是低估了泊松的漠视,和文字的使用P值将导致anti-conservative(过于乐观)的偏见。Kasowski等。解决这对偏见和调整通过使用额外的调用标准微分占领。
结论
为什么要开发新的统计方法序列统计数据?如果有大量复制,数据分布问题可以避免用非参数方法,如rank-based或排列测试。然而,它是可取的(可能)考虑与小数量的复制实验条件。为了比较观察不同预期的随机变异,我们可以改善我们的照片,后者在两个方面:首先,我们可以使用分布的家庭,如正常,泊松和负二项分布,为了确定更高的时刻,因此尾部行为,统计的微分表达式,根据观察到的低阶均值和方差等的时刻。第二,我们可以分享信息,例如,分布参数,基于概念之间的基因,这些数据从不同的基因变化遵循类似的模式。在这里,我们描述了这种方法的一个实例,我们将讨论我们的选择。
选择分布
在大数量时,正态分布可能会提供一个好的近似between-replicate可变性,这不是为降低计算值的情况下,其离散性和偏态意味着概率估计从正常的近似计算是不够的。
对泊松近似,一个关键的纸是由Marioni工作et al。(6研究了]技术RNA-Seq的再现性。他们从两个组织样本中提取总RNA,从肝脏和肾脏的同一个人。RNA样本他们把7整除,准备一个图书馆从每个整除根据协议推荐的Illumina公司和取样的每个库一巷Solexa基因组分析仪。对于每一个基因,然后计算出七项的方差相同的组织样本,发现非常好的协议与泊松模型预测的方差。符合我们的参数部分模型,泊松噪声变化的最小数量预计在计票过程。因此,Marioni等。得出结论,RNA-Seq技术重现性很好,和之间的变异技术复制接近散粒噪声极限。从这个角度来看,Marioni等。(同样的布拉德et al。(22)建议使用泊松模型(确切概率法,或作为一个近似的似然比检验)来测试是否两个样品之间的差异表达基因。重要的是要注意,拒绝从这样一个测试只告诉我们,平均计算两个样本之间的差异大于一个期望之间技术复制。因此,我们不知道这种差异是由于不同组织类型、肾脏肝脏,或是否相同规模的差异也可能是发现如果一个人比较两样本相同的不同部分肝脏,或从肝脏的两个人。
图1表明,散粒噪声只占主导地位的计算值很低,虽然已经温和,样品的生物变异的影响超过了散粒噪声的数量级。
这是确认的比较与生物复制技术1]。在图7我们使用DESeq获取数据的方差估计Nagalakshmiet al。(1]。分析表明,不同技术复制几乎超过了散粒噪声水平,而生物复制更多的不同。测试基于泊松模型的微分表达式,如中讨论的引用(6,7,20.,22,23)应被谨慎,因为他们可能严重低估了生物多样性的影响,尤其是对高表达基因。
因此,最好使用一个模型,允许overdispersion。为泊松分布时,方差和均值相等,负二项分布是一个泛化,允许差异更大。发表的最先进的方法使用这种分布是可能的刨边机(8]。DESeq欠其基本思想刨边机,但在几个方面有所不同。
基因之间的信息共享
首先,我们发现使用总读算作测序深度的估计,因此观察到的调整数之间的样品(如推荐的罗宾逊et al。(8]等)可能导致复制高明显差异,因此在微薄的力量来检测真正的差异。
DESeq使用更健壮的大小估计方程(5);事实上,刨边机的功率增加时提供这些大小估计。(注:本文在审查时,刨边机修改为使用Oshlack和罗宾逊的方法13]。)
对于小数量的复制经常遇到在实践中,它是不可能同时获得可靠的估计的方差和均值参数NB分布。刨边机通过评估一个解决这个问题常见的分散参数。在我们的方法中,我们使用的可能性估计更灵活,mean-dependent当地的回归。典型实验中可用的数据量足够大以允许足够精确的局部估计的色散。在大动态范围是RNA-Seq典型,原始SCV经常出现明显改变,考虑到这个DESeq为了避免偏向某些领域动态范围的差异表达调用(参见图2和4)。
这种灵活性是最实质性的区别DESeq和刨边机模拟显示,刨边机和DESeq执行相对如果提供人工数据与常数SCV(补充注G在附加文件中1)。刨边机试图弥补单个参数噪声模型的刚度通过允许的调整每个基因实证的基于模型的方差估计方差。一个经验贝叶斯过程,类似于一个最初的开发limma包(24- - - - - -26),决定如何把这两个来源的信息优化。然而,对于典型的低复制数字,这个所谓的tagwise分散模式似乎有什么影响(图4),甚至减少刨边机在额外的权力(补充注F文件1)。
第三,我们提出一个简单的和健壮的方式估计原始数据的方差。罗宾逊和史密斯11)使用一种技术称为quantile-adjusted条件最大似然找到原始SCV的无偏估计。的分位数的调整指rank-based程序修改的数据,这样的数据似乎源于样本库大小相等。在DESeq,不同的库大小只是解决线性扩展(方程(2)和(3)),这表明分位数的调整是一个不必要的并发症。我们付出的代价是,我们需要让NB的总和变量的近似方程(10)本身就是NB分布。虽然似乎调整分位数和关注我们的近似构成原因在实践中,DESeq的方法是计算速度更快,也许,在概念上更简单。
第四,我们的方法提供了有用的诊断。补充图S3的额外文件等情节2对法官的可靠性测试。在图1 b和7,很容易看到的平均值对散粒噪声生物差异占主导地位;这些信息是有价值的,以决定是否测序深度或生物复制的数量检测能力的限制因素,因此有助于规划实验。一个热图如图5数据质量控制是很有用的。
材料和方法
R包DESeq
我们实现了环境统计的方法作为一个包R (27)和分发在Bioconductor项目(28]。作为输入,将一个表的统计数据。数据,以及元数据,如样本和基因注释,S4类进行管理CountDataSet,这是来自eSet,Bioconductor的标准数据类型类似于表的数据。包提供高级功能执行分析等部分所示应用程序只有几个命令,允许研究人员与R的小知识。示例演示了这一点在提供的文档包(包小插图)。此外,低级别功能提供了先进的用户希望偏离标准的工作流程。一个典型的计算,如分析部分所示应用程序需要几分钟的时间,个人电脑。
本文提供的分析被执行DESeq。读者希望详细检查他们会发现Sweave文档的评论R代码分析代码作为额外的文件2和附加文件中的原始数据3。
缩写
- ChIP-Seq:
-
(高通量)测序染色质免疫沉淀反应
- ECDF:
-
经验累积分布函数
- 罗斯福:
-
错误发现率
- 全球语言监测机构:
-
广义线性模型
- RNA-Seq:
-
高通量测序的RNA
- SCV:
-
²变异系数
- 注:
-
负二项(分布)
- 威仕特:
-
variance-stabilizing转换。
引用
王Nagalakshmi U, Z, Waern K,守C Raha D,格斯坦M,斯奈德M:定义的酵母基因组RNA的转录景观序列。科学。2008年,320:1344 - 1349。10.1126 / science.1158441。
威廉姆斯Mortazavi,英航,麦丘K, Schaeffer L,荒原B:映射和量化RNA-Seq哺乳动物的转录组。Nat方法。2008年,5:621 - 628。10.1038 / nmeth.1226。
罗伯逊G,赫斯特米,班布里奇M, Bilenky M,赵Y,曾庆红T, Euskirchen G,伯尼尔B, Varhol R,德莱尼,Thiessen N,格里菲斯OL,他一个,马拉M,斯奈德M,琼斯S:全基因组的DNA STAT1协会使用染色质免疫沉淀反应和大规模并行测序。Nat方法。2007年,4:651 - 657。10.1038 / nmeth1068。
Fak JJ, Licatalosi DD, Mele Ule J, Kayikci M,西南,克拉克助教,史怀哲AC,抱我,王X,达内尔JC,达内尔RB: HITS-CLIP收益率大脑可变RNA加工全基因组的见解。大自然。2008年,456:464 - 469。10.1038 / nature07488。
史密斯是Heisler LE Mellor J,燕麦饼干F,汤普森MJ, Chee M,罗斯FP,杰伯G,通过深条码测序Nislow C:定量表现型。基因组研究》2009年19:1836 - 1842。10.1101 / gr.093955.109。
Marioni JC,梅森CE、鬃毛SM、斯蒂芬斯M,吉拉德·Y: RNA-seq:评估技术与基因表达阵列再现性和比较。基因组研究》2008年,18:1509 - 1517。10.1101 / gr.079558.108。
王王王L,冯Z, X, X,张X: DEGseq: R包从RNA-seq数据识别差异表达基因。生物信息学。2010年,26日:136 - 138。10.1093 /生物信息学/ btp612。
罗宾逊博士Smyth GK:主持统计测试来评估不同的标签。生物信息学,2007,23 (21):2881 - 2887。10.1093 /生物信息学/ btm453。
惠特克L:泊松小数定律。生物统计学。1914年,10:36 - 71。10.1093 / biomet / 10.1.36。
罗宾逊博士麦卡锡DJ,史密斯GK:磨边机:Bioconductor包微分表达式数字基因表达数据的分析。生物信息学。2010年,26日:139 - 140。10.1093 /生物信息学/ btp616。
罗宾逊博士Smyth GK:负二项分布的小样本估计,SAGE数据的应用程序。生物统计学。2008年,9:321 - 332。10.1093 /生物统计学/ kxm030。
卡梅伦AC, Trivedi PK:回归分析统计数据。1998年,剑桥大学出版社
罗宾逊博士Oshlack答:一个比例微分表达式RNA-seq分析数据的归一化法。基因组医学杂志2010年11:r25 - 10.1186 / gb - 2010 - 11 - 3 - r25。
装载机C:当地的回归和可能性。1999年,施普林格
McCullagh P, Nelder JA:广义线性模型。1989年,查普曼&大厅/ CRC, 2
locfit:当地的回归、可能性和密度估计。(https://doi.org/cran.r-project.org/web/packages/locfit/]
Agresti答:分类数据分析。2002年,威利,2
P, Tommei D,前锋,史密斯,波拉德,伯顿P:转录特征的胶质母细胞瘤干细胞系使用标记排序。2010年
莫林Morrissy, RD,德莱尼,曾T,麦当劳H,琼斯,赵Y,赫斯特米,马拉马:下一代标记为癌症基因表达图谱测序。基因组研究》2009年19:1825 - 1835。10.1101 / gr.094482.109。
Kasowski M, Grubert F, Heffelfinger C, Hariharan M, Asabere, Waszak SM, Habegger L, Rozowsky J,施米,城市AE,香港,Karczewski KJ, Huber W, Weissman SM,格斯坦MB,戈倍尔乔,斯奈德M:转录因子的变化在人类中有约束力。科学。2010年,328:232 - 235。10.1126 / science.1183621。
业务,Benjamini Y Y:控制错误发现率:一种实用和强大的多个测试方法。57 J罗伊Stat Soc b . 1995: 289 - 300。
布拉德J, Purdom E,汉森K, Dudoit S:评价统计方法标准化和微分表达式mRNA-Seq实验。BMC生物信息学。2010年,11:94 - 10.1186/1471 - 2105 - 11 - 94。
辛格布鲁姆JS,汗Z, Kruglyak L, M, Caudy AA:测量短阅读顺序:基因差异表达定量比较2声道基因表达微阵列。BMC基因组学。2009年,10:221 - 10.1186/1471 - 2164 - 10 - 221。
史密斯GK: Limma:微阵列数据的线性模型。使用R和Bioconductor生物信息学和计算生物学的解决方案。编辑:绅士R,凯里V, Dudoit S, R Irizarry WH。2005年,纽约:施普林格,397 - 420。full_text。
史密斯GK:线性模型和经验贝叶斯方法评估微分表达式在微阵列实验。统计:麝猫杂志。2004年,3:第三条-
Lonnstedt我,速度T:复制微阵列数据。Stat罪。2002年,12:脉络。
接待员:统计计算的语言和环境。(https://doi.org/www.R-project.org]
绅士RC,凯里VJ,贝茨DM Bolstad B, Dettling M, Dudoit年代,埃利斯B, Gautier L,通用电气Y,贵族J, Hornik K, Hothorn T, Huber W, Iacus年代,伊R, Leisch F,李C, Maechler M,罗西尼AJ, Sawitzki G、C史密斯,史密斯G, Tierney L,杨JYH,张J: Bioconductor:打开软件开发计算生物学和生物信息学。基因组医学杂志。2004年,5:r80 - 10.1186 / gb - 2004 - 5 - 10 - r80。
幸福CI,费舍尔RA:拟合生物数据的负二项分布。生物识别技术。1953年,9:176 - 200。10.2307 / 3001850。
克拉克SJ,佩里约:负二项参数估计最大quasi-likelihoodκ。生物识别技术。1989年,45:309 - 316。10.2307 / 2532055。
无法无天的摩根富林明:负二项和混合泊松回归。J Stat。1987年,15:209 - 225。10.2307 / 3314912。
萨哈K,保罗年代:Bias-corrected负二项分布参数的极大似然估计量。生物识别技术。2005年,61:179 - 285。10.1111 / j.0006 - 341 x.2005.030833.x。
快速、准确的计算二项概率。(注意:这是一个原始论文的副本,这不再是网上。),(https://doi.org/projects.scipy.org/scipy/raw-attachment/ticket/620/loader2000Fast.pdf]
Langmead B, C杰尔,流行,扎尔茨贝格SL:超快和节约内存对齐的人类基因组DNA序列。基因组医学杂志。2009年,10:r25 - 10.1186 / gb - 2009 - 10 - 3 - r25。
与Python HTSeq:高通量测序分析数据。(https://doi.org/www-huber.embl.de/users/anders/HTSeq/]
DESeq。(https://doi.org/www-huber.embl.de/users/anders/DESeq]
确认
我们感谢保罗·伯顿分享神经干细胞数据公布之前,和Bartek Wilczyński,尼古拉斯·Delhomme和艾琳Ya-Hsin Liu弗隆同样分享RNA-Seq飞行数据。我们感谢尼古拉斯·Delhomme和朱利安Gagneur有用的评论的手稿。美国一个。部分由欧盟资助的研究和培训网络“染色质可塑性”。
作者信息
作者和联系
相应的作者
额外的信息
作者的贡献
SA和WH开发方法和写的手稿。SA实现方法并进行分析。
电子辅料
13059 _2010_9558_moesm2_esm.pdf
额外的文件2:补充二世。PDF文件提供所有的源代码分析本文提出,与评论,Sweave文档。21 MB (PDF)
作者提交的原始图像文件
权利和权限
本文是分布式根据创作共用署名4.0国际许可(http://creativecommons.org/licenses/by/4.0/),它允许无限制的使用,分布和繁殖在任何媒介,你提供给适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文的数据可用,除非另有说明。
关于这篇文章
引用这篇文章
安德斯·S。,Huber, W. Differential expression analysis for sequence count data.基因组医学杂志11R106 (2010)。https://doi.org/10.1186/gb - 2010 - 11 - 10 - r106
收到了:
修改后的:
接受:
发表:
DOI:https://doi.org/10.1186/gb - 2010 - 11 - 10 - r106
关键字
- 散粒噪声
- 当地的回归
- 负二项分布
- 负二项
- 大小的因素

评论
查看存档的评论(2)