文摘
本研究描述了宏基因组生物标志物的发现和验证新方法的类比较,测试生物一致性和效果评估。这解决的挑战,寻找生物,基因,或通路一致解释两个或两个以上的微生物群落之间的差异,这是一个宏基因组研究的核心问题。一些微生物和广泛验证我们的方法提供了一个方便的网络接口的方法http://huttenhower.sph.harvard.edu/lefse/。
背景
生物标志物发现已经被证明是一种最广泛适用的和成功的方法将分子和基因数据转化为临床实践。比较健康和病变组织的重要性凸显了任务,比如类发现小说(检测疾病的亚型)和类预测(确定一个新的样本)的亚型1- - - - - -4),最近的宏基因组分析表明,人类微生物群可以用作生物标记等宿主因素的生活方式(5- - - - - -7)和疾病(7- - - - - -10]。随着测序技术的不断发展,使微生物生物标志物越来越容易检查出来的,这使得临床诊断和微生物应用程序通过微生物群落的比较11,12]。
人类微生物组,由总微生物与人类宿主相关的补充,是一个重要的新兴领域宏基因组生物标志物的发现(13,14]。肠道微生物丰度的变化,口腔和皮肤相关疾病从肥胖(15- - - - - -17牛皮癣][18]。更普遍的是,微生物群落的宏基因组研究是一种有效的方法来识别任何无教养的样本的微生物或微生物代谢特征(19,20.]。宏基因组数据的分析通常寻求识别特定的生物,演化支,操作分类单位,或路径的两个或两个以上的组相对丰度差异的样本,而且微生物群落的几个特点已经被提议作为潜在生物标志物用于各种疾病。例如,单一致病菌可以信号疾病如果在一个社区21,22),增加和减少社会的复杂性,使被观察到在细菌性阴道炎23和克罗恩氏病8]。这些不同类型的微生物生物标记物是与疾病表型,但很少提供生物信息学方法存在解释类比较宏基因组数据。
识别最生物信息特征区分两个或两个以上的表型可以挑战在任何基因组数据集,并为宏基因组生物标志物尤其如此。需要健壮的统计工具来确保结论来自宏基因组数据的重现性,这是生物研究的临床应用的关键。相关的挑战与高维数据的数据类型或实验平台;潜在的生物标记物的数量,例如,通常是远高于样品的数量(24- - - - - -26]。宏基因组分析另外提出自己的具体问题,包括测序错误,嵌合读(27,28,复杂的生物学基础;发现了许多微生物社区非常高inter-subject可变性。例如,大的差异是发现即使在双胞胎的肠道微生物组29日),人类微生物组和环境社区被认为是具有长尾的稀有生物的存在(30.- - - - - -32]。此外,简单地识别潜在生物标志物没有阐明生物一致性和角色只是一个前兆理解microbe-microbe或宿主相互作用的潜在机制(33]。在许多情况下,有必要解释不仅有两个生物样品不同,但为什么。这个问题被称为类比较:表型之间的差异如何如肿瘤亚型或疾病状态的一致解释生物学途径或分子机制?
类已经提出了大量的方法发现或在宏基因组数据比较。梅金(34)是一个宏基因组分析工具中最近添加的系统发育的比较(35和统计分析36]。然而梅根,只能单对基因组进行比较,与邮票也是如此(37),并引入一个概念“生物相关性”的形式的置信区间。UniFrac [38]比较套基因组在严格分类级别使用系统的距离,虽然MG-RAST [39],ShotgunFunctionalizeR [40],mothur [41],METAREP [42所有过程宏基因组数据(主要是使用标准的统计测试t与一些修改测试)。大多数社区的方法从生态的角度分析依赖于无监督聚类分析基于主成分分析(43)或主坐标分析(44]。这些可以成功地检测组织相关的样品,但是他们不包括先验知识的表型或环境条件相关的团体,他们通常不负责集团关系识别的生物特性。Metastats [45)是目前唯一的方法,明确夫妻统计分析(评估基因组不同)和生物标志物的发现(检测特性描述的差异)基于重复t统计和费雪的测试随机排列。然而,这些方法,即使是那些提供细致入微的分析宏基因组数据,提供生物类的解释建立统计学意义,生物一致性和效应值估计预测生物标志物。
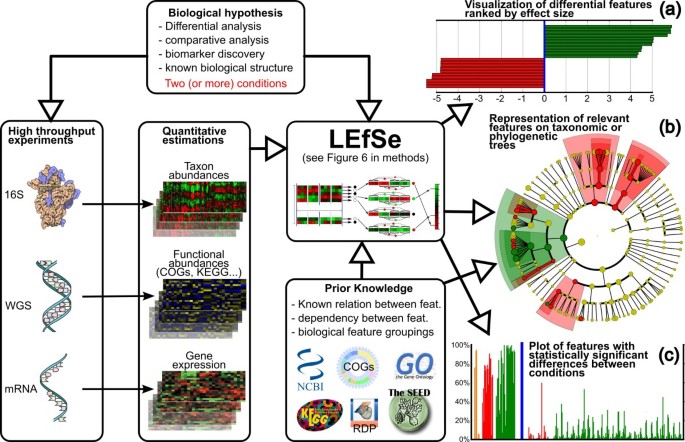
在这项工作中,我们目前的线性判别分析(LDA)效果(LEfSe)方法来支持高维类与特定关注宏基因组比较分析。LEfSe决定了功能(生物演化支、操作分类单位,基因,或功能)最有可能解释差异统计学意义的标准测试类之间的耦合与额外测试编码生物一致性和效果的相关性。类比较方法通常预测生物标志物组成特征违反没有差异的零假设类;我们另外检测特性的子集与丰富模式兼容算法编码生物假设和估计的大小显著变化。特别是,效果提供了一个观察现象的大小估计由于每个描述特性,因此它是一个有价值的工具,用于排序的相关性不同的生物方面和解决进一步的调查和分析。前生物知识的引入方法约束分析,从而有助于解决挑战传统与高维数据挖掘。LEfSe因此旨在支持生物学家提出的生物标志物,解释大部分的利益分化表型的影响(两个或两个以上)的生物标志物发现比较和假说驱动的调查。发现生物标志物在分类树的可视化提供了一个有效手段,总结结果在生物学上有意义的方式,这两种统计和视觉捕捉16 s分类法/固有的层次关系的发展史或本体的通路和生物分子功能。
我们从人类微生物组验证这种方法使用数据,溃疡性结肠炎小鼠模型,和环境样品,在每种情况下预测的生物或操作分类单位,简明地分化相比较的类。我们进一步评估LEfSe使用合成数据,观察它达到一个更好误判率与标准统计测试相比,在价格的适度增加假阴性率(也可以由用户根据需要调整)。LEfSe包括一个方便的图形界面的实现合并的星系框架(46,47)是在网上提供的(48]。
结果与讨论
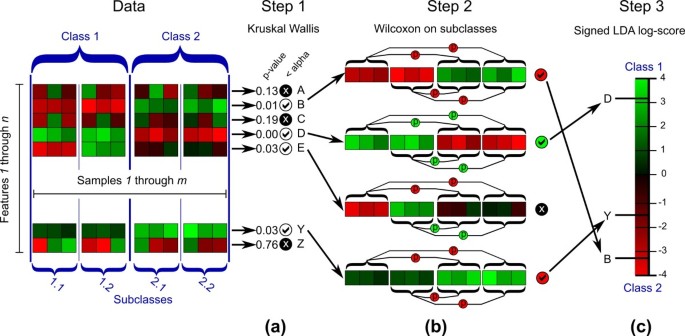
LEfSe是一种算法对于高维识别基因组特性的生物标志物的发现和解释(基因,通路,或类群)描述两个或两个以上的生物条件之间的差异(或类)(图1)。统计意义,它强调生物一致性和相关性影响,让研究人员确定不同的丰富特性也符合生物学意义的类别(子类;见材料和方法)。LEfSe第一强劲统计识别特性不同的生物类之间。然后执行额外的测试来评估这些差异是否符合预期对生物行为;例如,给定一组内的一些已知的人口结构输入样本,是一个功能更丰富的人口在所有子类或只有一个吗?具体来说,我们首先使用非参数的阶乘克鲁斯卡尔-沃利斯(千瓦)sum-rank测试(49)检测与重要的微分特性丰富的阶级的利益;生物一致性是随后调查之间的两两测试使用一组子类使用(未配对)Wilcoxon rank-sum测试(50,51]。作为最后一步,LEfSe用LDA (52)来估计每个不同的效果和丰富的功能,如果需要由调查员进行降维。
LEfSe矿山广泛的高通量基因数据找到相关生物学特性描述一个或多个实验条件。输入到系统的规范生物假说进行调查(条件和inter-condition样本分组),试验得到的高维数据,从文学或数据库,可选地,先验知识用于定义已知特性之间的关系(用于有意义的层次组织的发现生物标志物)或样品(用于测试生物潜在生物标志物)的一致性。LEfSe是一个三步算法(详细情况见图6)。(一)LEfSe第一次提供的列表功能之间的微分条件感兴趣的统计和生物意义,排名根据效果。(b)为已知的层次结构问题,系统或功能,然后提供一个映射分类或功能树的区别。(c)最后,系统生成一个柱状图可视化中的原始数据指定为每个相关特性问题结构。虽然LEfSe开发主要是为宏基因组数据包含分类单元或基因丰度,可以用于生物标志物发现之前在任何环境中,生物知识有关的结构比较,再加上在统计上有显著差异的高维基因特性。KEGG京都基因和基因组的百科全书;WGS,全基因组鸟枪。
我们有专门设计的生物标志物发现LEfSe宏基因组数据。我们因此总结我们的研究结果应用工具从16 s rRNA基因和全基因组鸟枪数据集来检测细菌生物和功能特征之间的差异丰富的两个或两个以上的微生物环境。这些包括身体网站在人类微生物组(粘膜表面和需氧/厌氧环境),成人和婴儿肠道菌群,炎症性肠病状态在老鼠模型中,细菌和病毒环境的社区,和合成数据定量计算评价。
在人类微生物类群描述身体的站点
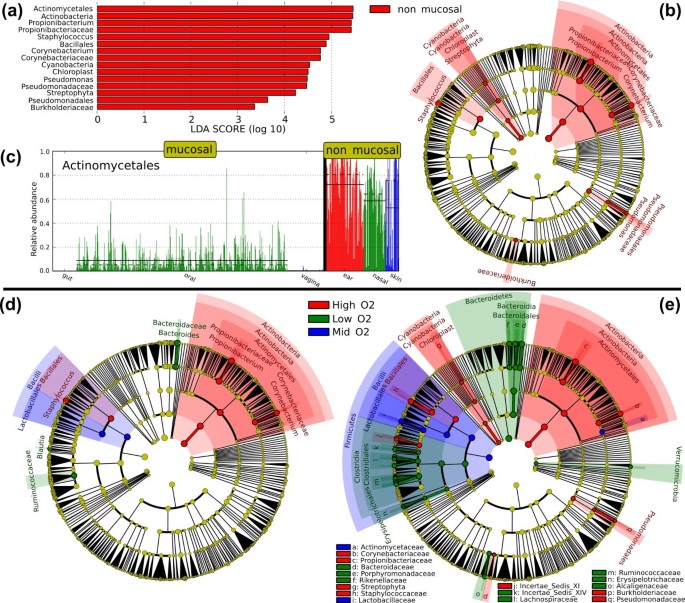
人体微生物群落组织在多个网站是当前活跃的研究领域,因为低收入和高通量方法显示差异和重叠的微生物群中多个身体站点(53,54]。我们检查了这些差异的16 s phylometagenomic数据集从24个人参加人类微生物组计划(13,55]。最少5000 16 s rRNA基因序列获得301年样本24名健康受试者(12男,12女性)覆盖18身体网站,包括6个主体网站类别:口腔(9子网站采样),阴道(3子网站采样),皮肤(2子网站采样),retroauricular折痕(2子网站采样),鼻腔样本(1)和肠道的示例(1)。我们通过对比验证LEfSe粘膜与non-mucosal身体网站类和通过比较三个层次的有氧环境(厌氧、microaerobic和有氧)。在这两种情况下,身体的每个类中不同站点被用作生物子类。
粘膜表面是由不同细菌殖民;non-mucosal微生物强烈丰富的放线菌
我们第一次分析集中在微生物群组成差异粘膜和non-mucosal身体网站。口腔、肠道和阴道网站分为粘膜社区和前窝的来源(皮肤),鼻腔,retroauricular non-mucosal折痕。粘膜环境从身体的其他网站有很大的不同,主要是由与人类免疫系统的交互特征,氧化挑战,水合作用[56]。
LEfSe提供了三个主要输出(图2),描述了影响大小的粘膜/ non-mucosal社区中观察到的差异,这些差异的系统发育分布基于核糖体数据库项目(RDP)细菌分类57),和原始数据驱动这些影响。LEfSe发现15细菌演化支显示统计学意义和生物体内non-mucosal网站(图一致的差异2)。
人类微生物组LEfSe结果。(两者)粘膜身体网站分析。粘膜微生物群落是多样化的,而身体non-mucosal网站的特点是几个演化支,包括放线菌。这里的分析报告进行初始数据从人类微生物组计划55,56)分配主体网站粘膜和non-mucosal类,并使用身体网站子类。这些图形输出生成的公开LEfSe可视化模块应用于微生物分类分析结果和整合先验知识(58]。LDA分数计算的(a)直方图特性不同粘膜和non-mucosal身体之间丰富的网站。LEfSe分数可以解释为一致的程度之间的相对丰度差异特性分析微生物群落的两类。直方图从而确定演化支在所有这些发现统计学和生物微分解释社区之间最大的差异。(b)分类的统计和生物粘膜和non-mucosal身体网站之间的差异一致。不同的颜色也代表着最丰富的类(红色指示non-mucosal、黄色非重要)。每个圆的直径的分类单元的数量成正比。这种表示方法,这里采用核糖体数据库项目(RDP)分类58),同时强调高层趋势和具体属——例如,多个不同的丰富的兄弟姐妹类群与父进化枝的变化一致。(c)直方图的放线菌目相对丰度(0 (1]区间)在粘膜和non-mucosal身体网站。子类(身体特定部位)不同颜色和均值和中位数相对丰富的放线菌目与实线和虚线表示,分别。(d, e)好氧生活分析。类群的进化分枝图报告(由小圆和阴影突出显示)显示不同的丰度值(根据LEfSe)的三个O2端依赖类描述的结果;对于每一个分类单元,颜色表示的类高值两个小圆圈和阴影。(d)的严格(所有类微分)版本LEfSe 13生物标志物检测而(e)的非严格的(至少有一个类微分)版本LEfSe检测60微生物生物丰度微分在有氧,厌氧或microaerobic条件。额外的文件2报告的非严格的版本LEfSe集中在厚壁菌门门,突出几个low-O2具体属Ruminococcaceae和Lachnospiraceae内。
non-mucosal体内最丰富的细菌类群不同网站属于门与流行有氧成员:放线菌,厚壁菌门和变形菌门,包括环境生物从Betaproteobacteria Gammaproteobacteria演化支。Non-mucosal过多属包括丙酸菌属,葡萄球菌(发现只在non-mucosal样品),棒状杆菌属,假单胞菌。还值得注意的是叶绿体的相关表征植物生物(叶绿体),相关类群的分布不同,像一些仅限于non-mucosal表面(环境暴露和潜在的化妆品)和其他消化(消化食品)。身体没有演化支始终存在于所有粘膜网站,展示这些社区的β-diversity(即差异他们的人口结构),但许多类群在放线菌,Bacillales以及其他演化支non-mucosal网站相对丰富。受试的β-diversity系统发育水平突出显示额外的文件1,量化的程度不同粘膜身体网站之间的距离大于等效non-mucosal站点之间的距离。这导致缺乏类群共同粘膜身体网站,因此没有分类群是由LEfSe粘膜的特征作为一个整体。
放线菌目通常最丰富的系统单元(订单)在non-mucosal社区,在几个皮肤样品比例高于90%,最多20%的绝大多数口腔粘膜样品和大幅降低阴道和肠道(图2摄氏度)。从定量的角度,分类顺序放线菌目构成本质上所有的发现门放线菌的成员,除了阴道网站报道大量Bifidobacteriales存在。Bifidobacteriales本身并不是发现粘膜和non-mucosal身体之间的差异丰富网站,因为这是一个功能只有阴道的样品,而不是身体的粘膜网站。的对比许多演化支的丰度与分布是显著的;例如,属Alloscardovia,Parascardovia和Scardovia存在于所有身体站点在丰度很低,而加德纳菌属只在阴道样品过多,超过三个数量级差异。类似的分布被发现的共性Bacillales更低丰度。在属级,丙酸菌属,葡萄球菌,棒状杆菌属和假单胞菌分化的分布和丰富。的葡萄球菌属尤其被LEfSe与LDA分数很高(超过5数量级),反映明显丰富non-mucosal网站(意思是10%、18%和21%的皮肤,retroauricular折痕和身体前鼻孔网站,分别)和持续低丰度在粘膜网站(平均不到0.001%)。
类有多个级别:不同的有氧、无氧和microaerobic社区在人类微生物组
同桌的人类微生物群的厌氧代谢的作用尚未充分研究由于研究这些社区文化的困难。我们因此进一步研究人类微生物群的aerobicity特征在高水平分组的身体网站可用氧气分子水平不同的三个类。的high-O2直接身体接触类包括网站和永久暴露于氧:皮肤、前鼻孔和retroauricular折痕。的mid-O2身体接触类包括口腔和阴道可以直接的网站,但不是永久,气压上暴露,low-O2接触类(肠道)主要是厌氧的。身体网站中包含三个类可能有其他特点除了不同氧气接触,一般来说,这些混杂因素会导致与好氧生活无关的特性,作为生物标志物检测。然而,LEfSe生物步骤确保一致性检测生物标志物特征的所有子类的类和其他类的所有子类。例如,丰富的口腔细菌进化枝由于oral-specific利基并不作为生物标志物检测,除非相同的利基也出现在阴道样品(mid-O其他身体部位2在任何high-O类),而不是礼物2或low-O2单一机构网站。所以LEfSe将检测生物标记物比传统的方法更自信地与好氧生活特征,不包含子类信息。此外,LEfSe特别能够分析顺序类与多个水平,同意建立微生物学,我们观察到特定微生物演化支内无处不在的这三个环境和特点,详细如下(图二维)。
LEfSe允许序数类有超过两个层次来分析在两个不同的保守派。第一个需要每一对之间重要的类群不同阶级的值(在这个例子中,aerobicity;见材料和方法);发现生物标志物必须准确区分所有单个类(高、中期和low-O2)。在这个例子中(图二维;严格的版本),我们发现13演化支LDA分数2以上,显示三种不同的丰富水平。另外,LEfSe可以确定重要类群不同的至少一个(也可能是多个)类值(s)(非严格版本);换句话说,生物标记区分至少有一个单独的类。使用这种方法(图2 e),我们发现60演化支与LDA分数至少2。
使用方法,每个氧气水平是广泛的,其特征是一个特定的进化枝。整体的丰度放线菌门更高的身体网站直接暴露于氧气分子与几个成员寄生在皮肤的放线菌目秩序。放线菌目包括丙酸菌属属,这是高度丰富的皮肤上,在moderate-O低2环境和缺席肠道。Lactobacillales(主要是细菌)是特定于温和的啊2低暴露水平,相反在high-O存在2接触类,再次缺席肠道。类杆菌(特别是拟杆菌在厌氧样本)无处不在;然而,有趣的是,这个家庭的成员更丰富的高氧可用性条件(特别是在皮肤和retroauricular折痕)比在中氧的可用性,显示系统分支内的利基多样性。这是在协议与观测,许多微生物财团显示极端生物地理学的微环境变化对营养,代谢产物和氧气可用性(58,59]。
双歧杆菌和额外的演化支弱势在溃疡性结肠炎的小鼠模型
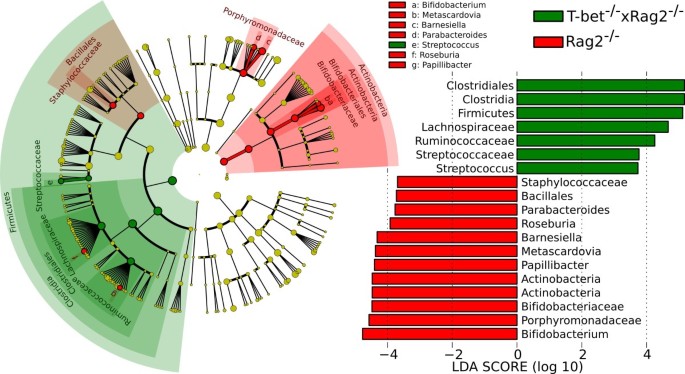
啮齿动物模型建立了提供一个独特的准确和容易处理模型研究肠道微生物群,包括慢性肠道炎症的分子和细胞机制驾驶(60- - - - - -63年]。特别是,炎症性肠病的小鼠模型63年)促进机械的贡献评价肠道微生物群的起始及延续慢性肠道炎症,发生在人类克罗恩病和溃疡性结肠炎64年]。已知一个宿主分子机制之间保持平衡免疫调控和共生的微生物区系T-bet,许多免疫细胞的转录因子表达的子集。失去在缺乏一种自适应免疫系统的结果在一个高度渗透和积极形式的溃疡性结肠炎65年,特别依赖于通过肠道菌群和传染性。我们因此试图调查粪便微生物群的特点在一个小鼠模型的自发结肠炎发生在一群Balb / cT-bet- / -×Rag2- / -老鼠使用16 s rRNA基因宏基因组数据(66年,67年]。
LEfSe是应用于微生物群20的数据T-bet- / -×Rag2- / -(例)和10Rag2- / -(控制)小鼠(数据集提供了额外的文件10丰富),发现19个不同分类演化支(α= 0.01)与LDA分数高于2.0(图3)。这些不同的丰富的演化支是符合我们之前的16 s rRNA-based使用完整的链接层次聚类序列分析和定量实时pcr实验上执行相同的粪便DNA样本(67年]。更具体地说,这标志着Bifidobacteriaceae和损失双歧杆菌属与T-bet- / -×Rag2- / -我们观察到这里可能会解释这结肠炎的积极响应双歧杆菌animalis无性系种群。lactis发酵的牛奶产品验证的低吞吐量的方法(67年]。
对比Rag2- / -(控制)和T-bet- / -×Rag2- / -老鼠(case)强调,在门级,厚壁菌门丰富T-bet- / -×Rag2- / -老鼠,而放线菌是丰富的Rag2- / -老鼠。在协议与以前培养的研究中,双歧杆菌属物种underabundant在T-bet- / -×Rag2- / -老鼠(68年],LEfSe强调几个额外的genus-level演化支,包括专门耗尽Roseburia和Papillibacter否则内过多的厚壁菌门。
在家庭层面上,Rag2- / -浓缩的Bifidobacteriaceae Porphyromonadaceae Staphylococcaceae和T-bet- / -×Rag2- / -浓缩的Lachnospiraceae确认我们的报告68年使用培养和实时定量PCR技术。LEfSe LDA进更多的内容详细的重组这些类群相对P价值发现的这些家庭在我们以前的工作,强调了双歧杆菌,有趣的是,几个演化支在梭状芽胞杆菌。其中包括Rag2- / -特殊技能Roseburia和Papillibacter属属于T-bet- / -×Rag2- / -特殊家庭(Lachnospiraceae和Ruminococcaceae)。的重要地位Metascardovia(Bifidobacteriaceae)Rag2- / -老鼠也是有趣的,因为它可能有一个类似的角色双歧杆菌属因为Metascardovia之前已经观察到主要在口腔68年]。这个分析突出了LEfSe的协议的效应值估计对低吞吐量确认并建议额外的演化支进一步实验研究。
比较与当前使用病毒和微生物宏基因组分析工具从环境数据通路
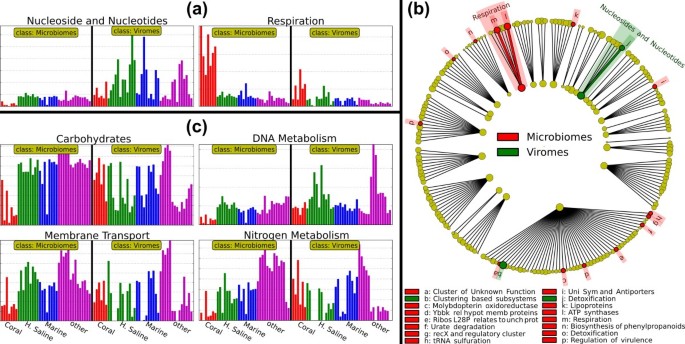
我们应用LEfSe的环境数据69年),一个数据集的目标描述的功能角色viromes(即病毒基因组)和微生物(细菌基因组)。这个任务是用于45)描述Metastats算法在相同的原始数据。在29日高层功能角色(包括未分类角色)子系统层次结构的种子70年]和NMPDR [71年)框架,LEfSe标识只有严格的核苷和核苷酸子系统不同丰富在所有环境子类,特别是高水平viromes微生物。这是一个精确的描述完全蛋白质功能的最常见的病毒基因组中编码,而细菌基因组的编码范围广泛的少特别丰富的功能。当LEfSe放松检测至少一个显著的变化一致,而不是所有环境子类,我们另外确定“呼吸”子系统在微生物对viromes大大丰富,可能反映出均匀有氧细菌新陈代谢捕捉到这些数据。
除了核苷和核苷酸和呼吸子系统,Metastats [45]报告五个其他高级功能角色不同丰富(P= 0.001)。然而,当考虑到子类结构在整个采样环境,这些额外的差异显示更少的变化一致。这是显示在图4,这些病例报告原始数据的直方图和LEfSe的不同结果,Metastats和千瓦单独测试。此外,由于子系统框架层次(三层),LEfSe的结果包括进化分枝图显示每个水平上的显著差异(见图4两级进化分枝图,和额外的文件2三级进化分枝图)。
LEfSe凸显出细菌微生物之间的通路持续微分和viromes内各种环境子类。(一)使用种子[71年目录的功能通路,LEfSe报告之间的核苷和核苷酸代谢和呼吸不同细菌微生物和viromes在环境样品中描述(70年]。前者是重要的使用严格的子类测试,后者更宽容的一个子类的测试。(b)两级进化分枝图报告的重要途径差异可视化使用种子层次结构(参见附加的文件3三级进化分枝图和详细的差异)。(c)Metastats [45]报告另外四通路微分在这些数据(碳水化合物、DNA代谢、膜运输和氮代谢)。只使用千瓦测试部分LEfSe(α= 0.05),我们得到的结果符合Metastats(不含氮代谢)。然而,如下所示,这些子系统的丰度直方图的概述表明他们不太一致的环境(例如,珊瑚和Hyper-saline子类的碳水化合物,膜运输和氮代谢)和失去意义在各个子类(如DNA代谢的子系统)。
考虑所有三个级别的种子功能特异性,LEfSe报告59子系统更丰富更丰富的微生物基因组,只有7在病毒基因组(附加文件3)。细菌基因组编码更大数量和生物分子的功能比大多数病毒基因组的多样性,,因此这些差别是可以预料到的。然而,他们也强调考虑特定于大多数宏基因组(更普遍,生态)分析,通常分析相对丰度。一些很常见的子系统viromes(即核苷和核苷酸)将迫使其他子系统的相对丰度下降,导致明显的under-abundance。子系统检测到特异性可能因此显示这一趋势在一定程度上由于正常化丰度在每个样本。这个问题是特定于LEfSe和Metastats,然而,和必须考虑的解释任何相对丰度数据,宏基因组或(72年]。
机能活动在婴儿和成人的微生物群表明post-weaning微生物专业
正如LEfSe可以确定生物体或通路是否不同丰富几个宏基因组样本,还可以关注个体酶或同源组。Kurokawa等。(73年]分析了13从九成人和四个还在吃奶的婴儿肠道基因组同源基因家族的功能。他们最初这样做是通过比较齿轮(74年,75年)发现在每个metagenome参考数据库;后,白等。(45]Metastats算法应用于直接检测婴儿和成人微生物组之间的差异。使用意义的α值0.01的低基数的类(特别是婴儿类),LEfSe发现366齿轮丰富成人或婴儿基因组——17的LDA得分高于3(附加文件4)。
17齿轮配置文件中LEfSe得分高于3,11也Metastats探测到。六个齿轮不被Metastats(附加文件5)外膜蛋白(COG1538)和Na+借耐多药外排泵(COG0534),浓缩在成人中,衍生品和转座酶灭活(COG2801 COG2963)转录监管机构/糖激酶(COG1940)和转录监管机构(COG1309),丰富了婴儿。所有六个齿轮拥有丰富资料,婴儿和成人个体之间是完全重叠(除了COG1538,成人的最低水平略低于最高的婴儿),因此名义上很歧视。另一方面,在192齿轮Metastats发现,9没有检测到LEfSe即使在LDA得分最低阈值(附加文件6)。所有婴儿和成人之间具有重叠的丰度值类(至少两个,通常,最高的样本越少丰富类重叠被认为是更丰富的类)。这种歧视性的缺乏权力阻止LEfSe突出成人和婴儿之间的差异是显著的,特别是考虑到低的婴儿数量样品。
有趣的是,LEfSe截然不同的列表的功能活动的核心婴儿和成人微生物暗示“通才”微生物活动在早期生活随着时间的推移和专业化(76年]。事实上,检查最高的五个不同的丰富的齿轮为每个类尺度效应,我们发现婴儿非常高层次的官能团与广泛的转录调节(COG1609、COG1940 COG1309和COG3711)。在成人中,所有五个代表更专门的同源组,包括COG1629(外膜受体蛋白,主要是铁运输),COG1595 (DNA-directed RNA聚合酶专业σ亚基,sigma24同族体),和COG4771 (ferrienterochelin外膜受体和大肠杆菌素)。由于不同的数量丰富的齿轮非常高(366),这个观察只是强调候选人生物标记列表的顶部由于LEfSe大小的影响量化,它允许最类间特征差异出现。出于同样的原因,我们可以很容易地证实,糖代谢中起着至关重要的作用在婴儿肠道和铁代谢在成人中,已经在(45,73年];得分最高的齿轮LDA的确拥有糖和葡萄糖为婴儿和成人iron-related功能功能活动。
LEfSe达到合成数据中假阳性率很低
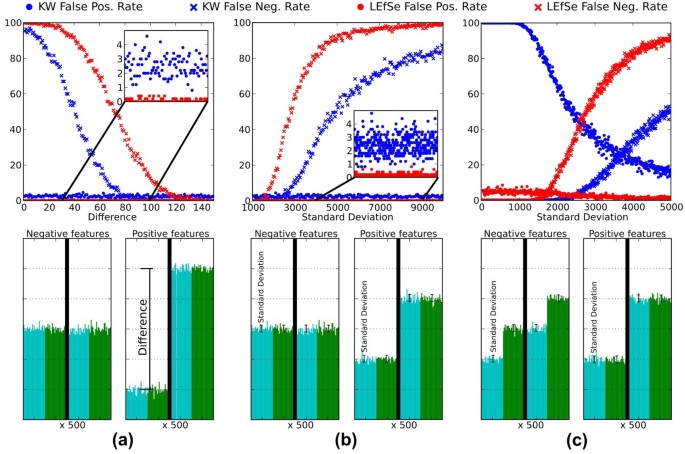
我们进一步调查的能力LEfSe检测生物标志物使用合成高维数据(见材料与方法的描述数据集)相比,仅千瓦测试(一种非参数的方差分析(方差分析)和Metastats [45]。LDA的效果一步LEfSe这里不考虑为简单起见,和人工数据详细的图5。
比较LEfSe千瓦单独试验假阳性和负率合成数据。这两个测试α= 0.05在所有情况下,和三个人工数据集包括100个样本,其中每一个都可以在两个类中,两个基数25的子类。的样本包括1000合成功能微生物类群,途径,等等;一半是消极的(而不是生物标记)和其他积极的一半。(一)LEfSe千瓦假积极的和消极的利率增加的值类方法之间的区别。消极的特性与参数正态分布(μ= 10000,σ= 100)类;积极特性包含类越来越多不同的意思。(b)性能标准偏差类内变化(而不是意味着之间的区别,固定在2000年)。(c)性能随着标准差增加内不一致的子类。负面特性有子类样本来自同一正态分布(因此不代表一致的生物标记)。(b)的积极特性分布。在所有情况下,LEfSe牺牲一小部分为了实现假阳性假阴性率接近于零的水平,确保生物标记的目标大效果将可再生和生物可判断的。
从理论上讲,前两个实验的设置(图5 a、b为千瓦)完全匹配应用程序条件测试。假阳性率(平均2.5%,不管距离特性的手段和正态分布的标准偏差)实际上是一致的α值0.05,考虑到负面特性总数的一半。LEfSe行为定性千瓦非常相似,但大大降低假阳性率(少于0.5%的绝大多数情况下对中值为2.5%)和较高的假阴性率。在生物学上,假阳性往往比假阴性(视为更戏剧性的77年- - - - - -79年];这通常归因于这样一个事实,这是不受欢迎的投资昂贵的实验后续的假阳性,而在高吞吐量设置,一些真正的阳性大于假阴性被知晓。减少假阳性的动机,我们认为至少LEfSe执行以及千瓦当没有意义的子类结构是可用的。另一方面,当子类可以确定内部类和他们中的一些人不同意这种趋势在类,LEfSe执行定性和定量比千瓦(图5度)。假阳性是事实上总是大大低于千瓦,而假阴性高只有非常嘈杂的特性。Metastats [45]似乎达到千瓦(附加文件非常相似的结果7关于LEfSe)相同的缺点。
结论
获得深入的结构、组织和微生物群落的功能提出了作为研究的一个主要挑战当前的十年(80年),它将通过实验和计算宏基因组分析。为此,我们开发了LEfSe算法比较宏基因组研究允许微生物类群的特征具体实验或环境条件、途径和生物机制的检测是否被充分代表在不同的社区,并在哺乳动物微生物宏基因组生物标志物的鉴定。LEfSe这里显示是有效地检测不同的丰富特性在人类微生物组(典型的粘膜或有氧类群)和小鼠结肠炎模型。比较与现有统计方法和先进的宏基因组分析环境、婴儿肠道微生物组,和合成数据显示,LEfSe持续提供降低误判率,可以有效地帮助解释生物学基础微生物群落的差异。
这些发现证明了类的概念解释包括统计和生物学意义是非常有益的在解决统计挑战与高维生物标志物的发现(28,81年,82年]。特别是LEfSe决定特性可能能够解释差异条件而不是功能,仅仅拥有类间分布不均匀。这是不同于最新的统计方法(45),类似于生物学先验知识的结合被证明是非常成功的在最近的全基因组关联研究(83年- - - - - -85年]。此外,特别是在(通常是嘈杂的)宏基因组数据集,效果可以作为正交措施补排名基于生物标记物P单独的值。类之间的差异非常显著(低P值),但如此之小,他们不太可能生物表型差异负责。另一方面,相对较大的生物标志物P值(例如,0.01)可能对应于一个巨大的影响大小,通过技术与统计显著性减少噪音。LEfSe调查两方面计算通过测试的一致性和类间差异特性丰富的效应大小的结构问题。这是随后执行标准统计显著性测试和集成LEfSe通过评估生物意义的组样本在每个子类条件之一。这种耦合与生物统计方法的一致性和效应值估计减轻可能的工件或统计不均匀性是常见的宏基因组数据,例如,主题或极端的差异的存在长尾的稀有生物(32,86年]。同样,尽管多个假设纠正统计学意义讲可能重现性结果,估计效果在高维设置是至关重要的,以解决生物一致性和可解释性。
生物学强调了这些调查与宏基因组的潜力对微生物生态学和转化应用。例如,某些细菌演化支经常发现生物标志物甚至在多样化的环境中,这表明一些物种可以在惊人的适应condition-specific礼仪。葡萄球菌Bacillales,例如,有区别的粘膜组织,有氧条件下,小鼠结肠炎,而没有任何变形菌门一直描述的这种情况下,即使他们总是代表了很大一部分的社区。这些观察结果可能反映了广泛的微环境的异质性和多面手的共存和专家细菌(87年- - - - - -89年]。
除了这些见解微生物学,宏基因组生物标记,包括特定生物的丰度、丰度的整个演化支,或存在缺乏特定的生物,可以描述主机表型,生活方式,饮食,以及疾病(5- - - - - -10]。如果消耗的双歧杆菌属物种在溃疡性结肠炎证明发生在人类疾病病因的早期,这可比转变微生物群的潜在的应用在人类疾病的检测90年,91年),特别是一些细菌财团可以检测到的变化容易和便宜。口腔微生物生物标记,例如,可以很容易地获得和分析微阵列芯片针对细菌分析(92年]。这些看起来特别有前途的临床应用(11),唾液微生物群落的似乎代表一个潜在的代理对其他人类微生物群(93年]。其他重要的宏基因组分析的临床应用包括益生菌治疗(94年,95年)和微生物移植(96年- - - - - -99年胃肠道疾病。
LEfSe,生物类的计算方法比较详细,从而导致微生物群落和导游的理解生物学家在检测小说宏基因组生物标志物。算法的有效性一直在强调真实和合成数据几个实验中,我们成功地在多个上下文中host-associated微生物群和环境微生物的特征。支持持续的宏基因组分析,我们实现了LEfSe友好的web应用程序,它可以提供原始数据和publication-ready图形化的结果,包括报告检测到微生物变化分类树的视觉和生物学上的总结。LEfSe在线免费银河系中工作流框架(46,47在以下链接()48]。
材料和方法
LEfSe算法在结果部分,介绍了概述和图6详细说明的格式输入(一个矩阵n行和米列)和执行的三个步骤计算工具:千瓦等级和测试(49)类,成对Wilcoxon测试(50,51不同的类的子类之间),LDA (52在相关的特性。
每一个n特性是用positive-valued向量表示包含其丰度米样本,每个样本与价值观描述它的类,可选地,子类和/或原始主题。阶乘KW等级和测试应用于每个特性对类因素;子类和主题信息作为分层子组当礼物。特性,根据KW等级和测试,不违反类间的零假设相同的值分布(违约P价值,α= 0.05)没有进一步分析。成对Wilcoxon测试应用于保留特性属于不同的类的子类。对于每个功能,成对Wilcoxon测试是不满意如果至少有一个比较有一个子类P价值高于选择α或者变异是不等于的符号在所有比较。例如,如果一个功能出现在两类样本三个子类,子类之间的所有九个比较在不同的类必须违反零假设,和所有的迹象中位数之间的差异必须是一致的。通过成对Wilcoxon测试的功能被认为是成功的生物标志物。LDA模型最终建立与类作为因变量,其余特性值,子类和主题值作为独立的变量。这个模型被用来估计的影响大小,所获得的平均差异类方法(使用修改的特性值)与类之间的差异意味着沿着线性判别第一轴,这同样重量特性的可变性和歧视性的能力。LDA得分为每个生物标志物得到计算这个值的对数(基地10)缩放后的[1,106]区间,不管LDA的绝对值分数,它引发的排名生物标志物的相关性。鲁棒性,LDA另外支持引导(默认30倍)和随后的平均。
LEfSe的前两个步骤采用非参数测试,因为宏基因组数据的性质。相对丰度,在大多数情况下,违反的主要假设的典型参数测试(正常人群在每个类),而非参数测试更健壮的潜在分布数据,因为他们是传播变为免费的方法。唯一Wilcoxon和千瓦测试的假设是,每个类的分布是相同的形状可能不同的中位数。例如,有机体违反的双峰或多峰丰度分布参数测试的假设,而不是那些非参数测试,除非数量的峰值分布(或者更普遍,分布)的形状也改变类之一。LDA用于效应值估计作为我们的实验确定方法相比它更准确地估计生物一致性组的差异意味着/中位数或支持向量机(svm) [One hundred.]。比较LDA和SVM方法对效应值估计的低吞吐量的小鼠溃疡性结肠炎模型(生物验证生物标记中可用的67年])是我们补充材料(附加文件报告8和9),显示了LDA的优势对upranking特性的潜在生物的兴趣。从理论上讲,这是出于LDA的能力找到最高的方差和支持向量机的轴的关注特性结合预测能力而不是单一特征的相关性。注意,当我们执行类比较而不是类预测,值得说明的是,效果估计精度的算法不是直接连接的预测能力(例如,支持向量机方法通常被认为是比LDA预测更准确)。
多级策略
比较以上两类需要特殊的策略应用Wilcoxon和LDA的步骤,而阶乘千瓦测试已经适合此设置。我们多级战略Wilcoxon测试取决于问题特定的策略选择的用户定义特征分布在不同n类。在最严格的策略中,我们要求所有n丰富的功能在统计上显著不同的在所有n类。实现这一策略,称为“严格”,要求所有Wilcoxon测试类之间是显著的。更宽松的策略,称为“非严格”,认为作为生物标志物特征如果至少有一个类是明显不同于其他所有人。更宽松的策略只需要满足的一个子集Wilcoxon测试。无论战略,LDA一步总得分最高的检测报告在所有两两类比较。
子类结构变体编码不同生物假说
生物标记类的不同的解释比较问题实现LEfSe通过修改要求成对Wilcoxon子类之间的比较。如果类包含子类代表不同的地层,我们只测试比较在每个子类(图相同4)。例如,评估治疗的效果在两个子类型相同的疾病,我们比较预处理和后处理的水平在每个子类和要求趋势观察到子类的类级别的独立是很重要的。要实现此变体,LEfSe执行Wilcoxon一步只比较具有相同名称的子类。另外,子类可能代表的功能水平可能不同但反是的问题没有规定明确的分层(图2)。在这两个设置,我们明确地要求所有成对比较拒绝零假设检测生物标志物;因此,不需要多个测试修正。
子类包含几个样品
当几个样品,非参数测试像Wilcoxon减少了功率检测的差异。这可以影响LEfSe当子类非常小,防止整体测试甚至拒绝零假设。出于这个原因,小子类应该避免在可能的情况下,例如,通过将他们排除在问题或组成的所有子类与小的基数。的情况下,删除或分组子类是不可能的或破坏了生物的一致性分析,LEfSe替代品Wilcoxon测试和一个测试来比较是否子类中位数不同预期的迹象。用户可以选择这个值比较的子类基数阈值代替Wilcoxon测试。
参数设置
除了结果,如上所述,否则所有本研究实验运行与LEfSeα为成对测试参数设置为0.05测试,正常类和子类和阈值的对数得分LDA分析被设置为2.0。这些参数的严格很容易可调(也通过web接口),允许用户检测较低的生物标志物P值和/或更高的效果,例如,要优先考虑额外的生物实验和验证。LDA的分数都是由引导30多个周期,每个采样三分之二的数据替换,LDA的最大影响系数LDA得分三个数量级。
数据描述
除了如上所述否则,分类丰度为16 s样本过滤产生的序列读取使用RDP分类器(101年与信心低于80% rebinned],“不确定”。下面描述的数据集,最后输入LEfSe是一个矩阵的相对丰度从读计数与每个样本归一化和获得。Witten-Bell平滑(102年)是用来容纳罕见的类型,但由于LEfSe的非参数方法,这微小的影响发现生物标志物和LDA得分。这也使得我们的生物标志物的发现方法来避免大多数序列质量问题的影响,只要任何排序偏差在不同条件下均匀,没有特定的假设上的统计分布和噪声模型是由算法作为非参数方法是标准的。
人类微生物组的数据
正常的16 s rRNA-based phylometagenomic数据集(健康)人类微生物组是通过人类微生物组计划(13),由454名FLX钛序列生成V3 V5变量地区获得301年样本24名健康受试者(12男,12女性)进入一个临床网站在休斯顿,TX。这些样本涵盖18个不同的网站,包括6个主体网站类别:口腔样本(9),肠道示例(1),阴道(3)样品,retroauricular折痕(2)样品,鼻腔(1样本)和皮肤(2)样品。详细协议用于注册、抽样、DNA提取、16 s扩增和测序人类微生物组项目数据分析和协调中心的网站(103年),和在别处也有描述55,56]。总之,基因组DNA分离使用莫生物PowerSoil工具包(104年)和接受16 s放大使用引物设计结合FLX钛适配器和样本条形码序列,使定向测序覆盖变量地区V5部分V3(引物:357 f 5“-CCTACGGGAGGCAGCAG-3”和926 r 5“-CCGTCAATTCMTTTRAGT-3”)。产生的序列处理使用数据管理管道中实现mothur [41),减少了测序错误率小于0.06%,模拟社区进行验证。作为管道参数的一部分,通过最初的质量控制步骤,一个明确的不匹配样本条形码和两个错配PCR扩增引物是允许的。序列与一个模棱两可的基地打电话或均聚物超过八个核苷酸在随后的分析中删除,正如前面提出的(105年]。基于提供的质量分数,所有序列都修剪基地调用时得分低于20是遇到了。所有序列都使用NAST-based序列一致对准器自定义参考基于席尔瓦对齐(106年,107年]。序列的短于200个基点,或者不一致预期的地区参考对齐被移除的进一步分析。嵌合序列被确定使用mothur ChimeraSlayer算法的实现(108年]。独特的读取与MSU RDP分类器分类v2.2 [58)使用提出的分类(109年),在RDP维持(RDP 10数据库版本6)。16 s rRNA读取序列中的可用阅读档案在[110年]。
T-bet- / -×Rag2- / -和Rag2- / -鼠标数据
T-bet- / -×Rag2- / -和Rag2- / -老鼠,他们的饲养,他们的食物被描述在67年]。动物研究和实验,根据哈佛大学的批准和开展动物常务委员会以及美国国立卫生研究院的指导方针。收集、处理和提取的粪便样本中的DNA进行描述(67年]。16 s rRNA的版本5和版本6的区域基因与纠错条形码针对放大和多路复用焦磷酸测序。测序进行使用罗氏FLX基因组定序器DNAVision(该市,比利时)和数据预处理去除序列与低质量分数。有7579±2379高质量的16 s读取/样本平均读278个基点的长度。
病毒和微生物环境数据
我们从网上检索的补充材料69年]80基因组(42 viromes, 38微生物)。我们确定了三个环境包含至少7样品和分组成珊瑚,hyper-saline,和海洋子类;第四个子类,其他组所有环境很少有样品。
婴儿和成人微生物组的数据
九成人的齿轮配置文件和四个还在吃奶的婴儿肠道菌群是获得的补充材料73年)和修改的在这项研究中使用。
合成数据集
我们建立了三个集合的人工数据集为了比较LEfSe千瓦和Metastats。所有数据集有1000特性和100个样本属于均匀两类,并从高斯正态分布采样的值。两个类中的样本进一步分为4个子类(两类)与平等的基数。1000功能,500因此跨类和特性有不同的手段应该作为生物标志物检测(优点),其他500个特性是均匀分布在类或至少一个子类中两类和不应该被歧视(负功能)。评估方法评估假阳性率(生物标记对发现的错误数的总数特性)和假阴性率(数量的正确检测到non-discriminant功能特性的总数,即敏感性)。这三个数据集的集合(图形如图5)不同子类中的值的分布和正态分布的均值/标准差。(一)子类在相同的类具有相同的参数(因此子类组织是没有意义的)。消极的特性都有μ= 10000和σ= 100,而积极的特性的一个类μ= 10000 - t(σ= 100),另一个μ= 10000 + t(σ= 100),t是一个参数从1到150。所有方法的性能评估在t参数的常规步骤。(b)数据集在这个集合中定义一样收集与t = 1000(一个),但所有的数据集和σ从1000年到10000年不等。(c)负类分布在第三组有不同的子类。特别是,第二个子类的第一个类有相同意思的第一个第二个类的子类。另外两个子类有不同的手段(μ= 10000 - t和μ= 10000 + t, t = 1000),但功能是不考虑微分自子类之间的区别是不一致的。积极的特性是相同的方式定义数据集(b)。
该方法的实现和可用性
LEfSe在Python实现,利用R统计职能的硬币(111年)和质量(112年]rpy2图书馆通过[113年)和matplotlib (114年库的图形输出。银河系中LEfSe提供图形界面框架(46,47),它允许用户选择参数(主三紧缩参数、多级设置和其他计算,统计,和图形喜好),模块间的管道数据在一个工作流框架中,生成可发布图形输出,并将这些结果与其他统计和宏基因组分析。LEfSe可用在[48]。
缩写
- 英国石油公司:
-
碱基对
- 千瓦:
-
克鲁斯卡尔-沃利斯
- LDA:
-
线性判别分析
- LEfSe:
-
线性判别分析效应大小
- 聚合酶链反应:
-
聚合酶链反应
- RDP:
-
核糖体数据库项目
- 支持向量机:
-
支持向量机。
引用
Golub TR:发现癌症的分子分类:类和类预测通过基因表达的监控。科学。1999年,286:531 - 537。10.1126 / science.286.5439.531。
Petricoin EF, Ardekani,希特BA,莱文PJ, Fusaro VA,斯坦伯格SM,米尔斯GB,西蒙·C, Fishman哒,科恩EC,李欧塔LA:使用血清蛋白质组学模式识别卵巢癌术语表。柳叶刀》。2002年,359:572 - 577。10.1016 / s0140 - 6736 (02) 07746 - 2。
Tothill RW,修补AV,乔治J,布朗R,福克斯某人,舀出,约翰逊DS, Trivett可,Etemadmoghadam D, Locandro B, Traficante N, Fereday年代,挂着是的,卫生,介入我,澳大利亚卵巢癌研究小组,Gertig D,看来,Bowtell DD:小说的分子亚型浆液性卵巢癌和endometrioid与临床结果。癌症研究杂志2008年14:5198 - 5208。10.1158 / 1078 - 0432. - ccr - 08 - 0196。
李魏X, c:探索在——和类间相关性分布的肿瘤分类。《美国国家科学院刊。2010年,107:6737 - 6742。10.1073 / pnas.0910140107。
德菲利波C, Cavalieri D, Di Paola M, Ramazzotti M, Poullet JB,设计学院年代,Collini年代,Pieraccini G, Lionetti P:饮食在塑造人类肠道微生物群的影响揭示了来自欧洲和非洲农村的儿童进行比较研究。《美国国家科学院刊。2010年,107:14691 - 14696。10.1073 / pnas.1005963107。
恩伯PJ, Backhed F,富尔顿L,戈登霁:食源性肥胖与显著但可逆改变鼠标远端肠道微生物组。细胞微生物。2008年,3:213 - 223。10.1016 / j.chom.2008.02.015。
雷再保险,彼得森哒,戈登霁:生态进化力量塑造在人类肠道微生物多样性。细胞。2006年,124:837 - 848。10.1016 / j.cell.2006.02.017。
Manichanh C, Rigottier-Gois L, Bonnaud E, Gloux K, Pelletier E, Frangeul L, Jarrin C,纳R·坎姆顿P,有主P,罗卡J,多尔J:减少粪便微生物群的多样性在克罗恩病了一个宏基因组的方法。肠道。2006年,55岁:205 - 211。10.1136 / gut.2005.073817。
索科尔H, Seksik P Furet JP Firmesse O, Nion-Larmurier我Beaugerie L, Cosnes J, Corthier G, Marteau P,多尔J:低项Faecalibacterium prausnitzii结肠炎微生物群。Inflamm肠道说。2009年,15:1183 - 1189。10.1002 / ibd.20903。
奥多瓦斯JM,驼鹿V:宏基因组:心血管疾病的微生物的作用。当今脂醇。2006年,17:157 - 161。mol.0000217897.75068.ba 10.1097/01.。
张L,汉森BS, Camargo点,黄DT:唾液牙周疾病的生物标志物的临床价值。牙周病学2000。2009年,51:25-37。10.1111 / j.1600-0757.2009.00315.x。
张L,法雷尔JJ,周H, Elashoff D, D,类似公园NH,贾D,黄DT:唾液转录组生物标志物的检测可切除的胰腺癌。胃肠病学。2010年,138:949 - 957。10.1053 / j.gastro.2009.11.010。e947
NIH HMP工作组,彼得森J,停车库建设年代,乔凡尼M,麦克因尼斯P,王L,城堡是的,Bonazzi V, McEwen我,Wetterstrand KA, C,贝克CC, Di弗朗西斯科·V,克罗夫特的TK,卡普RW,朗斯福德RD,惠灵顿CR、Belachew T,赖特M,吉布林C,大卫•H米尔斯M,所罗门R,马林斯C, Akolkar B, Begg L, C,戴维斯Grandison L,卑微的M,节日J, et al:美国国立卫生研究院人类微生物组计划。基因组研究》2009年19:2317 - 2323。
Hamady M, Fraser-Liggett厘米,恩伯PJ,牧地,骑士R,戈登霁:人类微生物组的项目。大自然。2007年,449:804 - 810。10.1038 / nature06244。
Magrini V,恩伯PJ,草地,狂欢节,马浩德马,戈登霁:一个肥胖相关的肠道微生物与增加能源产量的能力。大自然。2006年,444:1027 - 1131。10.1038 / nature05414。
邓肯•SH Lobley通用电气,Holtrop G,因斯J约翰斯通,路易P,弗林特HJ:人类结肠微生物群与饮食有关,肥胖和减肥。Int J肥胖(Lond)。2008年,32:1720 - 1724。10.1038 / ijo.2008.155。
恩伯PJ, Ridaura VK、信仰JJ,雷伊菲,骑士R,戈登霁:饮食对人类肠道微生物的影响:人性化的无菌老鼠的宏基因组分析。2009年Sci Transl地中海。1:6 ra14 - 10.1126 / scitranslmed.3000322。
高Z,曾碳氢键,闪光灯,裴Z,布莱塞MJ:实质性改变的皮肤细菌生物银屑病病变。《公共科学图书馆•综合》。2008年,3:e2719 - 10.1371 / journal.pone.0002719。
Tringe SG,冯仅仅是C,小林,Salamov AA,陈K, Chang HW, Podar M,短JM, Mathur EJ,德JC,博克P, Hugenholtz P,鲁宾EM:比较微生物群落的宏基因组。科学。2005年,308:554 - 557。10.1126 / science.1107851。
Solovyev VV,艾伦EE、Ram RJ Rokhsar DS,查普曼J,理查森点,泰森GW,鲁宾EM,班菲尔德摩根富林明,Hugenholtz P:通过重建社区结构和代谢的微生物基因组的环境。大自然。2004年,428:37-43。10.1038 / nature02340。
Lecuit M, Lortholary O: Immunoproliferative小肠疾病联系在一起空肠弯曲杆菌。地中海Mal感染。2005年,17 (2):35 S56-58。
Relman哒,施密特TM,麦克德莫特谋杀北爱皇家RP, Falkow史:无教养的杆菌的鉴定惠普尔氏病。郑传经地中海J。1992年,327:293 - 301。10.1056 / NEJM199207303270501。
奥克利BB,菲德勒TL,马拉佐JM,弗雷德里克·DN:人类阴道细菌群落的多样性和对临床定义细菌性阴道炎。:环境Microbiol。2008年,74:4898 - 4909。10.1128 / AEM.02884-07。
呸! VG, Tibshirani R,楚G:意义的微阵列分析应用于电离辐射的反应。《美国国家科学院刊。2001年,98:5116 - 5121。10.1073 / pnas.091062498。
史密斯GK:线性模型和经验贝叶斯方法评估微分表达式在微阵列实验。统计:麝猫杂志。2004年,3:第三条-
克拉克R, Ressom HW,王,宣J,刘MC,国家Ea,王Y:高维数据空间的属性:探索基因和蛋白质表达的影响的数据。Nat癌症。2008年,牧师8:37-49。10.1038 / nrc2294。
天鹅Ka,柯蒂斯·德·McKusick KB, Voinov AV, Mapa Fa,坎希拉先生:高通量基因图谱秀丽隐杆线虫。基因组研究》2002年12:1100 - 1105。
伍力JC,你们Y:宏基因组:事实和工件,*和计算挑战。J第一版Sci抛光工艺。2009年,25岁:71 - 81。
恩伯PJ, Hamady M, Yatsunenko T, Cantarel提单,邓肯,雷再保险公司Sogin ML,琼斯WJ,罗伊英航Affourtit JP,埃霍尔姆M, Henrissat B,希思AC,骑士R,戈登霁:肥胖和苗条的核心肠道微生物组双胞胎。大自然。2009年,457:480 - 484。10.1038 / nature07540。
Pedros-Alio C:海洋微生物多样性:可以决定吗?。趋势Microbiol。2006年,14:257 - 263。10.1016 / j.tim.2006.04.007。
Huber Ja Sogin ML,莫里森HG,韦尔奇D,休斯SM,尼尔公关实习JM, Herndl GJ:微生物多样性在深海勘探“罕见的生物圈”。《美国国家科学院刊。2006年,103:12115 - 12120。10.1073 / pnas.0605127103。
Gobet,海棠C, Ramette答:多元截止水平分析(MultiCoLA)大型社区的数据集。核酸研究》2010年38:e155 - 10.1093 / nar / gkq545。
Dethlefsen L, McFall-Ngai M, Relman DA:生态和进化视角human-microbe共生和疾病。大自然。2007年,449:811 - 818。10.1038 / nature06245。
Huson DH、欧什AF气J,舒斯特尔SC:梅根宏基因组数据的分析。基因组研究》2007年,17:377 - 386。10.1101 / gr.5969107。
Mitra年代,吉尔伯特是的,D, Huson DH:比较多个基因组使用系统网络基于生态指标。2010年ISME j ., 4: 1236 - 1242。10.1038 / ismej.2010.51。
Mitra年代,Klar B, Huson DH:视觉和统计的比较基因组。生物信息学。2009年,25岁:1849 - 1855。10.1093 /生物信息学/ btp341。
公园DH, Beiko RG:识别生物相关的宏基因组社区之间的差异。生物信息学。2010年,26日:715 - 721。10.1093 /生物信息学/ btq041。
Lozupone C,骑士R: UniFrac:一个新的比较微生物群落的系统方法。:环境Microbiol。2005年,71:8228 - 8235。10.1128 / aem.71.12.8228 - 8235.2005。
Meyer F, Paarmann D, D’索萨M,奥尔森R,玻璃EM, Kubal M, Paczian T,罗德里格斯,史蒂文斯R, Wilke, Wilkening J,爱德华兹RA:宏基因组拉斯特服务器——公共资源的自动系统发育和功能基因组分析。BMC生物信息学。2008年,9:386 - 10.1186/1471 - 2105 - 9 - 386。
Kristiansson E, Hugenholtz P, Dalevi D: ShotgunFunctionalizeR: R-package基因组的功能比较。生物信息学。2009年,25岁:2737 - 2738。10.1093 /生物信息学/ btp508。
城堡PD,威斯克SL, Ryabin T,小厅,哈特曼M,霍利斯特EB, Lesniewski RA,奥克利BB,公园DH,罗宾逊CJ, Sahl JW,度假B, Thallinger GG,范霍恩DJ,韦伯CF:引进mothur:开源,独立于平台的,支持的软件描述和比较微生物群落。:环境Microbiol。2009年,75:7537 - 7541。10.1128 / AEM.01541-09。
Goll J, Rusch D, Tanenbaum DM、Thiagarajan M,李K,给我买fergie的。英航Yooseph S: METAREP:同时,宏基因组报告- - - - - -一个开源高性能比较宏基因组的工具。生物信息学。2010年,26日:2631 - 2632。10.1093 /生物信息学/ btq455。
Jolliffe:主成分分析。1986年,纽约:斯普林格出版社
高尔半岛JC:一些距离属性潜伏根和向量的方法用于多变量分析。生物统计学。1966年,53:325 - 338。
白色小纳N,流行M:统计方法在临床宏基因组样本检测不同的丰富特性。公共科学图书馆第一版医学杂志2009年5:e1000352 - 10.1371 / journal.pcbi.1000352。
Goecks J, Nekrutenko,泰勒J:星系:一个全面的方法来支持可访问,可再生的,透明的计算在生命科学的研究。基因组医学杂志2010年11:r86 - 10.1186 / gb - 2010 - 11 - 8 r86。
Blankenberg D·冯·昆明理工G, Coraor N, Ananda G,拉撒路R,摩根M, Nekrutenko,泰勒J:星系:一个基于web的基因组分析实验的工具。咕咕叫Protoc杂志。2010年,第十九章:单元19.10.1-21 -
Kruskal WH,沃利斯佤邦:使用标准之一方差分析的行列。J是Stat Assoc。1952年,47:583 - 621。10.2307 / 2280779。
Wilcoxon F:个人比较排名方法。生物识别技术。1945年,1:80 - 83。10.2307 / 3001968。
曼HB,惠特尼博士:在一个测试的两个随机变量是否随机大于另一个。Ann数学统计。1947年,18:50 - 60。10.1214 /中耳炎/ 1177730491。
费舍尔RA:使用多个测量分类问题。安优生学。1936年,7:179 - 188。10.1111 / j.1469-1809.1936.tb02137.x。
木豆贝洛F,赫特尔C:口腔作为肠道乳酸杆菌的天然宿主。系统:Microbiol。2006年,29日:69 - 76。10.1016 / j.syapm.2005.07.002。
Costello EK到来CL, Hamady M,菲勒N,戈登,骑士R:细菌社区人体栖息地的变化在时间和空间。科学。2009年,326:1694 - 1697。10.1126 / science.1177486。
人类微生物组项目临床抽样协议。(http://hmpdacc.org/micro_analysis/microbiome_sampling.php]
特纳JR:肠道粘膜屏障功能在健康和疾病。Nat Immunol。2009年,牧师9:799 - 809。10.1038 / nri2653。
科尔JR,王Q, Cardenas E,鱼J,柴B,法里斯RJ, Kulam-Syed-Mohideen, McGarrell DM,沼泽T, Garrity通用,Tiedje JM:核糖体数据库项目:改进的比对和核糖体rna分析的新工具。核酸研究》2009年,37:d141 - 145。10.1093 / nar / gkn879。
希尔伯特F, Scherwitzel M, Paulsen P,绍斯塔克议员:生存空肠弯曲杆菌条件下的大气氧含量的支持下假单胞菌spp .围住Microbiol。2010年,76:5911 - 5917。10.1128 / AEM.01532-10。
Godon J J, Moriniere J, Moletta M, Gaillac M, Bru V,为jp:稀有与特定的生态位相关联的细菌世界:“增效剂”的例子。环境Microbiol。2005年,7:213 - 224。10.1111 / j.1462-2920.2004.00693.x。
沙阿Sa,布朗辛普森SJ,低频,Comiskey M,德容YP,艾伦·D Terhorst C:结肠腺癌发展溃疡性结肠炎的小鼠模型。Inflamm肠道说。1998年,4:196 - 202。
皮萨罗T:小鼠模型为克罗恩病的研究。摩尔地中海趋势。2003年,9:218 - 222。10.1016 / s1471 - 4914 (03) 00052 - 2。
琼斯Panwala厘米,JC,维尼杰:炎症性肠病的小说模式:老鼠多重耐药基因缺陷,mdr1a,自发发展结肠炎。J Immunol。1998年,161:5733 - 5744。
Wirtz年代,纽赖特MF:炎症性肠病的小鼠模型。阿德药启2007 59:1073 - 1083。10.1016 / j.addr.2007.07.003。
裁缝RB:疾病的机制:克罗恩病和溃疡性结肠炎的发病机制。Nat中国Pract杂志。2006年,3:390 - 407。10.1038 / ncpgasthep0528。
Garrett WS,主通用,Punit年代,Lugo-Villarino G, Mazmanian SK, Ito年代,格利克曼约,格里姆彻韩:传染病引起的溃疡性结肠炎T-bet不足的先天免疫系统。细胞。2007年,131:33-45。10.1016 / j.cell.2007.08.017。
Garrett WS,加利尼CA, Yatsunenko T, Michaud M,杜布瓦,德莱尼ML, Punit年代,Karlsson M, Bry L,格利克曼约,戈登,Onderdonk AB,格里姆彻韩:肠杆菌科与肠道微生物群一致行动,产生自发的和母亲般地传播结肠炎。细胞微生物。2010年,8:292 - 300。10.1016 / j.chom.2010.08.004。
Veiga P,加利尼CA,比尔C, Michaud M,德莱尼ML,杜布瓦,Khlebnikov, van Hylckama Vlieg我,Punit年代,格利克曼约,Onderdonk,格里姆彻LH, Garrett WS:双歧杆菌animalis无性系种群。lactis发酵奶产品可以减少炎症通过改变一个利基colitogenic微生物。《美国国家科学院刊。2010年,107:18132 - 18137。10.1073 / pnas.1011737107。
渡边啊,主管Yoshimi B Kai-P L,菅伸子M: Metascardovia criceti将军11月,Sp。11月,仓鼠牙菌斑。Microbiol Immunol。2007年,51:747 - 754。
Dinsdale EA,爱德华兹RA,大厅D, F安利格,布莱巴特M, Brulc JM,可•福尔兰正M, Desnues C,海恩斯M, L, McDaniel L,马莫兰,纳尔逊•柯尼尔森C,奥尔森R,保罗J,布里托BR,阮Y,天鹅BK,史蒂文斯R,情人节DL,瑟伯房车,Wegley L,白色的英航,Rohwer说道F:九生物群落的功能宏基因组分析。大自然。2008年,452:629 - 632。10.1038 / nature06810。
巴特勒Overbeek R,贝格利T, RM, Choudhuri合资,壮族,为什么Cohoon M, de Crecy-Lagard V,迪亚兹N, Disz T,爱德华R, Fonstein M,弗兰克•埃德格迪斯年代,玻璃EM, Goesmann,汉森,Iwata-Reuyl D,詹森R,静态N, Krause L, Kubal M,拉森N,左翼B, McHardy AC, Meyer F, Neuweger H,奥尔森G,奥尔森R,奥斯特曼,Portnoy V, et al:子系统基因组注释和其使用方法在项目1000基因组注释。核酸研究》2005年,33:5691 - 5702。10.1093 / nar / gki866。
格林JM,柯林斯F,莱夫科维茨EJ,鲁斯D,本篇报告RH, Sobral B,史蒂文斯R,白啊,Di弗朗西斯科·V:国家过敏症和传染病研究所的生物信息学资源中心:病原体信息学的新资产。感染Immun。2007年,75:3212 - 3219。10.1128 / IAI.00105-07。
克雷布斯CJ:生态:分布和丰富的实验分析。2008年,本杰明·卡明斯
伊藤Kurokawa K, T, Kuwahara T,大岛渚K (H,丰田章男,Takami H,盛田昭夫H, Sharma VK,斯利瓦斯塔瓦TP,泰勒TD,野口H, Mori H, Ogura Y,埃利希DS,伊藤K,高木涉T,淡比Y, Hayashi T,服部年宏M:一般比较宏基因组显示基因集富集在人类肠道微生物组。DNA研究》2007年,14:169 - 181。10.1093 / dnares / dsm018。
Tatusov RL:基因组的角度对蛋白质的家庭。科学。1997年,278:631 - 637。10.1126 / science.278.5338.631。
Tatusov RL,纳塔尔哒,Garkavtsev四世Tatusova助教,Shankavaram UT,饶B, B Kiryutin加尔佩林,Fedorova ND, Koonin EV:齿轮数据库:新发展系统分类的蛋白质从完整的基因组。核酸研究》2001年,29日:22。10.1093 / nar / 29.1.22。
Turroni F, Foroni E, Pizzetti P, V Giubellini, Ribbera, Merusi P, Cagnasso P, B Bizzarri de 'Angelis GL,沙纳罕F,杰费里•范•辛德伦D,文图拉M:探索bifidobacterial人口的多样性在人类肠道。:环境Microbiol。2009年,75:1534 - 1545。10.1128 / AEM.02216-08。
Pawitan Y,米歇尔•年代,科斯切尔尼,Gusnanto, plone(答:错误发现率、灵敏度和微阵列研究样本量。生物信息学。2005年,21日:3017 - 3024。10.1093 /生物信息学/ bti448。
铃木Y, Nei M:假阳性选择被ML-based方法:例子Sig1基因的硅藻这种weissflogii和税收人类t细胞嗜淋巴细胞的病毒的基因。另一个星球杂志。2004年,21日:914 - 921。10.1093 / molbev / msh098。
在生物信息学研究中Boulesteix l:过于乐观。生物信息学。2010年,26日:437 - 439。10.1093 /生物信息学/ btp648。
2020年愿景。大自然。2010年,463:26 - 32。
Hamady M,骑士R:人类微生物组项目:微生物群落分析的工具,技术,和挑战。基因组研究》2009年19:1141 - 1152。10.1101 / gr.085464.108。
伍力JC, Godzik,弗里德伯格:宏基因组的底漆。公共科学图书馆第一版杂志。2010年,6:e1000667 - 10.1371 / journal.pcbi.1000667。
里奇MD:利用先验知识和全基因组关联识别途径参与多发性硬化症。基因组医学。2009年,1:65 - 10.1186 / gm65。
Tintle N,兰提里F, Lebrec J,孙M,巴拉德D, Bickeboller H:先验信息纳入全基因组关联分析。麝猫论文。2009年,17 (1):33 s74 - 80。
林W-Y,李含水量:结合先验知识来促进发现在年龄相关性黄斑变性的全基因组关联研究。BMC Res笔记。2010年,3:26 - 10.1186/1756 - 0500 - 3 - 26所示。
里德J,骑士R:“罕见的生物圈”:一个现实。Nat方法。2009年,6:636 - 637。10.1038 / nmeth0909 - 636。
泰勒兆瓦,思楚普PJ, Dahllof我Kjelleberg年代,斯坦伯格PD:海洋sponge-associated细菌的宿主特异性,对海洋微生物多样性和潜在的影响。环境Microbiol。2003年,6:121 - 130。10.1046 / j.1462-2920.2003.00545.x。
Tamames J, Abellan JJ, Pignatelli M,卡马乔,莫亚:环保原核类群的分布。BMC Microbiol。2010年,10:85 - 10.1186/1471 - 2180 - 10 - 85。
Kassen R:实验进化的专家,多面手,多样性的维护。J另一个星球。2002年,15:173 - 190。10.1046 / j.1420-9101.2002.00377.x。
弗兰克DN,步伐NR,彼得森哒,戈登霁:宏基因组方法定义和炎症性肠病的发病机制。细胞微生物。2008年,3:417 - 427。10.1016 / j.chom.2008.05.001。
年轻的C, Sharma R, Handfield M, Mai V, Neu J:婴儿的风险坏死性小肠结肠炎的生物标志物:线索预防?。儿科研究》2009年,65:91 - 97 r。10.1203 / PDR.0b013e31819dba7d。
Asikainen年代,Doğan B,奥Z,贴纸BJ, Bodur,观察牙龈沟液内细胞激素Oscarsson J:指定物种在预测细菌多样性。PLoS ONE。2010年,5:e13589 - 10.1371 / journal.pone.0013589。
张黄D, L,法雷尔J,周H, Elashoff D,高K,贴纸B:唾液胰腺癌检测生物标记物。肿瘤防治杂志。2009年,27日:4630 -
Culligan EP,希尔C,看到理查德·道金斯:益生菌和胃肠道疾病:成功,问题和未来前景。肠道Pathog。2009年,1:19 - 10.1186/1757 - 4749 - 1 - 19所示。
Preidis GA, Versalovic J:针对人类微生物组用抗生素,益生菌和益生元:胃肠病学,进入宏基因组时代。胃肠病学。2009年,136:2015 - 2031。10.1053 / j.gastro.2009.01.072。
Borody TJ,沃伦EF花环年代,Surace R,清道夫O:使用粪便细菌疗法治疗溃疡性结肠炎。中国新药杂志。2003年,37:42-47。10.1097 / 00004836-200307000-00012。
简颂JK Khoruts A Dicksved J, Sadowsky MJ:成分的变化人类粪便微生物细菌疗法后复发艰难梭状芽胞杆菌相关的腹泻。中国新药杂志。2010年,44:354 - 360。
Manichanh C,里德J吉波特P, Varela E, Llopis M, Antolin M, Guigo R, R,骑士Guarner F:重塑肠道微生物与细菌移植和抗生素的摄入量。基因组研究》2010年,20:1411 - 1419。10.1101 / gr.107987.110。
马你D, Franzos:成功的治疗重型艰难梭状芽胞杆菌感染粪便细菌疗法。安实习生地中海。2008年,148:632 - 633。
Chang Y-w,林C-j:排名功能使用线性支持向量机。机器学习研究》2008,3:53 - 64。
嘉里蒂王Q,通用,Tiedje JM,科尔JR:朴素贝叶斯分类器核糖体rna序列的快速分配到新的细菌分类。:环境Microbiol。2007年,73:5261 - 5267。10.1128 / AEM.00062-07。
贝尔TC,佳詹,威滕IH:文本压缩。1990年,新世纪公司
高熔点数据分析和协调中心。(http://www.hmpdacc.org/tools_protocols/tools_protocols.php]
莫生物PowerSoil工具包。(http://www.mobio.com/]
休斯SM, Huber是的,莫里森HG, Sogin ML,韦尔奇DM:大规模并行DNA焦磷酸测序的精度和质量。基因组医学杂志。2007年,8:r143 - 10.1186 / gb - 2007 - 8 - 7 - r143。
Pruesse E, Quast C, Knittel K, Fuchs BM,路德维希W, Peplies J, Glockner FO:席尔瓦:一个全面的在线资源质量检查和对齐的核糖体RNA序列数据与ARB兼容。核酸研究》2007年,35:7188 - 7196。10.1093 / nar / gkm864。
城堡PD:微生物生态学研究的高通量DNA序列调整器。PloS ONE。2009年,4:e8230 - 10.1371 / journal.pone.0008230。
Gevers哈斯BJ, D,伯爵,Feldgarden M,病房DV, Giannoukos G, Ciulla D, Tabbaa D,汉兰达SK, Sodergren E,给我买fergie的。B DeSantis TZ,人类微生物组的财团,彼得摩根富林明,骑士R, Birren BW:嵌合16 s rRNA序列形成和检测在桑格和454 - pyrosequenced PCR扩增子。基因组研究》2011年,21日:494 - 504。10.1101 / gr.112730.110。
嘉里蒂通用,李若本TG,小科尔,哈里森SH, Euzeby J, Tindall奉:细菌和古菌的分类大纲。2007年,[http://www.taxonomicoutline.org/index.php/toba/article/viewFile/190/223]
顺序读取存档:SRP002012人类微生物组计划454年临床生产试点(PPS)。(http://trace.ncbi.nlm.nih.gov/Traces/sra/?study=SRP002012]
Hothorn TH, Hornik K, van De由马Zeileis答:实现一个类的排列测试:硬币包。J统计软件。2008年,28日:1。
维纳布尔斯WN,里普利BD:现代应用统计与美国2002年,施普林格,4
猎人JD: Matplotlib: 2 d图形环境。计算科学Eng。2007年,9:90 - 95。
确认
我们要感谢整个人类微生物组项目财团,包括四个测序中心(Broad研究所,华盛顿大学,贝勒医学院和J Craig Venter研究所),相关调查人员从许多额外的机构,和美国国立卫生研究院办公室主任路线图计划。这部分工作是支持由格兰特DE017106国家牙科和颅面研究所(JI)、国家卫生研究院的基金AI078942 (WSG)和布罗Wellcome基金(WSG),是由国立卫生研究院1 r01hg005969 CH。
作者信息
作者和联系
相应的作者
额外的信息
作者的贡献
NS和CH构思研究;NS和LM方法实现;NS:霁:LW: DG: WG和CH分析结果;NS:霁:LW: DG: WG和CH写手稿。所有作者阅读和批准了手稿的最终形式。
电子辅料
13059 _2011_2561_moesm1_esm.pdf
额外的文件1:补充图S6。受试的柱状图β-diversity(社区不同)不同粘膜(红色)和non-mucosal(绿色)网站。(PDF 65 KB)
13059 _2011_2561_moesm2_esm.pdf
额外的文件2:补充图S1。进化分枝图代表viromes之间的差异和微生物子系统框架。(PDF 2 MB)
13059 _2011_2561_moesm3_esm.pdf
额外的文件3:补充图S2。直方图的LDA对数的生物标志物发现LEfSe比较微生物和viromes子系统框架内。(PDF 384 KB)
13059 _2011_2561_moesm4_esm.pdf
额外的文件4:补充图S3。直方图的LDA对数的齿轮生物标志物发现LEfSe比较成人和婴儿肠道菌群。(PDF 184 KB)
13059 _2011_2561_moesm5_esm.pdf
额外的文件5:补充图S4。功能特性(齿轮)discrimantive成人和婴儿之间的比较微生物根据Metastats LEfSe但不是发现在判别特性与LDA得分高于3。如果我们考虑所有判别特性没有threhold LDA得分,LEfSe识别366齿轮,其中185不是Metastats判别。(PDF 230 KB)
13059 _2011_2561_moesm6_esm.pdf
额外的文件6:补充图S5。功能特性(齿轮)discrimantive成人和婴儿之间的比较微生物根据Metastats但不是LEfSe探测到。即使值和方差显示的差异区别的,总有一些微生物(至少两个)类之间的重叠。这是由于严格的α-value(0.01)为LEfSe千瓦测试,我们使用非参数统计数据(不同于Metastats)。不过,请注意,即使使用低α-value LEfSe检测生物标记物比metastats(366和192)。(PDF 289 KB)
13059 _2011_2561_moesm7_esm.pdf
额外的文件7:补充图S9。比较LEfSe和Metastats使用合成数据描述的图5材料和方法。LEfSe应用详细摘要;Metastats我们使用缺省设置(即α= 0.05和N排列= 1000),至于LEfSe和千瓦,我们禁用每个样本归一化特性是独立的。(a, b)Metastats的假阳性率高(平均5%)比LEfSe(平均低于0.5%)和低假阴性率。(c)当子类信息是有意义的(见图5数据集的表示),LEfSe执行大大优于Metastats假阳性和假阴性。总的来说,在这些合成数据,Metastats达到千瓦(图相比非常相似的结果5),他们两人可以利用额外的信息关于在课堂结构,从而实现业绩不佳而LEfSe当这样类型的信息是可用的。(PDF 376 KB)
13059 _2011_2561_moesm9_esm.pdf
额外的文件9:补充图S8。比较最高的特性基于svm的影响大小(Papillibacter左),最高LDA-based效应大小(双歧杆菌属在中心),放线菌门(右边)。从视觉分析,Bifidobacerium显示了一个更大的效果,这也是明显看着类意味着之间的比率,这表明LDA的更好的选择比SVM方法效果的评估。在手稿,详细的相关性双歧杆菌属已被实验验证。此外,大比分差由放线菌的SVM方法相比双歧杆菌属和Papillibacter是不一致的。(PDF 71 KB)
13059 _2011_2561_moesm10_esm.txt
额外的文件10:T-bet- / -×Rag2- / -- - - - - -Rag2- / -数据集。输入LEfSe文件分析的溃疡性结肠炎小鼠的表型。(TXT 83 KB)
作者提交的原始图像文件
权利和权限
关于这篇文章
引用这篇文章
Segata, N。,Izard, J., Waldron, L.et al。宏基因组生物标志物的发现和解释。基因组医学杂志12R60 (2011)。https://doi.org/10.1186/gb - 2011 - 12 - 6 - r60
收到了:
修改后的:
接受:
发表:
DOI:https://doi.org/10.1186/gb - 2011 - 12 - 6 - r60
关键字
- 线性判别分析
- 细菌性阴道炎
- 身体的网站
- 效果评估
- 核糖体数据库项目