摘要
背景
目前rna测序数据的标准化方法允许样本间比较以识别差异表达(DE)基因或样本内比较以发现和验证基因特征。大多数关于规范化方法优化的研究通常使用模拟数据来验证方法。我们描述了一种新的方法,GeTMM,它允许使用相同的规范化数据集进行样本间和样本内分析。我们使用来自263例结肠癌(无生物重复)的实际(即非模拟)RNA-seq数据,并使用相同的读取计数数据,将GeTMM与最常用的归一化方法(即TMM (edgeR使用)、RLE (DESeq2使用)和TPM)在分布、RNA质量影响、亚型分类、复发评分、DE基因召回率以及与RT-qPCR数据的相关性方面进行比较。
结果
我们观察到GeTMM和TPM在样本内比较方面有明显的优势,而GeTMM在样本间比较中的表现与TMM和RLE标准化数据相似。对于DE基因,归一化方法的召回率具有可比性,而GeTMM显示的DE基因假阳性数量最少。值得注意的是,我们在低RNA质量的样品中观察到有限的有害影响。
结论
我们表明,GeTMM在样本内比较方面优于现有方法,同时使用相同的规范化数据在样本间规范化方面执行等效方法。这些组合的特性增强了RNA-seq的通用性,也增强了与公共领域中许多基于阵列的基因表达数据的可比性。
背景
近年来,转录组的分析已经从使用微阵列转向使用可能更强大和信息量更大的cDNA大规模平行测序(RNA-seq) [1]。在RNA-seq中,序列读取与参考基因组对齐,并且与特征(如基因)对应的读取数是与所述特征的长度和丰度成正比的度量。在进行下游分析之前,必须执行标准化以纠正测序运行之间的差异(例如文库大小和相对丰度)。
当前的归一化方法允许样本间或样本内比较。当对样本间DE基因感兴趣(样本间比较)时,最常用的两种归一化方法是edgeR [2]和DESeq [3.,4]。与其他归一化算法(Total count、UpperQuartile、Median、Quantile,以及LimmaQN、limmaoom、CuffDiff和Bayseq使用的归一化算法相比,这两种方法的归一化算法(edgeR的修剪均值m值TMM和DESeq的相对对数表达式RLE)表现出一致的良好性能[5,6,7,8]。值得注意的是,TMM和RLE并没有纠正观察到的基因长度的读取计数,这在理论上与样本间比较无关。然而,这种方法不允许样本内比较,因为在相同水平表达时,较长的基因比较短的基因获得更多的读取计数。因此,样本在没有校正的情况下看起来高度相关,而实际上经过长度校正后相关性要低得多(参见附加文件)1),在极端情况下,可以根据基因长度而不是表达水平进行关联。这个问题扩展到基于相关性的方法,例如,一个样本的一组基因与另一个样本相关,就像在分层聚类中经常做的那样(相关性被用作相似性度量)。此外,基于已建立的特征基因面板与新样本(如结直肠癌的共识分子亚型(CMS))的相关性的分类器将产生错误的结果,而不纠正基因长度的基因表达水平。
包括基因长度校正的最常用的归一化方法是TPM (Transcripts Per kilobase Million) [9],其他方法如RPKM [1] / FPKM [10(Reads/Fragments Per Kilobase Per Million Reads分别被证明是不充分和有偏差的[5,6,11,12]。
理想情况下,规范化方法应该生成一个数据集,在该数据集上可以执行样本间和样本内分析。因此,我们引入了GeTMM(基因长度校正TMM),这是一种新的归一化方法,将基因长度校正与归一化过程TMM相结合,在edgeR中实现,允许在同一归一化数据集上进行样本间和样本内比较。我们使用了263例结肠癌患者原发肿瘤大队列的真实(即非模拟)RNA-seq数据,并使用我们的新方法GeTMM与TMM、RLE和TPM一起对这些数据进行规范化[6]。我们研究了归一化数据集在分布、RNA质量影响、亚型分类(即CMS分类)方面的几个特性[13],临床复发评分[14], DE基因的召回率以及与同一样本生成的RT-qPCR数据的相关性。本研究的主要目的是确定GeTMM在样本间分析方面是否与其他归一化方法相同,以及基因长度校正是否以及在多大程度上影响样本内分析。
方法
队列描述
MATCH研究是一项多中心观察队列研究,在荷兰鹿特丹地区的七家医院之一接受手术的263名结肠癌患者的新鲜冷冻肿瘤组织被使用。纳入标准和其他临床特征已被描述[15]。
RNA分离、cDNA合成、qPCR和RNA-seq
关于rna分离的详细描述已在前面描述过[16,17];简单地说,根据制造商的说明(Tel-Test Inc., USA),使用RNA- bee®从30 μm的切片中分离RNA。使用NucleoSpin RNA II组织试剂盒(machery - nagel GmbH & Co. KG,德国)去除和清理基因组DNA前后的RNA质量和数量,使用Nanodrop ND-1000 (Thermo Scientific, Wilmington, USA)和多芯片电泳系统(Shimadzu, Kyoto, Japan)进行评估。dna去除和清理后,使用多芯片电泳系统评估RNA完整性数(RIN)(附加文件)2评估安捷伦生物分析仪的RIN值与multi测量的质量之间的关系)。根据制造商说明书(Fermentas, St Leon-Rot, Germany),用RevertAid H - First Strand cDNA合成试剂盒从1 μg总RNA生成cDNA。RT-qPCR使用Mx3000P QPCR机(Agilent Technologies,荷兰),根据制造商说明使用ABgene Absolute Universal或Absolute SYBR Green与ROX PCR反应混合物(Thermo Scientific,美国)。用δ - δ Cq方法定量33个转录本水平的内含子跨越试验如前所述进行了评估[16,17],并在附加文件中进行了总结3..
对于RNA-seq,使用Ribo Zero (Illumina, USA)去除gDNA、清理和去除核糖体RNA后的总RNA 500 ng作为Illumina TruSeq链RNA-seq方案(配对端)的输入。没有使用生物重复。在Illumina HiSeq2500 (2x101bp, 177个样本)或NextSeq (2x76bp, 86个样本)仪器上对文库进行汇总和测序。池大小和每次运行的样本量是根据组织学检查估计的肿瘤细胞百分比来确定的[15]。我们使用STAR [18]算法(版本2.4.2a)在GRCh38参考基因组上对齐RNA-seq数据(设置在附加文件中)4)。为了获得每个基因的读取计数,使用了“quantMode GeneCounts”,其中只包括那些具有足够比对分数和唯一映射的读取。来自NextSeq机器的76 bp的读取长度足以准确定位到参考基因组,并且我们发现来自不同机器的数据没有偏差。
基因注释来源于GENCODE Release 23 (https://www.gencodegenes.org/)。的外显子特定计数CDK1和MKI67,每个基因的所有独特的哈瓦那外显子被提取并用于featurets [19可以设置-t exon, -O和-f。这些设置以及-p的缺失(用于配对端计数)确保了对每个外显子的重叠读取计数。这确保了外显子存在的所有证据都被计算在内。
RNA-seq数据的规范化
将所有样本的原始读取计数合并到单个读取计数矩阵中。该矩阵被用作每种不同归一化方法的输入。最常用的RNA-seq归一化方法是TMM,在edgeR中实现。2]和RLE,在DESeq2 [3.,4]。这两种方法都不采用任何基因长度归一化,因为它们的目的是识别样本之间的DE基因,因此假设基因长度在样本之间是恒定的。TPM方法增加了以前使用的RPKM(用于单端测序协议)或其成对端对应的FPKM。TPM使用一种简单的规范化方案,其中每个基因的原始读取计数除以其以kb为单位的长度(Reads per Kilobase, RPK), RPK的总和被认为是该样本的库大小。接下来,将文库的大小除以100万,并将其用作缩放因子来缩放每个基因的RPK值。因此,TPM确实对基因长度进行了校正,但缺乏复杂的样本间校正;它没有考虑到可能的少数高表达基因,因此构成了该样本的总文库大小的很大一部分。DESeq2和edgeR通过估算用于重新调整计数的校正因子来解决这个问题(参见[2,3.]查阅详情)。简而言之,edgeR采用M值的修剪平均值(TMM) [2],其中高表达基因和表达差异较大的基因被排除在外,因此使用基因子集的加权平均值来计算归一化因子。DESeq2使用的RLE也假设大多数基因不是DE;在这里,计算每个基因在一个样本中的读取数与该基因在所有样本中的几何平均值的比率。样本中所有基因比率的中位数用作校正因子。其中TMM (edgeR)估计应用于文库大小的校正因子,RLE (DESeq2)的校正因子应用于单个基因的读取计数。
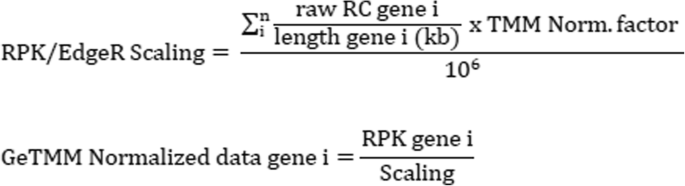
这种归一化的数据在样本之间具有更好的可比性,但仍然存在无法比较样本内基因表达水平的问题。为了获得同样适用于样本间和样本内分析的归一化数据集,提出了以下GeTMM方法:首先,计算样本中每个基因的RPK:原始读取计数/长度基因(kb)。在使用tmm归一化的edgeR中,通常库大小(总读计数;RC)通过估计的归一化因子进行校正,并缩放到每百万读取,但在GeTMM中,总RC被总RPK取代(图2)。1)。
在实践中,为了获得GeTMM规范化数据,需要从原始读取计数和基因长度(以kb为单位)中预先计算RPK值,并将这些值用作edgeR包的输入。参见附加文件4对于r中的一步一步的过程,使用gencode的注释来计算基因长度:将具有唯一的exon_id注释到同一gene_id的所有外显子的长度求和。DESeq2只允许整数作为输入,因此基因长度校正产生的分数被DESeq2拒绝输入。
edgeR和DESeq2作为r包提供(https://bioconductor.org/),随后使用R (v3.2.2)进行分析。为了获得规范化数据,使用原始读取计数矩阵(以制表符分隔的文本文件)作为输入。获取规范化数据的R命令列在附加文件中4.每种方法输出规范化的读取计数,这些计数经过log2转换(当读取计数为0时将基因设置为NA)。
CMS分类使用“CMSclassifier”软件包(https://github.com/Sage-Bionetworks/CMSclassifier),使用单样本预测参数。的Onco类型DX®(14按照RT-qPCR数据的描述进行复发评分,并使用RNA-seq归一化值作为算法的输入。总之,使用了7个基因的表达数据;砰,砰,砰(基质板),Mki67, myc, mybl2(细胞周期图)和GADD45B。未缩放的复发评分(Rsu)计算为(0.1263 x平均间质面板)- (0.3158 x平均细胞周期面板)+ (0.3406 x)GADD45B).复发评分(RS)计算为44.16 x (Rsu + 0.30)。信噪比(SNR)计算为(mean1 - mean2)/Sp,其中Sp为混合方差Vp的平方根。计算公式为Vp = [(n1 - 1) V1 + (n2 - 1)V2]/(n1 + n2 - 2),其中V1和V2是每个组的方差,n1和n2是样本组的大小。
统计数据
使用R (v3.2.2)进行统计检验,适当时使用非参数检验(Mann-Whitney U检验、Spearman秩相关)。为了识别DE基因,使用了edgeR和DESeq2包中包含的默认测试(对DESeq2进行Wald测试,对edgeR进行负二项分布的精确测试)。对于edgeR,根据文档建议,使用0.4的常见分散值。此外,对于edgeR和DESeq2,以及RT-qPCR, TPM和GeTMM,使用学生t检验。计算均方根误差(RMSE)时,使用标准化数据(z归一化,从样本中观察到的表达值中减去基因的平均表达值,并将其除以基因表达值的标准差)。正文中说明了统计检验。p-值是双侧的,p值和fdr (Benjamini-Hochberg,必要时)在低于0.05时被认为是显著的。
结果
我们使用263名结肠癌患者的原发肿瘤组织来生成RNA-seq数据。没有生物或技术上的复制。我们将这些数据与人类参考基因组(GRCh38)进行比对,并生成每个基因的读取计数。该读取计数矩阵用于几个规范化过程:TMM(由edgeR实现)[2]、RLE(由DESeq version 2实现)[3.]和TPM,此外还有一种新提出的结合edgeR - GeTMM使用的归一化的基因长度校正方法。为了验证结果,用于生成序列文库的相同RNA也用于33个基因的RT-qPCR分析(见附加文件)3.详情)。我们的研究不是为了确定与RT-qPCR数据相容性最高的方法,而是为了比较GeTMM在样本间和样本内分析中的性能与其他归一化方法的性能。
RNA-seq数据分布
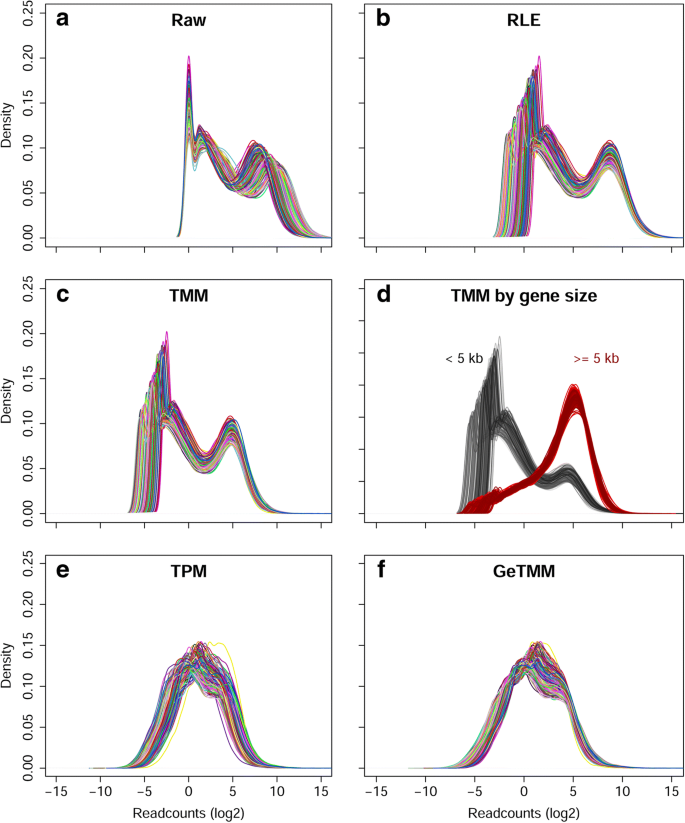
样本的库大小(即映射读数)在580万到3780万之间(平均1600万,中位数1420万)。生成密度图以获得读取计数分布的概述(图2)。2)。图2a显示了原始读取计数(未归一化,以log2为尺度),它清楚地显示了在初始峰值为0之后的双峰分布,峰值为1.1~ 1.4 log2-read计数,峰值为7~ 10 log2-read计数。通过DESeq2和edgeR分别对RLE和TMM进行归一化后,可以看到类似的双峰分布(图2)。2 b, c),两者都不能校正基因长度。按基因< 5kb和> = 5kb拆分TMM归一化数据(图2)。二维)表明双峰性主要与基因长度有关;正如预期的那样,较长的基因通常具有较高的读取数。采用基因长度校正的方法- TPM和GeTMM -都显示出更接近高斯分布(图2)。2 e, f)。
与RT-qPCR生成数据的比较:样本间分析
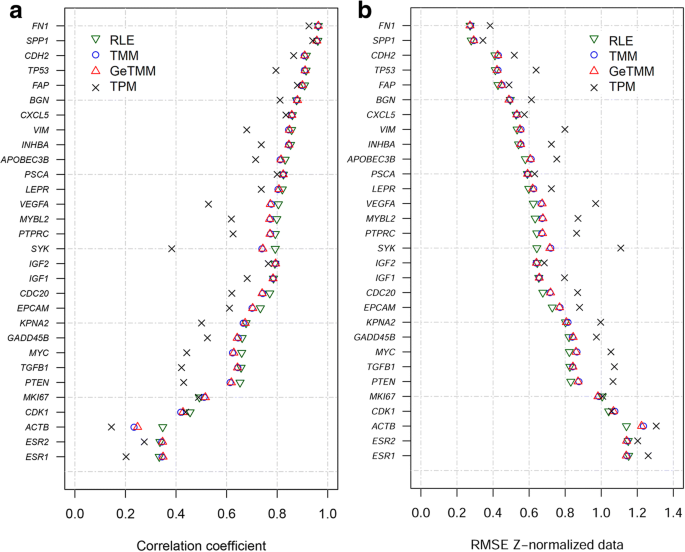
为了评估不同归一化方法对下游分析的影响,我们测量了33个基因(其中3个内参基因-)的表达水平hmb,HPRT1和真沸点),在与测序相同的RNA分离物中使用RT-qPCR。RT-qPCR数据使用内参基因归一化,并作为金标准进行比较。为了评估不同归一化方法对样本间分析的影响,我们将30个基因的归一化RNA-seq数据与所有样本的RT-qPCR水平相关联(图2)。3,附加文件5和附加文件6获取详细示例)。总体而言,GeTMM的相关系数与RLE和TMM归一化数据的相关系数非常相似,并且高于TPM的相关系数(图2)。3)。对于大多数基因,RLE的绝对相关系数最高,但与GeTMM的平均和中位数差异在个体系数上差异很小(分别为0.014和0.008)。此外,RLE、TMM和GeTMM归一化数据之间无显著差异(Mann-Whitney检验,见附加文件)7),而TPM的系数则明显低于其他方法(p= 0.02,p= 0.04和pRLE、TMM、GeTMM分别= 0.03)。对这些数据进行斯皮尔曼秩相关分析(以确定可能的非正态分布表达数据的影响)显示了相同的结果(附加文件)8)。并计算各方法与RT-qPCR数据的均方根误差(RMSE);为了做到这一点,我们首先使用z归一化对数据进行标准化,这样每个基因的数据的均值和标准差分别约为0和1。如果没有z归一化,对RMSE的有意义的解释将被RNA-seq归一化方法的表达范围差异所掩盖。RMSE值(图2)3 b), TMM和RLE同样具有可比性,而TPM的误差普遍较高。
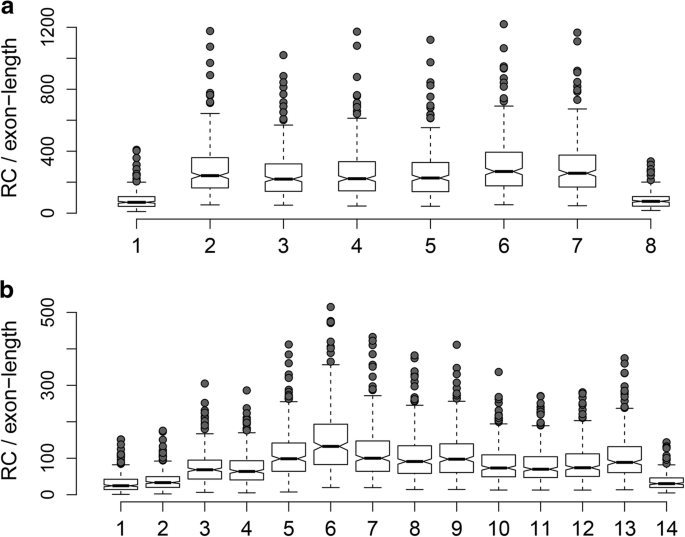
本部分研究的目的不是评价RT-qPCR数据获得的相关系数,而是将RT-qPCR数据作为基准,以便相互比较RNA-seq归一化程序。尽管如此,我们还是进一步研究了这五种表现出抗抑郁能力的基因R< 0.6;MKI67,CDK1,ACTB, ESR1和ESR2.根据RNA-seq数据,后两个基因的相关性较差可能是由于这些基因的表达量非常低(中位读取计数均为22)ESR1和ESR2),表明这些基因的测序深度不够。ACTB是30个基因中表达量最高的基因,在5种方法中有4种方差最小(RT-qPCR、RLE、TMM和GeTMM分别为0.25、0.13、0.16和0.16),这可能是相关性较低的原因。为CDK1和MKI67,我们重新分析了所有263个样本,以获得每个外显子的reads。的外显子1的表达较低CDK1,这可能解释了RT-qPCR与RNA-seq数据相关性较差的原因,因为RT-qPCR产物跨越了外显子1和2(图2)。4)。类似的分析MKI67没有显示出同样的效果;这里的RT-qPCR检测跨越了外显子10至11,两者的表达水平与整体基因表达水平相似(图2)。4 b)。所以除了转录本XM_006717864,这是唯一截断的转录本MKI67没有被RT-qPCR检测覆盖,主要存在于我们的样本队列中,我们没有发现这种低相关性的明显解释。
与RT-qPCR生成数据的比较:样本内分析
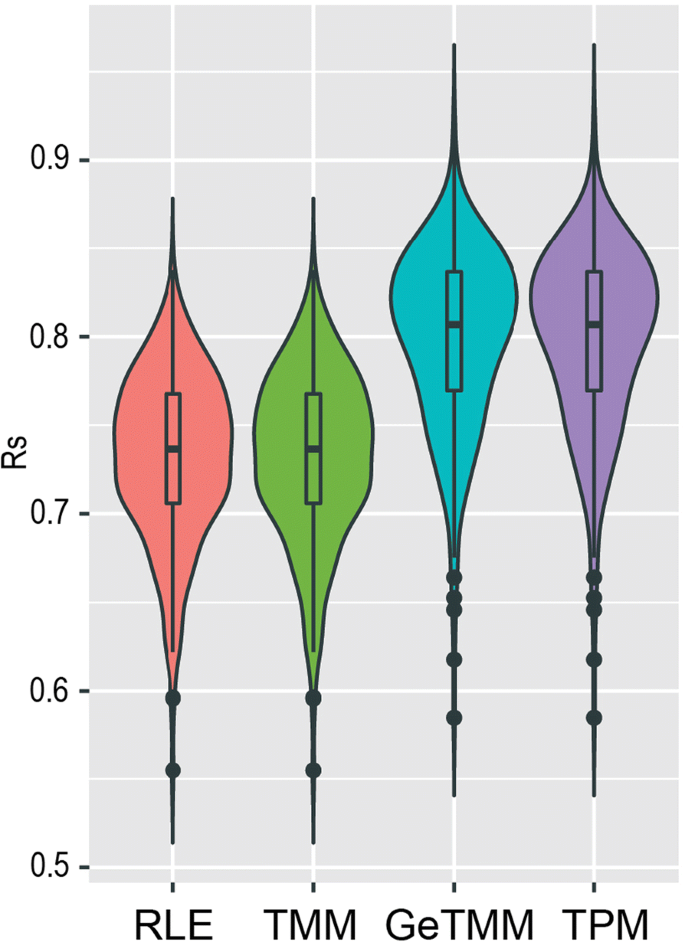
之前(20.],在MicroArray质量控制(MAQC)和Sequence Quality Control SEQC工作中,将RNA-seq归一化方法与RT-qPCR数据进行比较[21],使用另一种设置;通过RT-qPCR在单个样本中测量了996个基因,这些基因与同一样本的RNA-seq测量的基因表达水平相关(Spearman秩)。为了模拟SEQC结果,我们使用30个基因的RT-qPCR数据重复分析,并计算每个样本RT-qPCR与不同RNA-seq归一化方法之间的Spearman秩相关系数,每种方法得到263个相关系数(图2)。5)。GeTMM和TPM(包含基因长度校正的方法)与RT-qPCR数据的相关性均高于RLE和tmm归一化数据(Mann-Whitney)p< 0.0001)。263例中有262例的GeTMM具有较高的相关系数。
GeTMM的性能不受RNA质量差的影响
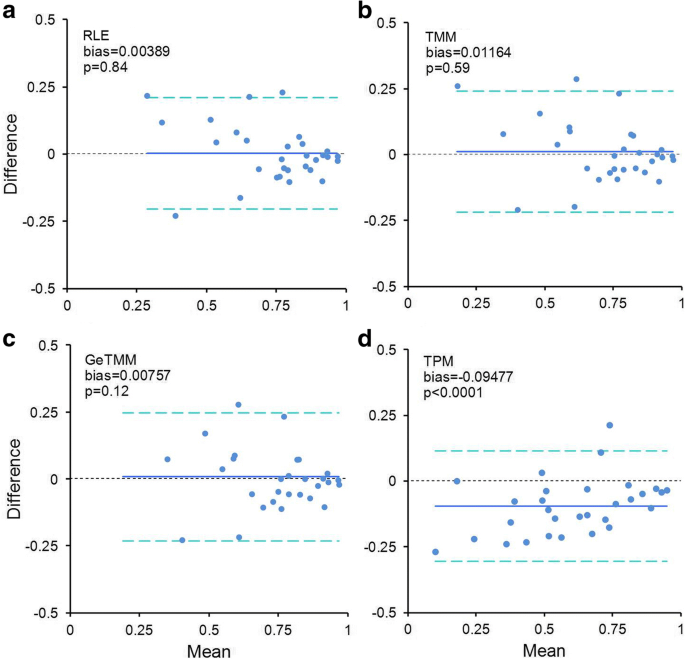
接下来,我们用RT-qPCR数据对清理后RNA完整性(RIN)值< 7(中位数RIN 5.3)的76个样本重复样本间相关性分析,并将其与具有最高RIN值(RIN bbb9,中位数RIN 9.5)的76个样本进行比较。低RIN组的中位数库大小略低于高RIN组的652万,为558万(Mann-Whitney)p= 0.02,参见附加文件9A)。然而,使用所有表达基因的主成分分析显示,无论采用何种归一化方法,都没有分离低/高RIN组(附加文件)9抵扣)。接下来,我们将低RIN组和高RIN组各归一化方法的RT-qPCR数据与RNA-seq数据分别进行关联,比较两组之间的相关系数。数字6-d显示了四种方法的Bland-Altman差异图,其中平均偏差和p与所有样本中RNA-seq和RT-qPCR的样本间比较类似,GeTMM的结果与TMM和RLE归一化数据相似,即低RIN组和高RIN组的相关系数相似。与低RIN组相比,使用TPM进行标准化确实导致高RIN组的相关系数显著降低(偏差= - 0.09477,p < 0.0001),再次表明与TPM相比,GeTMM具有优势。
GeTMM最接近RT-qPCR的差异表达分析结果
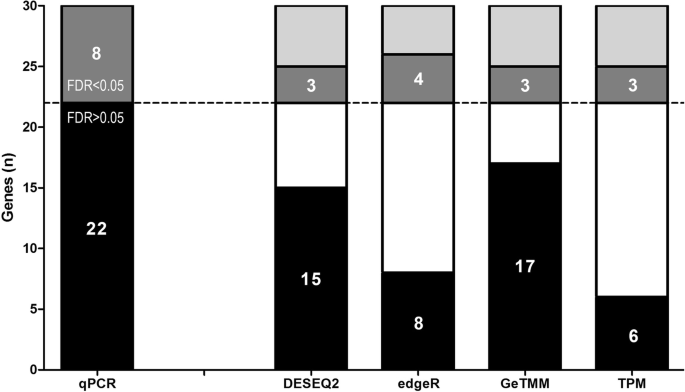
不同归一化方法与RT-qPCR数据的相关性已经表明,GeTMM的表现与TMM和RLE相当,但优于TPM。为了进一步研究在生物学相关背景下不同归一化方法对样本间分析的影响,由于已知左半结肠和右半结肠的肿瘤在生物学上是不同的,因此我们检查了左侧和右侧结肠肿瘤中的基因的差异表达。总之,右侧肿瘤经常出现超甲基化、超突变、微卫星不稳定和BRAF而左侧肿瘤通常是微卫星稳定的,并且经常携带APC和喀斯特突变(22]。这一特征大致将我们的队列分成两半(48%为左侧,52%为右侧)。我们对RT-qPCR数据集中的所有30个基因进行标准t检验,经多次检验校正(Benjamini-Hochberg)后,8个基因的FDR < 0.05:Mybl2, myc, epcam, syk, apobec3b, spp1, cdk1和IGF1.接下来,为了检验RNA-seq归一化方法对相关生物变异的去除/压缩量是否存在差异,我们计算了这8个基因的信噪比(SNR)。同样,GeTMM的表现与TMM和RLE规范化数据相似,显示出非常相似的信噪比,但优于TPM(参见附加文件)10)。到目前为止,我们使用的是DESeq2和edgeR归一化数据(分别为RLE和TMM),然而,这些方法都是用于DE基因的归一化和鉴定。每个都使用了一个统计测试,该测试是为在各自的包中使用而设计的(分别为DESeq2和edgeR进行Wald测试和精确测试)。因此,为了比较GeTMM与DESeq2和edgeR在识别DE基因方面的性能,我们分别在各自的数据集上运行edgeR和DESeq2实施的统计检验,而对于TPM和GeTMM数据,我们对30个基因使用Student 's t检验。数字7显示了FDR调整后的比较结果p-值归一化方法。根据RT-qPCR数据,在22个非DE基因中,与edgeR(14/22)、DESeq2(7/22)和TPM(16/22)相比,GeTMM的“假阳性”数量最少(5/22)。所有方法的召回率相似(edgeR的召回率为4 / 8,其他方法的召回率为3 / 8)。当用t检验分析TMM (edgeR)和RLE (DESeq2)归一化数据时,edgeR的召回率降至3个基因,而DESeq2的召回率为4个基因。edgeR和DESeq2都将5个基因称为“假阳性”(与GeTMM称为显著的5个基因相同)。
基因长度校正使TMM在Oncotype DX®复发评分中获益
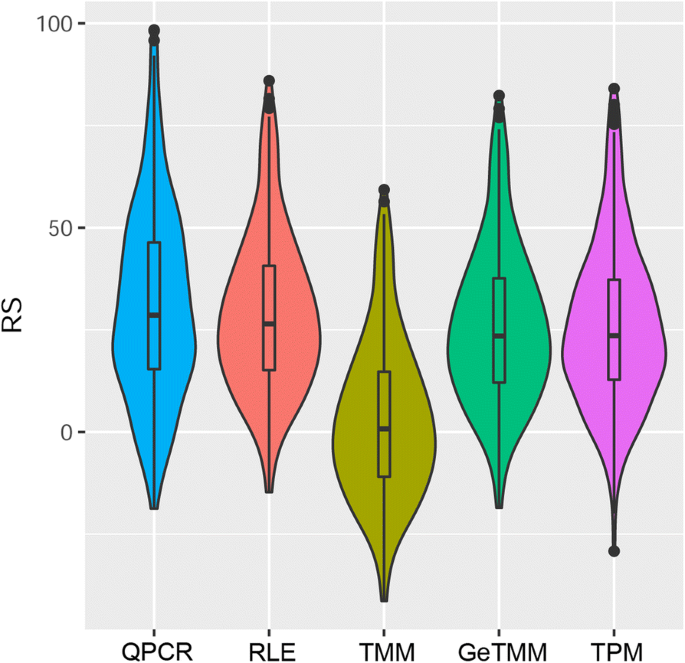
一种常用的评估结肠癌复发风险的工具是Onco的复发评分(RS)算法类型DX®(14],该公司使用7种癌症基因小组。根据RT-qPCR数据和RNA-seq归一化数据集计算所有样本的RS(图2)。8)。RT-qPCR生成的分数分布与使用RNA-seq生成的分数非常相似,除了TMM衍生的RS。整体较低的分数将影响RS评估,因为原始RS被缩放,因此负分数将被设置为零。使用TMM, 41%的患者(n= 109)会得到这个分数。显然,在边缘归一化的基础上使用基因长度校正的GeTMM改善了RS分数的范围和分布。
基因长度校正影响CMS预测
最后,使用不同归一化方法的数据确定每个样本的CMS分类[13]。在这个分类中,预测了五个可能的组:CMS1-4和混合/不确定。分类的类型是基于每个亚型特定于单个样本的基因特征的相关性,使其成为样本内类型分析。在预测的CMS组中,RLE和TMM归一化数据(均未进行基因长度校正)、TPM和GeTMM(均进行基因长度校正)完全一致。然而,基因长度校正对CMS组的预测有相当大的影响:当比较TMM和GeTMM时,在不同的组中预测了40个样本(15.2%)1)。
讨论
目前的研究表明,在样本间分析中,GeTMM与两种常用的、在几个RNA-seq归一化方面表现最好的方法——RLE(由DESeq2使用)和TMM(由edgeR使用,两者都不使用基因长度校正)——相当[6,7,8],而在样本内比较中优于这些方法。因此,GeTMM生成一个直接适用于多个端点的规范化数据集。在263例原发性结肠肿瘤的真实(即非模拟)数据的大队列中,评估不同方法对基因表达数据分布、不同RNA质量样本、亚型分类、复发评分、DE基因召回率、RMSE分析以及与RT-qPCR数据的相关性的影响。重要的是,目前的研究主要集中在RNA-Seq数据在样本之间和样本内部差异表达分析中的应用,因此没有涵盖融合事件检测、变异分析和基因同工型等其他应用[23]。对于后者,本研究中使用的归一化方法(包括GeTMM)没有被开发出来区分可能的亚型,这需要使用更复杂的模型和不同的统计量在转录水平上估计表达[10,24,25]。因此,所研究的归一化方法可能不完全适合这种转录水平分析。
当意识到一些常用的标准分析容易受到基因长度诱导偏差的影响时,基因长度校正对下游分析的影响比最初看起来更重要。除了引言中所述的理论例子外,另一个例子是例如在乳腺癌中,其中AIMS [26方法的开发,以获得真正独立的单样本分类器,以稳健地调用分子亚型。在此,在每个样本中评估亚型特异性基因;例如,当GRB7(532 bp的转录本)的表达量高于BCL2(一个239 bp的转录本),它增加了HER2亚型的证据[26]。如果不校正基因长度,这种预测方法将无法在RNA-seq数据上正常工作GRB7读取计数将比BCL2当两个基因的表达水平相等时,Read计数。在本研究中评估这些样本内类型分析时,当将同一样本内通过不同方法测量的一组基因进行关联时,与TMM (edgeR)和RLE (DESeq2)归一化的数据相比,GeTMM和TPM产生了明显更好的结果。以前也进行过类似的分析[20.使用MicroArray Quality Control (MAQC)项目提供的数据,其中RT-qPCR测量了更多的基因,但只使用了两个样本。在我们的研究中,我们使用了263个样本,从而更好地捕获了基因表达水平的生物学变异。在临床适用性方面,本研究表明基因长度校正影响结直肠癌亚型(CMS)的预测[13]。考虑到CMS分类器的方法,其中单个样本的基因表达数据与4个CMS组中的每一个特定的一组基因的质心相关,使用包括基因长度校正的规范化更有意义,以避免低估或高估样本内基因的真实表达水平。值得注意的是,我们并没有声称预测了真正的CMS分类,但假设GeTMM分类反映了更可靠的预测,使用不含基因长度校正的方法,23个样本将从CMS组变为混合/不确定组,1个样本将从CMS2变为CMS4。在计算复发评分(Onco)时类型DX®)edgeR的总体分布要低得多,几乎一半的患者评分低于零分。通过包括基因长度校正(从而产生GeTMM)来弥补这一点,导致得分非常可比,并且与RT-qPCR产生的分数在相同的范围内。这说明了使用像GeTMM这样的规范化方法的重要性,它产生的数据集既适合样本间分析,也适合样本内分析。
几个指标被用来评估标准化方法,总结在表中2.一般来说,TPM不足以纠正样本之间的差异。这与先前报道的使用RPKM和FPKM归一化的结果相呼应[5,6,11,12],可以合理地得出结论,必须放弃仅通过文库大小归一化作为检测样本间DE基因的可行方法。RLE和TMM归一化数据在RT-qPCR的分布、相关性和RMSE以及对RNA质量的敏感性方面仅略有差异,而在CMS分类方面则完全没有差异。然而,edgeR采用的统计检验在识别DE基因方面似乎过于乐观,而DESeq2的统计检验则更为保守,这一差异也被其他人观察到[8]。考虑到RLE和TMM归一化后的数据非常相似,报告DE基因的差异更可能是两种方法采用的统计检验的差异,而不是归一化本身的差异。通过对所有归一化方法使用t检验证实了这一点,显示出非常可比的结果;因此,在鉴定DE基因的样本间分析中,GeTMM的表现与edgeR和DESeq相似。
使用低或高RIN值样本子集的分析显示,与RT-qPCR生成的数据的相关性差异很小。似乎具有低RIN值的样品可能产生适合表达分析的测序数据。尽管如此,这个结论是从单一的相关分析中得出的,并且可能非常特定于所使用的整个方案(RNA分离,文库准备等),因此可能不适用于所有的研究和方案。尽管如此,先验地忽略具有低RIN值的样品进行测序可能会被证明是浪费的,尽管对生成的测序数据执行稳健的QC以发现失败的样品是谨慎的。
最后,本研究以RT-qPCR为标准,可以对RNA-seq归一化方法进行比较。RT-qPCR以其精确和可重复的测量而闻名,与通常的序列数据覆盖相比,可能具有更大的动态范围。缺点是RT-qPCR只测量基因的一小部分,可能会错过或受到剪接变异的影响,并且可能受到引物区域snp的影响。在这方面,RNA-seq生成的数据可能更接近于一个基因实际表达水平的标志。在未来,RNA-seq可能会取代RT-qPCR成为表达数据的金标准,前提是使用一种有充分依据的归一化方法。
结论
这项研究表明,GeTMM产生了一个通用的标准化RNA-seq数据集,适用于样本间和样本内的比较。GeTMM的这一质量将进一步增强RNA-seq作为探索和比较基因表达谱的可靠方法的能力,因此在当前数据共享努力的时代可能会变得越来越有趣。
缩写
- CMS:
-
共识分子亚型
- 德:
-
差异表达
- 罗斯福:
-
错误发现率
- FPKM:
-
Fragments Per Kilobase Per Million reads
- GeTMM:
-
基因长度校正TMM
- MAQC:
-
芯片质量控制
- RIN:
-
RNA完整性编号
- RLE:
-
相对日志表达式
- RPK:
-
每千碱基读取数
- RPKM:
-
Reads Per Kilobase Per Million Reads
- 拉尔夫-舒马赫:
-
复发评分
- RT-qPCR:
-
逆转录-定量聚合酶链反应
- SEQC:
-
序列质量控制
- TMM:
-
m值的均值
- TPM:
-
每百万份成绩单
参考文献
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B.基于RNA-Seq的哺乳动物转录组定位与定量。地理学报。2008;5:621-8。
Robinson MD, Oshlack A.用于RNA-seq数据差异表达分析的标度归一化方法。中国生物工程学报。2010;11:R25。
Anders S, Huber W.序列计数数据的差异表达分析。中国生物医学工程学报,2010;22(1):491 - 491。
Love MI, Huber W, Anders S.用DESeq2调节RNA-seq数据的折叠变化和分散估计。基因组生物学,2014;15:550。
Bullard JH, Purdom E, Hansen KD, Dudoit S. mRNA-Seq实验归一化和差异表达统计方法的评价。中国生物医学工程学报。2010;11(4):591。
马丽丽,李建军,李建军,李建军,李建军,李建军,李建军,李建军,等。Illumina高通量RNA测序数据分析归一化方法的综合评价。生物通报,2013;14:7 71 - 83。
Rapaport F, Khanin R, Liang Y, Pirun M, Krek A, Zumbo P, Mason CE, Socci ND, Betel D. RNA-seq数据差异基因表达分析方法的综合评价。中国生物医学工程学报,2013;19(4):591 - 591。
Soneson C, Delorenzi M. RNA-seq数据差异表达分析方法的比较。生物信息学报。2013;14:91。
李斌,Ruotti V, Stewart RM, Thomson JA, Dewey CN。基于读图定位不确定性的RNA-Seq基因表达估计。生物信息学。2010;26:493 - 500。
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, Salzberg SL, Wold BJ, Pachter L. RNA-Seq转录本装配和定量揭示细胞分化过程中无注释转录本和同型异构体转换。生物工程学报。2010;28:511 - 511。
Oshlack A, Wakefield MJ。RNA-seq数据中的转录物长度偏差混淆了系统生物学。生物指南。2009;4:14。
Wagner GP, Kin K, Lynch VJ。使用RNA-seq数据测量mRNA丰度:RPKM测量在样本之间不一致。生物科学学报,2012;31(1):1 - 5。
王晓东,王晓东,王晓东,de Reynies A, Schlicker A, Soneson C, Marisa L, Roepman P, Nyamundanda G, Angelino P,等。结直肠癌的一致分子亚型。中华医学杂志。2015;21:1350-6。
张建军,张建军,张建军,等。肿瘤生物学在结肠癌诊断中的应用。中华医学杂志,2010;10:691。
Kloosterman WP, Coebergh van den Braak RRJ, Pieterse M, van Roosmalen MJ, Sieuwerts AM, Stangl C, Brunekreef R, Lalmahomed ZS, Ooft S, van Galen A,等。原发性结肠癌致癌基因融合的系统分析。巨蟹座,2017;77:3814-22。
Sieuwerts AM, Lyng MB, Meijer-van Gelder ME, de Weerd V, Sweep FC, Foekens JA, Span PN, Martens JW, Ditzel HJ。辅助性他莫昔芬获益基因标记预测晚期接受他莫昔芬治疗的激素初始雌激素受体阳性乳腺癌患者预后的能力评估。中国生物医学工程学报,2014;8(1):444 - 444。
Sieuwerts AM, Meijer-van Gelder ME, Timmermans M, Trapman AM, Garcia RR, Arnold M, Goedheer AJ, Portengen H, Klijn JG, Foekens JA。雌激素受体不同的ADAM-9和ADAM-11如何预测复发性乳腺癌患者对他莫昔芬治疗的反应:一项回顾性研究中华肿瘤杂志2005;11:731 - 721。
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,等。超快速通用rna序列比对技术。生物信息学。2013;29:15-21。
廖勇,史伟。featurets:一种高效的通用程序,用于分配序列读取基因组特征。生物信息学。2014;30:923-30。
李鹏,朴宇,沈海生,刘洪洪。Illumina高通量RNA-Seq数据差异分析归一化方法的比较。生物信息学报。2015;16:347。
财团SM-I。测序质量控制联盟对RNA-seq准确性、可重复性和信息含量的综合评估。生物工程学报,2014;32(3):903 - 14。
Muller MF, Ibrahim AE, Arends MJ。结直肠癌的分子病理分型。中文信息学报,2016;469:125-34。
王志强,王志强,王志强。RNA-Seq:转录组学的革命性工具。Nat Rev Genet. 2009; 10:57-63。
李波,杜威,CN。RSEM:从有或没有参考基因组的RNA-Seq数据中准确定量转录本。生物医学工程学报。2011;12:323。
Mehta S, Tsai P, Lasham A, Campbell H, Reddel R, Braithwaite A, Print C. TP53 RNA剪接的研究说明了RNA-seq方法的缺陷。巨蟹座学报,2016;76:751 - 759。
张丽娟,张丽娟。乳腺癌固有分子亚型的绝对分配。中华癌症杂志,2015;37:357。
致谢
作者要感谢Markus J. van Roosmalen, Mark Pieterse和Christina Stangl在肿瘤组织测序方面的工作。RNA-seq在乌得勒支测序设备进行。
研究小组:Peter Paul LO Coene, Jan Willem T Dekker, David DE Zimmerman, Geert WM Tetteroo, Wouter J Vles和Wietske W Vrijland, Rolf Torenbeek, Mike Kliffen, JH Carel Meijer和Anneke A vd Wurff。
资金
资助号:本工作由荷兰癌症协会(KWF)支持[资助号:UU 2012-5710和UVA 2013-6331];NutsOhra[批准号0903-011];荷兰消化基金会[资助号FP13-20];由荷兰科学研究组织(NWO)资助的荷兰癌症基因组学(CGC.nl);个人ERC高级资助[ERC- 20120adg -322737]和Daniel den Hoed基金会。
数据和材料的可用性
支持本文结论的数据集可通过欧洲基因组表型档案获得,登录号为EGAS00001002197 (https://ega-archive.org/)。
作者信息
作者及隶属关系
财团
贡献
MS, HvdW, JvR和WK生成,分析和解释RNA-seq数据。AG, VdW, MvdV-D和SB处理所有组织样品并分离RNA。ZL和RC收集组织样本和临床资料。JF, JIJ, JM和AS监督研究,并对稿件进行了严格的修改。MS, RCvdB和SW是撰写稿件的主要贡献者。所有作者均已阅读并批准最终稿。
相应的作者
道德声明
伦理批准并同意参与
所有患者对临床数据和肿瘤组织的收集和使用均给予书面知情同意(机构审查委员会Erasmus MC大学医学中心;mec - 2007 - 088)。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
附加文件
附加文件1:
基因长度校正对相关性的影响。模拟2个样品中10个基因的表达数据。校正基因长度后,基于reads计数的相关性显示出不同的结果。RPK表示每千基读数。(PDF 189kb)
附加文件2:
相关RIN生物分析仪与multi。将生物分析仪(Agilent)测量的RIN值与60例训练集(a)中多纳(MultiNA)测量的28S/总浓度进行比较。结果趋势线在73例独立队列(B)中得到验证。(PDF 199kb)
附加文件3:
RT-qPCR检测的详细信息。(xlsx14kb)
附加文件4:
详细介绍获取规范化数据的STAR算法和R命令的设置。(DOCX 13kb)
附加文件5:
各方法的相关系数与RT-qPCR的比较。(xlsx10kb)
附加文件6:
的表达PSCA以及几种RNA-Seq归一化方法的比较。(TIFF 9492 kb)
附加文件7:
用方法比较相关系数。箱线图显示了30个基因的相关系数,将4种方法与RT-qPCR生成的数据进行比较。P-数值来源于曼-惠特尼检验。(PDF 3492kb)
附加文件8:
Spearman与30个基因RT-qPCR数据的相关性。30个基因的相关系数(x轴)比较RNA-seq归一化方法与RT-qPCR生成的数据。(PDF 618kb)
附加文件9:
库大小和PCA图由RIN。A表示低RIN值(RIN < 7)或高RIN值(> = 9)的样本中的库大小(log10)。B-E为PCA图,通过归一化方法,用低RIN(红色)或高RIN(蓝色)的样品着色。(PDF 2842kb)
附加文件10:
信噪比。(xlsx9kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
Smid, M, Coebergh van den Braak, r.r.j., van de Werken, H.J.G.et al。基因长度校正的m值裁剪平均值(GeTMM)处理RNA-seq数据在样本间分析中表现相似,同时改善了样本内比较。BMC生物信息学19, 236(2018)。https://doi.org/10.1186/s12859-018-2246-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12859-018-2246-7
关键字
- RNA序列
- 归一化方法
- GeTMM
- 刨边机
- TPM
- DESeq2
- 结肠直肠癌