摘要
背景
近年来,由于广泛采用16S rRNA基因测序和宏基因组学等非培养分析技术,微生物群落的研究发生了革命性的变化。这些基于序列的方法的一个潜在混淆因素是DNA提取试剂盒和其他实验室试剂中存在污染。
结果
在这项研究中,我们证明了在常用的DNA提取试剂盒和其他实验室试剂中,污染DNA是普遍存在的,不同试剂盒和试剂盒批次之间的成分差异很大,并且这种污染严重影响从含有低微生物量的样品中获得的结果。污染既影响基于pcr的16S rRNA基因调查,也影响鸟枪宏基因组学。我们提供了一个广泛的潜在污染属的清单,以及如何减轻污染的影响的指导方针。
结论
这些结果表明,在应用基于序列的技术研究低生物量环境中存在的微生物群时应谨慎。强烈建议阴性对照样品同时测序。
背景
微生物群落的培养无关研究正在彻底改变我们对微生物学的理解,并揭示了微生物、动物和植物之间微妙的相互作用。两种广泛使用的技术是pcr扩增标记基因(如16S rRNA)的深度序列测量,或全基因组霰弹枪宏基因组学,其中对整个群落DNA进行测序集体.虽然这两种方法都很强大,但它们有重要的技术警告和限制,这可能会扭曲在序列数据集中观察到的分类分布和频率。这些限制在文献中已得到充分报道,包括与样本收集、样本存储和保存、DNA提取、扩增引物、测序技术、读取长度和深度以及生物信息学分析技术相关的选择[1]、[2].
另一个相关的问题是在样品制备过程中引入了受污染的微生物DNA。DNA污染的可能来源包括分子生物学级别的水[3.] - [9], PCR试剂[10] - [15]和DNA提取试剂盒本身[16].与水和土壤相关的细菌属匹配的污染序列包括不动杆菌、碱性菌、芽孢杆菌、缓生根瘤菌、草基菌、军团菌、雷氏菌、中根瘤菌、甲基菌、微菌、新鞘菌、假单胞菌、拉氏菌、鞘单胞菌、窄养单胞菌而且、黄以前曾报道过[3.] - [15]、[17]、[18].对于研究含有低微生物量样本的研究人员来说,受污染DNA的存在是一个特别的挑战。在这些情况下,少量的起始物质可能会被污染的DNA有效地淹没,并产生误导性的结果。
尽管文献中报道了这种污染DNA的存在,通常与基于pcr的研究有关,但尚未报道其对高通量16S rRNA基因分析和鸟枪宏基因组学研究的可能影响。在我们的实验室中,我们通常对阴性对照进行测序,包括“空白”DNA提取和随后的PCR扩增。尽管在DNA提取步骤中没有添加样品模板,这些阴性对照样品通常会产生一系列污染细菌种类(见表2)1),在同一批DNA提取试剂盒同时处理的人源样本中也经常可见。在低生物量样本(如血液或肺)中,污染序列的存在比在高生物量样本(如粪便)中更大,这表明存在一个关键的临界点,即污染DNA在序列库中占主导地位。
许多最近出版的刊物[19] - [37]描述重要或核心微生物群成员,通常是生物学上意想不到的成员,与先前描述的污染物属重叠。在此激励下,在我们自己实验室处理低投入DNA样本时,通过阴性对照样本的结果,我们调查了污染对微生物群研究的影响,并探索了限制这种污染影响的方法。在这项研究中,我们确定了常用DNA提取试剂中存在的污染物的范围,并证明了它们对微生物区系研究的重大影响。
结果
纯邦戈里沙门氏菌培养物的S rRNA基因测序
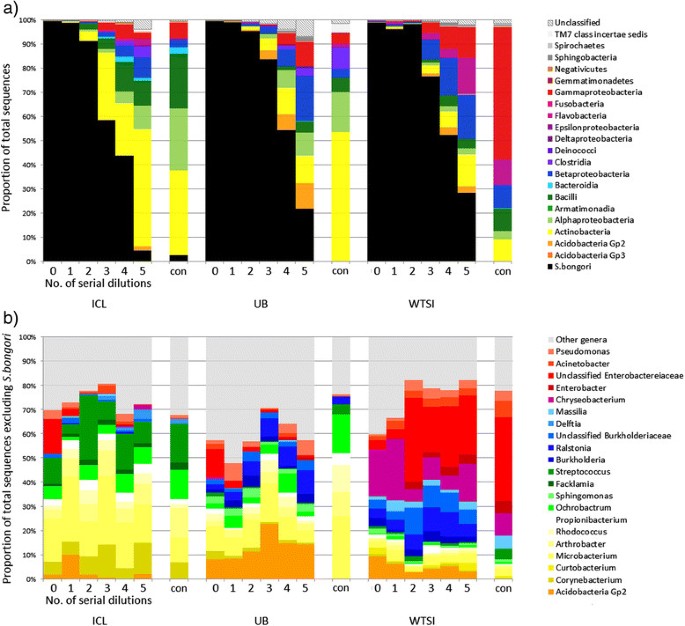
为了证明污染DNA的存在及其对高生物量和低生物量样品的影响,我们使用了纯培养的16S rRNA基因序列分析沙门氏菌bongori它经历了五轮连续的十倍稀释(相当于大约108细胞作为输入进行DNA提取的原始未稀释样品,至103.细胞稀释5)。美国bongori之所以选择它,是因为我们在之前的研究中没有观察到它是一种污染物,它可以与其他污染物区分开来沙门氏菌16S rRNA基因测序。以纯培养为起始模板,不论起始生物量,任何生物以外美国bongori因此,在随后的DNA测序结果中观察到的DNA必须来自污染。稀释系列的等分被送到三个研究所(伦敦帝国理工学院,ICL;伯明翰大学;威康信托基金会桑格研究所,WTSI),并用不同批次的FastDNA SPIN Kit for Soil (Kit FP)进行处理。使用20和40个PCR循环生成16S rRNA基因扩增子,并返回WTSI进行Illumina MiSeq测序。
美国bongori在原始的未稀释培养物中鉴定出唯一的生物体,但随着随后的稀释,一系列污染细菌群的相对丰度增加,而美国bongori并发读数减少(图1).通过第五次稀释,相当于输入生物量大约103.沙门氏菌细胞、污染是测序结果的主要特征。这一模式在所有三个位点上都是一致的,并且在40次PCR循环中最为明显。这些结果突出了低生物量样品的一个关键问题。最稀释的20-PCR周期样品导致低PCR产物产量,导致在多重测序样品池中代表性不足,因为无法实现等摩尔混合(每个样品的读取计数在附加文件中列出)1:表S1a)。相反,使用40个PCR循环产生足够的PCR产物进行有效测序(每个样品至少返回14,000个reads,参见附加文件1:表S1a),但所得到的序列数据的很大一部分来自污染,非沙门氏菌DNA。值得注意的是,即使只使用20个PCR循环,污染仍然是最低输入生物量的主要因素[见附加文件]1:图S1]。
来自10倍稀释纯培养物和对照的16S rRNA基因测序分类分配的总结。未稀释的DNA提取液中含有大约108细胞和对照(图中标注“con”)为无模板pcr。在ICL, UB和WTSI实验室提取DNA,并进行40个PCR循环扩增。每一列代表一个样本;部分(一)而且(b)在不同的分类水平上描述相同的样本。一)的比例美国bongori序列以黑色显示。非的比例丰度沙门氏菌类别级别的读数由其他颜色表示。随着样品变得更稀,来自培养微生物的测序细菌扩增子的比例减少,污染物变得更加主要。b)占至少一个实验室结果>0.5%的属的丰度,不包括美国bongori.非配置文件沙门氏菌每个实验室/试剂盒批次内的读数是一致的,但在不同站点之间有所不同。
序列分析显示,所有位点之间都有一些相似的分类,包括酸杆菌Gp2,小细菌属、丙酸菌属而且假单胞菌(图1b).观察到不同地点之间的差异,然而Chryseobacterium,肠杆菌属而且Massilia在WTSI更占优势,Sphingomonas在布法罗大学棒状杆菌属,Facklamia而且链球菌在ICL中,与一般放线菌的较大比例(图1a).这说明不同实验室之间的污染物含量存在差异,这可能是由于试剂/试剂盒批次之间的差异或从更广泛的实验室环境引入的污染物。例如,许多污染操作分类单位(OTUs)代表通常在土壤和水中发现的细菌属关节杆菌,伯克霍尔德菌,金杆菌,嗜铬杆菌,假单胞菌,拉氏菌,红球菌而且Sphingomonas,而其他人,如棒状杆菌属,丙酸菌属而且链球菌,是常见的人类皮肤相关生物。通过测序PCR“空白”阴性对照,特别是没有添加模板DNA的PCR扩增超纯水,我们能够区分来自DNA提取试剂盒的分类群和来自其他来源的DNA(如PCR试剂盒试剂、实验室耗材或实验室人员)。所有PCR空白对照中均无63个类群,但在一个或多个序列稀释中以>0.1%的比例丰度存在美国bongori样本[见附加文件1:图S2],表明它们是在DNA提取阶段引入到样品中的。其中包括在三个地点都观察到的几个丰富的属,如酸杆菌Gp2,洋葱,未分类伯克霍氏科和Mesorhizobium.它还包括类群,如Hydrotalea而且Bradyrhizobium这可能表明,同一类型DNA提取试剂盒的不同批次之间的污染物存在差异。
细菌生物量的定量PCR
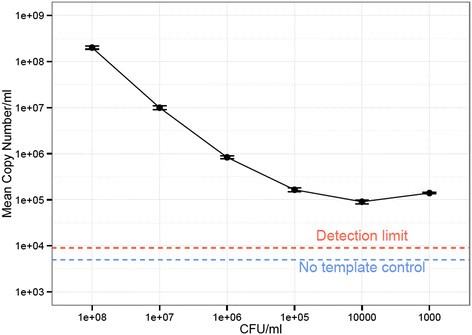
为了评估样本中存在多少背景细菌DNA,我们对细菌16S rRNA基因进行了qPCR,并参考标准曲线计算了存在的基因拷贝数。假设完全没有污染,16S rRNA基因的拷贝数应该与稀释有关美国bongori然后线性化简。然而,在第三次稀释时,拷贝数保持稳定,并没有进一步减少,这表明从DNA提取试剂盒中,每μl洗脱量约有500份背景DNA(图2).
纯的散弹宏基因组学美国bongori用四种商业DNA提取试剂盒进行培养
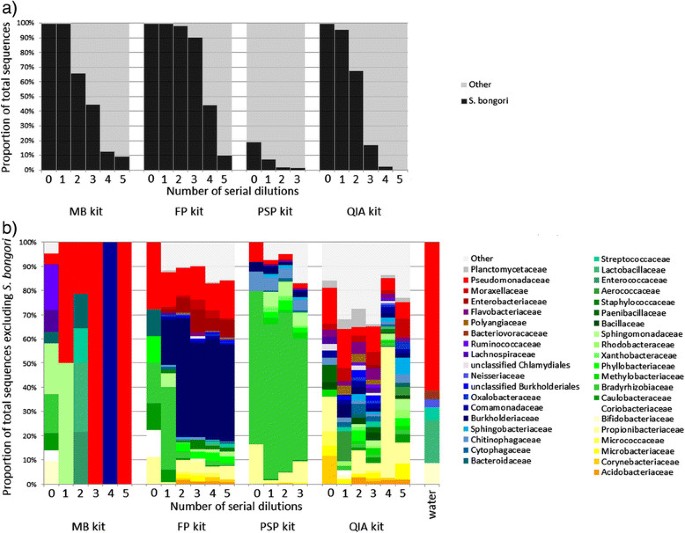
在确定了16S rRNA基因测序结果可能被污染的DNA所混淆后,我们接下来调查了在不涉及靶向PCR步骤的鸟枪宏基因组学研究中是否出现类似的模式。我们假设,如果污染来自DNA提取试剂盒,那么它也应该出现在宏基因组测序结果中。为了调查问题是否仅限于一家制造商,研究人员使用了来自四家不同制造商的DNA提取试剂盒。等分来自同一美国bongori用FastDNA SPIN Kit for Soil (FP)、MoBio ulclean Microbial DNA Isolation Kit (MB)、QIAmp DNA Stool Mini Kit (QIA)和PSP SPIN Stool DNA Plus Kit (PSP)在UB下进行稀释系列。与16S rRNA基因测序一样,发现随着样品稀释度的增加,reads映射到的比例美国bongori参考基因组序列减少(图3.a).无论哪种试剂盒,到第四次序列稀释时,污染始终是序列数据的主要特征,这相当于输入约为104沙门氏菌细胞。
样品在同一实验室内同时处理。如果污染来自实验室环境,那么在每个结果中都应该有相似的细菌组成。相反,观察了一系列环境细菌,这些细菌在每个试剂盒中具有不同的特征(图3.b). FP具有稳定的套件配置,主要由洋葱, PSP是由Bradyrhizobium而QIA试剂盒的细菌DNA组合最为复杂。在PSP、FP和QIA试剂盒的稀释液中,至少有四分之三的稀释液中存在缓生根瘤菌科、伯克霍氏菌科、几丁菌科、康单胞菌科、丙酸杆菌科和假单胞菌科。然而,在不同的kit中,科水平上各类群的相对丰度不同:FP以伯克霍氏菌科(burkholderaceae)和肠杆菌科(Enterobacteriaceae)为标志,PSP以缓生根瘤菌科(Bradyrhizobiaceae)和几丁菌科(Chitinophagaceae)为标志。与其他试剂盒相比,QIA试剂盒的污染相对多样化,其中空气球菌科(Aerococcaceae)、芽孢杆菌科(bacillus aceae)、黄杆菌科(Flavobacteriaceae)、微杆菌科(microbacteraceae)、拟杆菌科(Paenibacillaceae)、Planctomycetaceae)和多血管科(Polyangiaceae)的比例高于其他试剂盒。Kit MB没有明显的污染物特征。这可能是由于测序的读数非常少,稀释2读210次,稀释3读79次,随后稀释的读不到20次[见附加文件]1:表S1b。虽然读取计数只是DNA浓度的半定量测量,但这可能表明该试剂盒的背景污染水平相对较低。
在“空白”水质控制中检测到的污染物类群相对较少,以假单胞菌,在连续稀释的宏基因组样品中检测到。这进一步证明,观察到的污染很可能在很大程度上来自DNA提取试剂盒本身。因此,这些宏基因组结果清楚地表明,污染成为低生物量样品的序列数据的主要特征,并且用于提取DNA的试剂盒可以对观察到的细菌多样性产生影响,即使没有PCR扩增步骤。减少输入生物量再次增加了这些污染物对观察到的微生物群的影响。
污染提取试剂盒对低生物量菌群研究的影响
在确定了不同数量的DNA提取试剂盒中的污染不是恒定的或可预测的之后,我们接下来展示了这对真实数据集的影响。最近在泰国和缅甸边境的一个难民营进行的一项研究使用了现有的鼻咽拭子档案[38]以检查婴儿鼻咽微生物群的发育。在2007/2008年出生的20名儿童队列中,每个月抽取样本,直到两岁,这些拭子的16S rRNA基因图谱用454磷酸测序法测序。
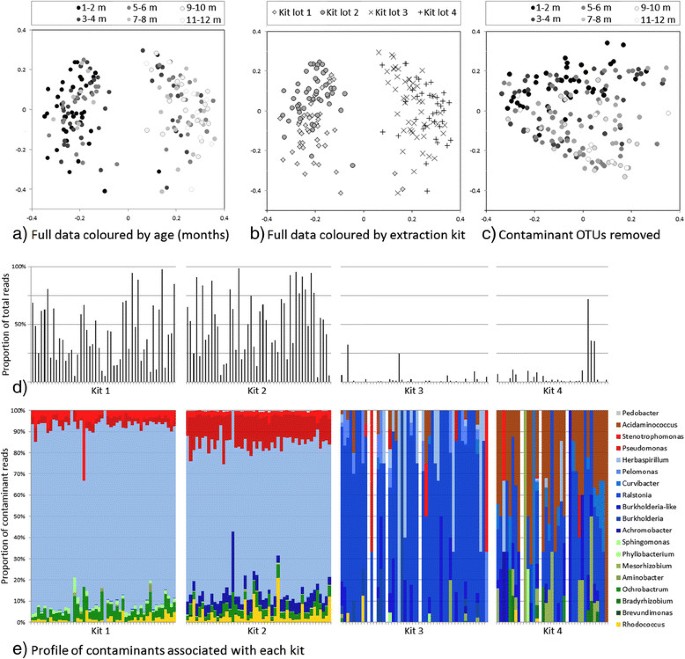
主坐标分析(PCoA)显示了两个不同的聚类,将早期采集的样本与后续采样时间点采集的样本区分开来,这表明鼻咽部存在早期的鼻咽部微生物群(图2)4a).使用4批FP试剂盒提取样品,并记录每个样品使用的试剂盒。对OTUs的进一步分析表明,样品具有不同的群落,这取决于所使用的试剂盒用于DNA提取(图4b,d,e),并且前两个试剂盒的相关OTUs构成了其样本的大部分读取(图4d).由于样本是按时间顺序提取的,而不是随机顺序,这导致了前两个试剂盒的OTUs与年龄相关的错误结论。图中左边的OTUs驱动集群4A和b (P值<0.01),归类为无色杆菌,Aminobacter, Brevundimonas, Herbaspirillum,Ochrobactrum,Pedobacter,假单胞菌,红球菌属,Sphingomonas而且Stenotrophomonas。OTUs向右行驶数据点(P值<0.01)包括在内氨基酸球菌属而且Ralstonia.重要OTUs的完整列表显示在附加文件中1:表S2。一旦污染物被识别并去除,PCoA对样品的聚类就不再具有明显的模式,这表明污染是样品排序的最大驱动因素(图4c).从原始样品档案中获得新的等分,并使用不同的试剂盒批次进行再处理和测序。之前观察到的污染物OTUs没有被检测到,进一步证实了它们在原始鼻咽样本中不存在(手稿准备中,Salter S, Turner P, Turner C, Watthanaworawit W, Goldblatt D, Nosten F, Mather A, Parkhill J, Bentley S)。
因此,这个数据集可以作为一个案例研究,说明来自试剂盒的污染物可能对微生物区系分析和随后的结论产生的重大且可能具有误导性的影响。
讨论
本文的结果表明,在DNA提取试剂盒试剂和更广泛的实验室环境中,细菌DNA或细胞的污染不仅应该是需要PCR扩增的16S rRNA基因测序项目的问题,也应该是霰弹枪宏基因组学项目的问题。
DNA受到PCR试剂、试剂盒和水的污染已被多次报道[3.] - [15]、[17].所鉴定的类群大多是土壤或水栖细菌,通常与固氮有关。对此的一种解释可能是在超纯水储存罐中经常使用氮气而不是空气。3.].DNA提取试剂盒试剂受到污染亦有报导[16]和试剂盒污染是低生物量研究的一个特别挑战,这可能提供很少的模板DNA来与试剂中的扩增DNA竞争[12]、[39].污染问题一直困扰着研究,在新病毒发现领域有一些引人注目的例子,例如XMRV和慢性疲劳综合征的错误关联[40],以及早期人类和病原体的古代DNA研究[41]、[42].在不同实验室的分析中,古冰芯样品的微生物含量也不一致[39].
在分析低生物量样品时,尽管试剂污染的报道如此引人注目,但这一问题的重要性显然仍未得到菌群研究界的重视。良好的对照研究,如在西格尔等.他们通过支气管肺泡灌洗采样检查了肺微生物群,并在大量测序“背景”对照的背景下报告了他们的结果[43].然而,最近许多描述低生物量环境微生物群落的基于DNA测序的出版物没有报告初始样品的DNA定量、阴性对照的测序或描述其污染物去除或识别程序。我们的文献检索表明,有许多低生物量微生物群研究报告的类群,通常在统计上值得注意或核心成员,与我们在这里报告的阴性对照试剂盒试剂和水的类群重叠(见表)1).虽然有可能这些可疑类群确实存在于这些样本中,但在许多情况下,它们在生物学上是出乎意料的:例如,与人类疾病有关的根际相关细菌[27]、[44].引人注目的是,劳伦斯et al。[18]最近用一个在网上分析Bradyrhizobium是包括1000人基因组计划在内的测序数据集的常见污染物。在证明了污染DNA可能对基于序列的数据得出的结论产生的关键影响之后,能够确定哪些观察结果是真实的变得重要起来。对于环境样品,如土壤或水,污染物的识别问题需要特别注意,因为污染物可能是与样品中真正存在的污染物难以区分的分类群。
为了减少潜在的污染,已经设计了许多方法来处理试剂,包括:45]或紫外线[13]、[46] - [48],脱氧核糖核酸酶治疗[10]、[13]、[47]、[49] - [51],限制消化[10]、[13]、[47]、[52]、[53],氯化铯密度梯度离心[10]以及与8-甲氧基补骨脂素的DNA插层和交联[47]、[54],单叠氮丙啶[55]或单叠乙锭[56]、[57].然而,对这些方法的测试显示出不同程度的成功。辐射可降低酶的活性,脱氧核糖核酸酶失活也可破坏聚合酶,限制性内切酶可引入更多污染DNA,未结合的DNA插入器抑制预期模板的扩增[56]、[58].去污的另一种方法是使用大范围引物扩增PCR优先扩增模板DNA [59但这一点,以及PCR试剂的处理,不能解释通过DNA提取试剂盒引入的污染。
一个简单的在网上微生物群研究的方法是使用阴性对照或污染物数据库确定测序的污染物,以便将其从下游分析中筛选出来[17]、[60].如果发现污染生物在生物学上也是可信的,不应排除在分析之外,则可采用其他方法[61].统计方法和基本可视化来比较阴性对照和样本之间的相对丰度或秩序信息可能有助于确定类群是否也是真实的。可供选择的生物信息学方法,如寡分型[62],可以提供污染物OTUs和属于同一属或种的真正OTUs之间的精细区分。对于鸟枪宏基因组学研究,使用菌株特异性基因或跨多个标记基因使用系统发育信息也可能提供必要的区分。偏离中立的社区形成模型,比较源社区(工具包对照)和接收社区,在这种情况下也可能有用[63].
通过添加阴性测序控制(具体来说,与真实样品使用相同的DNA提取和PCR扩增试剂盒处理的无模板“空白”,在同一次运行中测序),可以识别源自污染的reads,并将它们与来自实际组成分类群的reads区分开来。我们制定了一套可能有助于限制试剂污染影响的建议(方框1)。通过了解常见的污染物种,仔细收集不同批次的采样、提取和PCR试剂盒的对照,并对这些对照进行测序以监测这些对照的含量,应该有可能有效地减轻微生物区系研究中污染物的影响。
结论
我们已经证明,提取试剂盒和实验室试剂中的细菌DNA污染可以显著影响微生物区系研究的结果,特别是在调查含有低微生物生物量的样品时。无论是16S rRNA基因测序项目,还是霰弹枪宏基因组项目,这种污染都是一个值得关注的问题,前者需要有目标的PCR扩增和富集,后者则不需要。菌群研究界对这一问题的认识对于确保研究得到充分控制和避免从不依赖培养的调查中得出错误结论非常重要。
方法
样品
对于16S rRNA基因和宏基因组分析,沙门氏菌bongori菌株NCTC-12419在37℃无抗生素的Luria-Bertani (LB)板上培养过夜。用单个菌落接种LB肉汤,在37°C下摇晃培养过夜。的OD600检索时是1.62,相当于10左右9菌落形成单位(CFU)/ml。从培养物中取20 μl接种于LB上,孵育一夜后观察其为纯培养物。在新鲜LB中从发酵剂中提取5份10倍稀释液。每种稀释液均立即等分(1ml)储存在-80°C下,并将副本用干冰运输到伦敦帝国理工学院和伯明翰大学。
在鼻咽菌群研究中,样本取自泰国Maela难民营一组婴儿的鼻咽拭子,如前所述[38].在脱脂牛奶、色氨酸、葡萄糖和甘油培养基(STGG)中进行漩涡处理,然后在-80°C保存。
DNA提取
对于16S rRNA基因分析工作,三个研究所(帝国理工学院,ICL;伯明翰大学;威康信托基金会桑格研究所(WTSI)提取了DNA美国bongori根据制造商的协议,使用不同生产批次的FastDNA土壤旋转试剂盒(MP Biomedicals, Santa Ana, California, USA试剂盒批次#38098,#15447和#30252),平行进行分份。每个研究所提取一次。UB和WTSI从200 μl样品中提取DNA,用50 μl洗脱;ICL从500 μl样品中提取,100 μl洗脱。这意味着我们的DNA提取在五倍连续稀释范围内的样品生物量从大约108减少到103.细胞。
宏基因组测序,各200 μl美国bongori在布法罗大学使用四种市售DNA提取试剂盒进行稀释。所有试剂盒最终洗脱量均为100 μl /份。FP套件(批号38098)是根据制造商的协议使用的,除均质器步骤外。这是用Qiagen Tissue Lyser进行的:以30/秒的速度进行1分钟,然后在冰上冷却30秒,重复三次。ulclean微生物DNA分离试剂盒(MO BIO Laboratories, Carlsbad, California, USA)(试剂盒MB,批次#U13F22)根据制造商的方案使用,除均质化外,均质化被10分钟的涡流取代。QIAmp DNA粪便迷你试剂盒(Qiagen, Venlo, Limburg, Netherlands)(试剂盒QIA,批号145036714)根据制造商的粪便病原体检测方案使用。加热步骤为90°C。PSP Spin Stool DNA Plus试剂盒(STRATEC Molecular, Birkenfeld, Germany)(试剂盒PSP,批次#JB110047)根据制造商的粪便匀浆规程使用。每个样品与每个试剂盒一起加工一次。所有的提取试剂都包含在所有试剂盒中,除了洗涤步骤中添加的乙醇。 In addition to these samples, a negative control was included consisting of ultrapure water that had not been processed with any DNA extraction kit.
对于鼻咽菌群研究,从每个样品中提取200 μl,并使用制造商对FP试剂盒方案进行涡流修改。然后DNA被运送到WTSI进行进一步处理和测序(见下文)。
qPCR
克隆的近全长16S rRNA基因,得到标准曲线弧菌natriegens使用引物27 F和1492R扩增的DSMZ 759 [64]注入TOPO TA载体(Life Technologies, Carlsbad, California, USA),使用荧光法定量(quantum - it, Life Technologies),稀释以产生标准曲线从108到103.每μl拷贝数。使用ViiA 7 Real-time PCR系统(Life Technologies)和SYBR Fast qPCR Master Mix (KAPA Biosystems, Wilmington, Massachusetts, USA)对每个细菌16S rRNA基因的V4区进行定量PCR美国bongori稀释提取(使用FastDNA SPIN Kit for Soil (MP Biomedicals),试剂盒号15447)。所用引物为:S-D-Bact-0564-a-S- 15,5 '- aytgggydtaaagng和s - d - bact -0785-b- a- 18,5 - tacnvgggtatctaatcc [65]生成一个253 bp的扩增子。反应(15 μl)分为3个重复,包括无模板对照。引物10 μl稀释0.3 μl, SYBR Fast mastermix 7.5 μl,微生物DNA游离PCR水(MOBIO) 1.9 μl, 1:5稀释模板液5 μl(避免移液小于5 μl)。循环条件为90°C持续3分钟,然后进行40次循环:95°C持续20秒,50°C持续30秒,72°C持续30秒。熔体曲线在15分钟内从60°C运行到95°C。
测序
的样本美国bongori培养16S rRNA基因分析使用针对基因V1-V2区域的条形码融合引物(27f_Miseq: AATGATACGGCGACCACCGAGATCTACAC TATGGTAATT CC AGMGTTYGATYMTGGCTCAG和338R_MiSeq: CAAGCAGAAGACGGCATACGAGAT nnnnnnnnnnnnn AGTCAGTCAG AA GCTGCCTCCCGTAGGAGT,其中n字符串代表每个研究样本使用的唯一12位mer条形码,然后在Illumina MiSeq平台上使用2 × 250 bp周期进行测序。在WTSI、ICL和UB使用Q5高保真PCR试剂盒(New England Biolabs, Ipswich, Massachusetts, USA)进行PCR扩增,使用新鲜试剂和耗材、高压灭菌的微离心管、过滤的移液头,并在罩内进行,以降低空气污染的风险。每个样本分别在94°C 30秒、53°C 30秒、68°C 2分钟的条件下进行20和40个PCR循环扩增。每批都包括pcr扩增超纯水样品的阴性对照。PCR产物在琼脂糖凝胶上可见:所有40°循环样品和20°循环样品的前四次稀释都可见条带。数据存入ENA项目接入EMBL: ERP006737;样本详细信息和个人登录号码详细在附加文件1:表S1a。
对于宏基因组测序,所有样本都使用Nanodrop (Thermo Scientific, Waltham, Massachusetts, USA)和Qubit (Life Technologies)机器进行量化,并且在Illumina Nextera XT文库制备前不需要稀释(根据制造商的协议进行处理)。库在Illumina MiSeq上以配对的250碱基模式进行多路复用,遵循标准的MiSeq清洗协议。数据保存在ENA项目接入EMBL: ERP006808下。样例详细信息和个人登录号码提供在附加文件1:表S1b。
在鼻咽菌群研究中,从182支棉签中提取的DNA进行了pcr扩增和条形码,如前所述,在454平台上对16S rRNA基因V3-V5区域进行了测序[66].
序列分析
对于16S rRNA基因分析,数据处理使用母亲[67].母亲MiSeq SOP [68除了screen之外,其余都是。seqs(使用97.5百分位值的最大长度)和chimera检验(使用Perseus执行)[69而不是UCHIME。读计数后处理和每个样本中存在的属级系统型的数量在附加文件中显示1:表S1a。
对于宏基因组分析,使用Trimmomatic对低质量区域和适配器序列进行质量检查和修剪[70].在微生物RefSeq蛋白质数据库中,使用LAST在六帧翻译模式下对分类分配进行相似测序[71].分类学分配使用MEGAN进行确定,MEGAN对分类学分配采用最低共同祖先(LCA),使用设置最小支持度2,最小评分250,最大期望0.1,最高百分比10.0 [72].
对于鼻咽菌群研究,使用母Schloss SOP对数据进行处理、清洗和分析[73]并随机次采样到每个样本200个序列读取。作为污染鉴定程序的一部分,转移瘤包[74]用于识别与每个提取试剂盒批次显著相关的OTUs。与母亲一起生成Jaccard PCoA图,比较包含和不包含这些标记OTUs的数据集。
作者的贡献
SJS、MJC、NJL和AWW设计了实验。SJS、ET、STC和NJL进行了实验。SJS, MJC, NJL和AWW分析数据并编制数字。PT提供的数据证明了萃取试剂盒污染物的影响。WOC、MFM、NJL和JP提供了资源、指导和支持。SJS、MJC、NJL和AWW共同撰写了这篇论文。所有作者都阅读并批准了最终的手稿。
额外的文件
参考文献
古宁V, Engelbrektson A, Ochman H, Hugenholtz P:稀有生物圈的褶皱:焦磷酸测序误差可能导致多样性估计的人为膨胀。环境微生物学报,2010,12:118-123。10.1111 / j.1462-2920.2009.02051.x。
Fv W, Göbel UB, Stackebrandt E:环境样品中微生物多样性的测定:基于pcr的rRNA分析的陷阱。中国生物医学工程学报,1997,26(3):329 - 329。10.1111 / j.1574-6976.1997.tb00351.x。
Kulakov LA, McAlister MB, Ogden KL, Larkin MJ, O 'Hanlon JF:工业系统中超纯水的细菌污染分析。环境科学学报,2002,29(3):344 - 344。10.1128 / aem.68.4.1548 - 1555.2002。
McAlister MB, Kulakov LA, O’hanlon JF, Larkin MJ, Ogden KL:从超纯水中分离出的三种细菌的生存和营养需求。中国生物医学工程学报,2002,29:75-82。10.1038 / sj.jim.7000273。
Kéki Z, Grébner K, Bohus V, Márialigeti K, Tóth EM:特殊寡营养培养基在超纯水源细菌群落培养中的应用。微生物免疫学报,2013,60:345-357。10.1556 / AMicr.60.2013.3.9。
Bohus V, Kéki Z, Márialigeti K, Baranyi K, Patek G, Schunk J, Tóth EM:电厂超纯水含储罐中的细菌群落。微生物免疫学报,2011,58:371-382。10.1556 / AMicr.58.2011.4.12。
McFeters GA, Broadaway SC, Pyle BH, Egozy Y:运行实验室水净化系统中的细菌分布。环境科学与技术,1998,29(3):349 - 349。
Nogami T, Ohto T, Kawaguchi O, Zaitsu Y, Sasaki S:超纯水细菌污染的估计:抗dna抗体的应用。中华医学杂志,1998,29(4):357 - 357。10.1021 / ac9805854。
Shen H, Rogelj S, Kieft TL:灵敏的实时PCR检测商业试剂中低水平的嗜肺军团菌污染。生物化学学报,2006,30(4):357 - 357。10.1016 / j.mcp.2005.09.007。
Rand KH, Houck H:Taq聚合酶含有来源不明的细菌DNA。生物化学学报,1999,4:445- 445。10.1016 / 0890 - 8508 (90) 90003 - i。
梅华德,李志刚,李志刚,李志刚。DNA污染的研究进展Taq在军团菌5S核糖体RNA引物扩增过程中发生的聚合酶。分子细胞探针,1994,8:11-14。10.1006 / mcpr.1994.1002。
Tanner MA, Goebel BM, Dojka MA, Pace NR:来自不同环境设置的特定核糖体DNA序列与实验污染物相关。环境科学与技术,1998,29(3):366 - 366。
Corless CE, Guiver M, Borrow R, Edwards-Jones V, Kaczmarski EB, Fox AJ:实时通用16S rRNA PCR的污染和敏感性问题。中华微生物学杂志,2000,38(4):457 - 457。
王丽娟,王丽娟,王丽娟,王丽娟,等:16S rDNA V1和V3可变区PCR扩增中混合细菌DNA污染的鉴定。中国生物医学工程学报,2003,19(3):337 - 337。10.1016 / s0378 - 1097(02) 01190 - 4。
李宝军,李宝军,邹宁,罗素生:细菌噬菌体样DNA序列在Taq DNA聚合酶试剂中的存在。临床微生物学杂志,2004,42:2164 - 2164。10.1128 / jcm.42.5.2264 - 2267.2004。
Mohammadi T, Reesink HW, vandenbrouck - grauls CM, Savelkoul PH:从商业核酸提取试剂盒试剂中去除污染DNA。中国生物医学工程学报,2005,29(4):457 - 457。10.1016 / j.mimet.2004.11.018。
Barton HA, Taylor NM, Lubbers BR, Pemberton AC:从低生物量碳酸盐岩中提取DNA:一种减少污染的改进方法和低生物量污染物数据库。中国生物医学工程学报,2006,26(3):344 - 344。10.1016 / j.mimet.2005.10.005。
Laurence M, Hatzis C, Brash DE:新一代测序中阻碍低丰度微生物发现的常见污染物。公共科学学报,2014,9:e97876-10.1371/journal.pone.0097876。
Oberauner L, Zachow C, Lackner S, Högenauer C, Smolle KH, Berg G:被忽视的多样性:16S焦磷酸盐测序揭示重症监护病房复杂细菌群落。科学通报,2013,3:1413-10.1038/srep01413。
La Duc MT, Kern R, Venkateswaran K:航天器和相关环境的微生物监测。中华微生物学杂志,2004,47:150-158。10.1007 / s00248 - 003 - 1012 - 0。
凌志,刘旭,罗勇,袁磊,Nelson KE,王勇,向春,李磊:中国健康大学生人体微生物区系的焦磷酸测序分析。中国生物医学工程学报,2013,30(4):359 - 364。
Benítez-Páez A, Álvarez M, Belda-Ferre P, Rubido S, Mira A, Tom’S I:用16S rDNA焦硫测序检测拔牙后的短暂菌血症:一项初步研究。公共科学学报,2013,8:e57782-10.1371/journal.pone.0057782。
Amar J, Serino M, Lange C, Chabo C, Iacovoni J, Mondot S, Lepage P, Klopp C, Mariette J, Bouchez O, Perez L, Courtney M, Marre M, Klopp P, Lantieri O, Doré J, Charles M, Balkau B, Burcelin R:组织细菌参与人类糖尿病发病:一个概念的证据。中华糖尿病杂志,2011,29(4):344 - 344。10.1007 / s00125 - 011 - 2329 - 8。
Branton WG, Ellestad KK, Maingat F, Wheatley BM, Rud E, Warren RL, Holt RA, Surette MG, Power C: HIV/AIDS患者大脑微生物种群:?-变形菌门占主导地位,与宿主免疫状态无关。公共科学学报,2013,8:e54673-10.1371/journal.pone.0054673。
Borewicz K, Pragman AA, Kim HB, Hertz M, Wendt C, Isaacson RE:肺移植中肺微生物组的纵向分析。中国生物医学工程学报,2013,37(3):344 - 344。10.1111 / 1574 - 6968.12053。
Dong Q, Brulc JM, Iovieno A, Bates B, Garoutte A, Miller D, Revanna KV, Gao X, Antonopoulos DA, Slepak VZ, Shestopalov VI:健康人结膜细菌多样性。中华眼科杂志,2011,29(4):344 - 344。10.1167 / iovs.10 - 6939。
陈璇,夏蒙基,李振华,李振华,李振华:微生物失调与乳腺癌的相关性研究。公共科学学报,2014,9:e83744-10.1371/journal.pone.0083744。
Kuehn JS, Gorden PJ, Munro D, Rong R R, Dong Q, Plummer PJ, Wang C C, Phillips GJ:奶牛乳腺炎细菌群落谱分析的研究进展。公共科学学报,2013,8:e61959-10.1371/journal.pone.0061959。
Srinivas G, Möller S,王娟,Künzel S, Zillikens D, Baines JF, Ibrahim SM:自身免疫性皮肤起泡易感基因-微生物相互作用的全基因组图谱。Nat通信,2013,4:2462-10.1038/ncomms3462。
Boissière A, Tchioffo MT, Bachar D, Abate L, Marie A, Nsango SE, Shahbazkia HR, Awono-Ambene PH, Levashina EA, Christen R, Morlais I:疟疾蚊子媒介的中肠微生物群冈比亚疟蚊以及与恶性疟原虫感染。公共科学图书馆,2012,8:e1002742-10.1371/journal.ppat.1002742。
McKenzie VJ, Bowers RM, Fierer N, Knight R, Lauber CL:共生两栖动物在野生种群中拥有独特的皮肤细菌群落。中华医学杂志,2012,6:588-596。10.1038 / ismej.2011.129。
Carlos C, Torres TT, Ottoboni LM:巴西珊瑚物种粘液的细菌群落和物种特异性关联。科学通报,2013,3:1624-10.1038/srep01624。
程晓霞,田晓玲,王玉生,林瑞敏,毛志才,陈宁,谢碧:松林线虫微生物群宏基因组分析揭示了一种对异种生物降解至关重要的共生关系。科学通报,2013,3:1869-
杨晓东,李志强,李志强:蚯蚓肾病细菌的研究进展。中国生物工程学报,2013,29(3):344 - 344。10.1016 / j.ympev.2012.12.005。
Knowlton C, Veerapaneni R, D 'Elia T, Rogers SO:格陵兰岛和南极洲古冰芯部分的微生物分析。生物学(巴塞尔)。2013, 2: 206-232。
Shtarkman YM, Koçer ZA, Edgar R, Veerapaneni RS, D 'Elia T, Morris PF, Rogers SO:冰川下沃斯托克湖(南极洲)的吸积冰包含了来自水生、海洋和沉积物中的细菌和真核生物的不同序列。公共科学学报,2013,8:e67221-10.1371/journal.pone.0067221。
DeLeon-Rodriguez N, Lathem TL, Rodriguez-R LM, Barazesh JM, Anderson BE, Beyersdorf AJ, Ziemba LD, Bergin M, Nenes A, Konstantinidis KT:对流层上部微生物群落:物种组成和流行度,热带风暴的影响和大气影响。自然科学进展,2013,29(3):379 - 379。10.1073 / pnas.1212089110。
Turner P, Turner C, Jankhot A, Helen N, Lee SJ, Day NP, White NJ, Nosten F, Goldblatt D:纵向研究链球菌引起的肺炎在泰缅边境,一群婴儿和他们的母亲被运送。公共科学学报,2012,7:e38271-10.1371/journal.pone.0038271。
韦勒斯列夫E,汉森AJ,波因纳尔HN:从化石冰和永久冻土中分离核酸和培养物。生态环境学报,2004,19:141-147。10.1016 / j.tree.2003.11.010。
Kearney MF, Spindler J, Wiegand A, Shao W, Anderson EM, Maldarelli F, Ruscetti FW, Mellors JW, Hughes SH, Le Grice SF, Coffin JM:报告有XMRV感染的患者样本中的多重污染源。公共科学学报,2012,7:e30889-10.1371/journal.pone.0030889。
古柏A,波因纳尔HN:古代DNA:要么做对,要么就不做。科学通报,2000,29 (3):379 - 379 / c。
罗伯茨C,英厄姆S:在古病理学中使用古代DNA分析:对已发表论文的批判性分析,并对未来的工作提出建议。中华骨古科学杂志,2008,18:600-613。10.1002 / oa.966。
Segal LN, Alekseyenko AV, Clemente JC, Kulkarni R, Wu B, Chen H, Berger KI, Goldring RM, Rom WN, Blaser MJ, Weiden MD:声门上类群肺微生物群富集与肺部炎症增加相关。微生物学学报,2013,1:19-10.1186/2049-2618-1-19。
Bhatt AS, Freeman SS, Herrera AF, Pedamallu CS, Gevers D, Duke F, Jung J, Michaud M, Walker BJ, Young S, Earl AM, Kostic AD, Ojesina AI, Hasserjian R, Ballen KK, Chen YB, Hobbs G, Antin JH, Soiffer RJ, Baden LR, Garrett WS, Hornick JL, Marty FM, Meyerson M:基于序列的肠慢根瘤菌在脊髓结肠炎综合征中的发现。中华实用医学杂志,2013,29(3):344 - 344。10.1056 / NEJMoa1211115。
Deragon JM, Sinnett D, Mitchell G, Potier M, Labuda D:利用γ辐照消除PCR DNA污染。核酸研究,1990,18:6149-10.1093/nar/18.20.6149。
Sarkar G, Sommer SS:揭示PCR污染。自然科学学报,2000,27(3):344 - 344。
Klaschik S, Lehmann LE, Raadts A, Hoeft A, Stuber F: real-time pcr检测低浓度细菌16S DNA试剂不同去污染方法的比较。中国生物医学工程学报,2002,22:331 - 332。10.1385 / MB: 22:3:231。
Tamariz J, Voynarovska K, Prinz M, Caragine T:紫外线照射对塑料制品和水中DNA外源的应用,用于低拷贝数DNA的扩增。法医学学报,2006,29(4):489 - 497。10.1111 / j.1556-4029.2006.00172.x。
Hilali F, Saulnier P, Chachaty E, Andremont A:检测低浓度16S rRNA基因的聚合酶链反应试剂的去污。中国生物工程学报,1997,37(3):357 - 357。10.1007 / BF02740812。
Heininger A, Binder M, Ellinger A, Botzenhart K, Unertl K, Döring G:主混合试剂DNase预处理提高了通用16S rRNA基因PCR结果的有效性。中华微生物学杂志,2003,41:1763-1765。10.1128 / jcm.41.4.1763 - 1765.2003。
Silkie SS, Tolcher MP, Nelson KL:试剂去污消除假阳性大肠杆菌qPCR。中国生物医学工程学报,2008,29(3):344 - 344。10.1016 / j.mimet.2007.12.011。
王晓明,王晓明,王晓明,等:Taq DNA聚合酶中细菌DNA的酶切酶解。中华微生物学杂志,1999,37:3402-3404。
Mohammadi T, Reesink HW, vandenbrouck - grauls CM, Savelkoul PH:实时PCR检测血小板浓缩物中真菌16S核糖体DNA的优化。中华微生物学杂志,2003,41:496 - 498。10.1128 / jcm.41.10.4796 - 4798.2003。
Hughes MS, Beck LA, Skuce RA: Taq DNA聚合酶中DNA序列的鉴定和消除。中华微生物学杂志,1994,32:2007-2008。
Vaishampayan P, Probst AJ, La Duc MT, Bargoma E, Benardini JN, Andersen GL, Venkateswaran K:低生物量洁净室环境中可活微生物群落的新视角。中华医学杂志,2013,7:312-324。10.1038 / ismej.2012.114。
Rueckert A, Morgan HW:从聚合酶链反应中去除污染DNA使用单叠氮乙胺。中国生物医学工程学报,2007,29(3):366 - 366。10.1016 / j.mimet.2006.11.006。
Patel P, Garson JA, Tettmar KI, Ancliff S, McDonald C, Pitt T, Coelho J, Tedder RS:一种单叠氮乙胺增强内控通用16S rDNA实时聚合酶链反应检测血小板浓缩液中细菌污染的方法的建立。《输血杂志》,2012,52:1423-1432。10.1111 / j.1537-2995.2011.03484.x。
Champlot S, Berthelot C, Pruvost M, Bennett EA, Grange T, Geigl EM:用于高敏感PCR应用的高效多策略PCR试剂DNA去污染程序。公共科学学报,2010,5:e13042-10.1371/journal.pone.0013042。
张世生,徐海林,程继昌,曾晓平:一种低丰度细菌宽范围检测的高效方法。公共科学学报,2011,6:e20303-10.1371/journal.pone.0020303。
邦费特,蔡巴G,齐默R, Friedel CC:挖掘RNA-Seq数据的感染和污染。公共科学学报,2013,8:e73071-10.1371/journal.pone.0073071。
Knights D, Kuczynski J, Charlson ES, Zaneveld J, Mozer MC, Collman RG, Bushman FD, Knight R, Kelley ST:贝叶斯社区范围内培养独立的微生物源跟踪。工程学报,2011,8:761-763。10.1038 / nmeth.1650。
Eren AM, Maignen L, Sul WJ, Murphy LG, Grim SL, Morrison HG, Sogin ML:利用16S rRNA基因数据区分密切相关的微生物类群。方法生态学报,2013,4:1111-1119。10.1111 / 2041 - 210 x.12114。
Morris A, Beck JM, Schloss PD, Campbell TB, Crothers K, Curtis JL, Flores SC, Fontenot AP, Ghedin E, Huang L, Jablonski K, Kleerup E, Lynch SV, Sodergren E, Twigg H, Young VB, Bassis CM, Venkataraman A, Schmidt TM, Weinstock GM:健康非吸烟者和吸烟者呼吸道微生物群的比较。中华呼吸危重症杂志,2013,37(3):357 - 357。10.1164 / rccm.201210 - 1913摄氏度。
Lane DJ: 16S/23S rRNA测序。细菌分类学中的核酸技术“,”编著:Stackebrandt E, Goodfellow M. 1991, Wiley, Chichester, UK, 115-175。
Klindworth A, Pruesse E, Schweer T, Peplies J, Quast C, Horn M, Gl°Ckner FO:经典和下一代基于测序的多样性研究的一般16S核糖体RNA基因PCR引物的评估。中国生物工程学报,2013,41:e1-10.1093/nar/gks808。
Cooper P, Walker AW, Reyes J, Chico M, Salter SJ, Vaca M, Parkhill J:人感染鞭虫Trichuris trichiura与粪便微生物群的改变无关。公共科学学报,2013,8:e76573-10.1371/journal.pone.0076573。
Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, Van Horn DJ, Weber CF:介绍母亲:用于描述和比较微生物群落的开源、平台独立、社区支持的软件。环境科学学报,2009,27(3):357 - 357。10.1128 / AEM.01541-09。
Kozich JJ, Westcott SL, Baxter NT, Highlander SK, Schloss PD:开发了用于分析MiSeq Illumina测序平台上扩增子序列数据的双指标测序策略和管理管道。环境科学学报,2013,29(3):344 - 344。10.1128 / AEM.01043-13。
Quince C, Lanzen A, Davenport RJ, Turnbaugh PJ:去除焦磷酸测序扩增子中的噪声。中国生物医学工程学报,2011,29(4):344 - 344。
Bolger AM, Lohse M, Usadel B: Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学,2014,30:2114-2120。10.1093 /生物信息学/ btu170。
王晓燕,王晓燕,王晓燕,王晓燕,王晓燕:适应性种子驯化基因组序列比较。基因组学报,2011,21:487-493。10.1101 / gr.113985.110。
胡森DH, Mitra S, Ruscheweyh HJ, Weber N, Schuster SC: MEGAN4环境序列综合分析。基因组学报,2011,21:1552-1560。10.1101 / gr.120618.111。
Schloss PD, Gevers D, Westcott SL:减少PCR扩增和测序伪影对16S rrna研究的影响。公共科学学报,2011,6:e27310-10.1371/journal.pone.0027310。
王晓明,王晓明,王晓明,等:应用统计学方法检测临床宏基因组差异丰度特征。中国生物医学工程学报,2009,29 (4):377 - 377 /journal.pcbi.1000352。
确认
SJS, JP, AWW和测序费用由Wellcome Trust资助(资助号098051)。澳门赛马会由威康信托中心呼吸道感染基础科学奖学金资助。STC由国家卫生研究所(NIHR)资助。WOC和MFM由威康基金会联合高级研究员奖支持,该奖项也支持EMT。这项研究得到了威康基金会临床培训奖学金(资助号083735/Z/07/Z)的支持。NJL由医学研究理事会生物医学信息学特别培训奖学金资助。AWW和阿伯丁大学罗维特营养与健康研究所获得苏格兰政府农村和环境科学与分析服务(RESAS)的核心资金支持。本文仅代表作者个人观点,并不代表威康基金会、英国国民医疗服务体系、英国国家卫生研究院或英国卫生部。
我们要感谢威康基金会桑格研究所的核心测序团队,Paul Scott在实验室的协助,并提供了一份来自多个置换扩增试剂盒的污染物清单,Phil James在qPCR方面的协助,以及Christine Boinett在R方面的建议。
作者信息
作者及隶属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
权利和权限
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(http://creativecommons.org/licenses/by/4.0),允许在任何媒介上不受限制地使用、传播和复制,前提是原创作品的名称要注明出处。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
索尔特,s.j.,考克斯,m.j.,塔瑞克,E.M.et al。试剂和实验室污染会严重影响基于序列的微生物组分析。BMC医学杂志12, 87(2014)。https://doi.org/10.1186/s12915-014-0087-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12915-014-0087-z
关键字
- 污染
- 微生物组
- 微生物群
- 宏基因组
- 16 s rRNA