文摘
背景
人类的肠道微生物群的大多数方面都会涉及到健康和疾病;然而,大多数的细菌在这个社区被认为是unculturable,所以研究依赖于分子的方法。这些方法通常不允许有机体的隔离,需要完全探索的功能角色细菌与宿主表型明确协会。使用相结合的文化和16 s rRNA基因测序,称为人文分子剖析,我们表明,绝大多数的细菌被16 s测序人类肠道微生物群的可以培养。
方法
五个新鲜,厌氧粪便样本使用33媒体和孵化的盘子厌氧培养和耗氧导致66年文化人文条件的分子分析。的可耕种的部分粪便微生物群是由比较操作分类单位(辣子鸡)恢复16 s测序文化无关的文化板块辣子鸡粪便样品的测序。有针对性的隔离Lachnospiraceae菌株使用条件定义为人文进行分子分析两个新鲜的粪便样本。
结果
我们表明,人文分子分析,利用66年文化条件加上16 s rRNA基因测序,使培养的平均95%的辣子鸡出席超过0.1%在粪便样本丰富。无教养的辣子鸡低丰度在凳子上。重要的是,比较culture-enrichment文化无关测序显示大多数的辣子鸡发现只有通过文化,突出文化的优势为研究肠道微生物群的多样性。应用人文分子分析目标Lachnospiraceae压力导致了79年的复苏隔离,12是对人类微生物组的项目“通缉犯”名单。
结论
我们表明,通过人文分子分析,绝大多数的细菌在人类肠道微生物群可以培养和该方法揭示细菌多样性与文化无关测序相比。此外,此方法可以应用于目标恢复特定的细菌群。这种方法允许隔离细菌从肠道微生物群的利益,提供新的机会去探索microbiota-host交互机制和人类微生物群的多样性。
背景
胃肠道微生物群是一个高度多样化的社区,但绝大多数的细菌被认为是最近以来unculturable sequencing-based研究显示更大的多样性比之前检测到文化(1,2]。因此,大多数研究描述人类肠道微生物依靠文化无关的测序方法。这些研究提供了洞察的社区构成肠道微生物群在健康个体3),它如何改变与环境扰动(4,5),及其在各种疾病的潜在作用[6,7]。然而,有限制的信息可以从分子方法获得单独的隔离生物需要定义特定的细菌导致的角色或维持健康和疾病状态。文化也决定了可行的人口在一个社区,虽然大多数DNA分子方法不区分获得生存或死亡的细胞。此外,文化使用选择性的媒体支持更丰富的细菌的生长和检测可能错过了测序深度在文化无关的研究不足8]。
一些最近的研究试图描述可耕种的人类肠道微生物群结合文化与下一代测序(9),但只有最多50%的业务分类单元(辣子鸡)检测到16 s rRNA基因测序的粪便样本可耕种的10,11]。Lagier et al。8)开发了一种文化组学方法,它使用了212粪便微生物群的培养条件。然而,细菌菌落与MALDI-TOF质谱分析发现,很难直接比较辣子鸡获得焦磷酸测序以确定微生物群的可耕种的比例(8]。本研究确定了174年的小说《细菌物种,证明文化仍然是一个有价值的探索肠道微生物的方法。另一项研究中,由Rettedal et al。11)测试几个文化条件来捕获比例代表和维护整个社区的肠道微生物群的结构。从十个媒体,88%的家庭水平分类群的恢复,有趣的是,辣子鸡文化被发现超过40%文化无关的方法,展示文化的潜在优势捕捉在肠道微生物多样性。另一项研究表明,个性化的培养条件,尽管只占phylotypes观察到50%的文化无关的排序,可以在类似的方式征服无菌鼠完整的粪便样本(10]。
在这项研究中,一种人文的方法分子分析(12与16 s rRNA)相结合广泛的细菌培养基因测序(无花果。1)是用于调查的比例从胃肠道容易培养的微生物群。使用66文化条件和应用相同的16 s rRNA基因测序方法培养社区和粪便样本,我们表明,绝大多数的辣子鸡可以通过人文分子检测分析和文化多样性大于文化无关的方法检测到。人文的分子分析进一步应用于有针对性的培养Lachnospiraceae隔离。
方法
样品收集
本研究通过汉密尔顿集成研究伦理委员会和捐助者提供之前同意参与。健康志愿者(HV1-7)没有胃肠道(GI)症状和6个月内没有使用抗生素的研究。肠易激综合征(IBS)患者(IBS1-4)被诊断为腹泻型或混合亚型根据罗马III标准(13)和招募GI麦克马斯特大学临床研究单位。
新鲜粪便样本被转移到无菌标本容器后立即排便和储存在密封袋含有厌氧袋(英国Oxoid)和冰敷,直到转移到厌氧室(5%股份有限公司2,5% H2,90% N2;替代高能激光实验室,科尼利厄斯,或者美国),发生在1 - 5 h的集合。之后,所有的工作都是在厌氧室完成。样本与无菌抹刀和机械混合培养如下所述。
培养的粪便样本
在厌氧室,0.1 g的粪便样本稀释900年μl预脑心浸液肉汤(BHI)(美国BD、火花、MD)半胱氨酸盐酸盐水合物(10 0.05%0稀释)。我们镀100μl 10−3和10−5在预稀释100毫米琼脂板(下面列出的媒体类型)。一组媒体孵化在37°C 5天在厌氧室和另一组37°C 5%孵化有限公司23天。孵化后,殖民地从每个盘子收集通过添加1毫升BHI肉汤和刮板的表面细胞刮刀。稀释(10−3和10−5)每个媒体的结合和500μl收获殖民地被冻结在10%脱脂牛奶作为股票和500−80°Cμl用于DNA提取如下所述。
媒体本研究包括(附加文件中使用1:表S1):脑心浸液(BHI)琼脂(BD),嗨+ 0.5 g / L半胱氨酸盐酸盐水合物,10 mg / L氯高铁血红素和维生素K 1 mg / L(补充设置),嗨+ 10 mg / L硫酸粘菌素和5 mg / L naladixic酸(补充组B),嗨+补充组A和B, BHI +补充设置一个和20 mg / L庆大霉素,嗨+补充组和1%丙酸,0.2×BHI或M9最小媒体(BD) + 1 g / L菊粉之一,0.5 g / L果胶、纤维素0.5 g / L, 0.5 g / L粘蛋白,淀粉或0.5 g / L,双歧杆菌选择性的媒体(丙烯酰胺,圣路易斯,密苏里州,美国),肠道微生物群中(10),苯乙醇琼脂(BD) + 5%羊的血液(Cedarlane、加拿大),拟杆菌胆汁七叶灵琼脂(14),放线菌隔离琼脂(BD),哥伦比亚血琼脂(BD) + 5%羊的血液,粘菌素naladixic羊血琼脂(BD) + 5%,麦凯琼脂(15),巧克力琼脂(BD),大豆胰蛋白酶的琼脂(BD) + 5 g / L酵母提取物(BD) +补充设置,挑剔的厌氧菌琼脂(美国Neogen兰辛,MI)、脱氧胆酸盐琼脂(14),MacConkey琼脂(BD),卡那霉素万古霉素湖血琼脂(14)、甘露醇盐琼脂(BD),德曼Rogosa夏普琼脂(BD),和煮熟的肉羹(丙烯酰胺)+ 1.2%的琼脂。所有媒体都准备好后,制造商的指示或如前所述,除非另有提及。
实验表明,培养社区是由可行的生物(附加文件2:图S4), 100毫克的冷冻IBS1样本1:10稀释70%的异丙醇,加热到70°C 1分钟消毒(“成长”样本)或从IBS1提取的DNA样本(DNA样本)。样品被稀释10−5与BHI 0.05%半胱氨酸如上所述,培养厌氧苯乙醇琼脂(豌豆)和M9 +纤维素板1 h在37°C盘刮之前和DNA提取如下所述。
比较的新鲜和冷冻样品,一个整除立即被培养,另一个是存储在−80°C长达一年(IBS1 HV2 4个月,IBS3 1年)。比较无氧和有氧样本,在收集粪便样本划分;一分是存储在4°C与厌氧袋直到培养和其他储存在4°C没有厌氧袋(有氧样本)。
DNA提取和16 s rRNA基因测序
DNA的提取和纯化进行了如前所述[16]。短暂,500μl收获殖民地或0.1克粪便样本机械均质0.2 g的0.1毫米玻璃珠(该款,卡尔斯巴德、钙、美国;一个额外的0.2 g(2.8毫米玻璃珠被添加到粪便样本)800年μl 200毫米短茸毛4、pH值8和100年μl胍thiocyanate-EDTA -N月桂酰肌氨酸。酶裂解与50μl溶菌酶(100毫克/毫升),50μl mutanolysin (10 U /μl)和10μl核糖核酸酶(10毫克/毫升)的1 h在37°C随后添加25μl 25%十二烷基硫酸钠(SDS)、25μl蛋白酶K(20毫克/毫升)和75μl 5 M氯化钠和进一步孵化1 h 65°C。上层清液收集和DNA提取phenol-chloroform-isoamyl酒精(25:24:1;σ,圣路易斯,密苏里州,美国),进一步纯化使用DNA清洁和Concentrator-25列(美国Zymo欧文CA)按照制造商的指示。孤立的DNA是储存在−20°C。
PCR扩增的V3地区16 s rRNA基因进行如前所述[17与以下修改:一个包含1.25毫米MgCl 60μl反应2每个核苷酸的2.5毫米,每个条形码底漆的100海里,和1.25 U Taq分为3×20μl放大反应。PCR条件由最初的变性在94°C 2分钟,30 94°C的周期30年代,50°C 30年代,72°C 30年代,紧随其后的是最后一个72°C伸长10分钟。纯化PCR产品由麦克马斯特基因组测序使用Illumina公司MiSeq平台设施(汉密尔顿,加拿大)。
序列处理和分析
16 s rRNA基因序列处理完成(如前所述)(16]。总之,Illumina公司序列读取被修剪的正向和反向引物V3地区Cutadapt [18)和PANDAseq paired-end序列的结合在一起19]。他们用辣子鸡相似使用AbundantOTU [97%20.)和分类法被分配使用核糖体数据库项目(RDP)分类器21)对绿色煤电参考数据库(2011年2月4日发布)[22)使用定量见解微生物生态学(QIIME) [23]。未赋值的辣子鸡和单件不在。culture-enrichment实验读的总数是30581472人。人文的分析实验,只有辣子鸡至少10序列读取包含并考虑培养自复制之间的阅读数量少的下面这个深度(数据没有显示)。
相对丰度分类总结,β多样性,稀疏与QIIME完成。16 s rRNA基因种系发生描绘辣子鸡从文化无关,人文,或者两种方法(图。2 b)是由修剪4 2011年2月发布的绿色煤电发展史集群相似度97%。这个发展史是修剪使用QIIME和可视化使用GraPhlAn v0.9.7 [24]。
Lachnospiraceae分离和鉴定
新鲜HV7和IBS4收集粪便样本培养厌氧BHI + 1 g / L菊粉(BHI +犬)和煮肉琼脂(牛肉)。我们有146个殖民地的纯度和单一的殖民地煮15分钟Chelex 5%溶解细胞。菌落PCR放大16 s rRNA基因进行使用8 f (5′-AGAGTTTGATCCTGGCTCAG-3′)和927 r (5′-CCGTCAATTCCTTTRAGTTT-3′)引物。PCR条件由最初的变性在94°C 2分钟,32 94°C的周期为1分钟,1分钟56°C, 72°C 2分钟,紧随其后的是最后一个72°C伸长10分钟。PCR产品被贝克曼库尔特桑格测序基因组学(丹弗斯,MA)使用8 f底漆。分类作业的隔离是由在线RDP分类器(21使用16 s rRNA)训练集10信心阈值的80%。16 s rRNA隔离比较反对的基因序列Lachnospiraceae从人类微生物组项目的共识序列(HMP)“通缉犯”名单(2014年11月18日获得)使用Megablast Geneious v5.6.4 [25]。
Lachnospiraceae种系发生
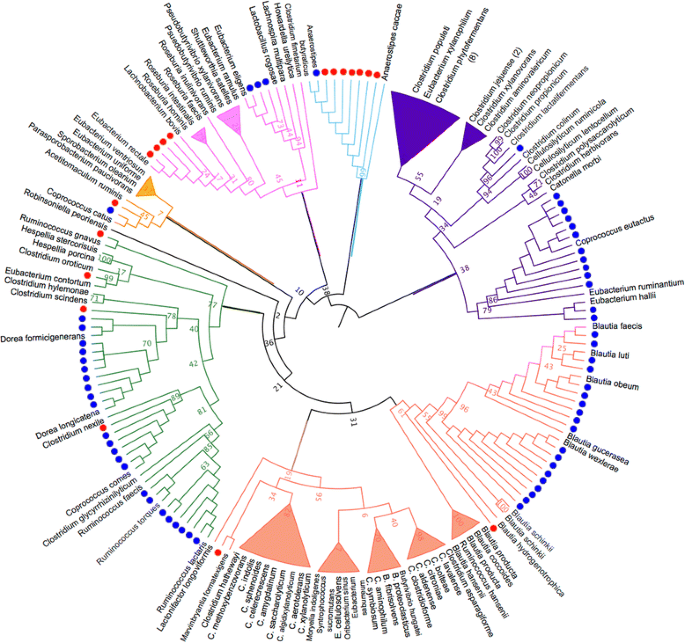
107年的16 s rRNA序列Lachnospiraceae从RDP类型隔离了21)(2014年11月12日)使用肌肉v3.8.31[对齐26)(埃德加,2004)。jModelTest v2.1.1 [27)是用来确定数据最适当地符合广义倒流(GTR)模型不变的网站和一个离散伽马分布。这些隔离的最大似然分子系统学是获得使用raxmlGUI v1.3.1 [28)实现RAxML v8.0.26 [29日]100引导程序复制。pplacer v1.1 [30.)是用于添加79年16 s rRNA基因序列的讲究的Lachnospiraceae隔离,如上所述。由此产生的发展史是无花果树可视化和中点扎根使用v1.4.0 (31日]。
结果
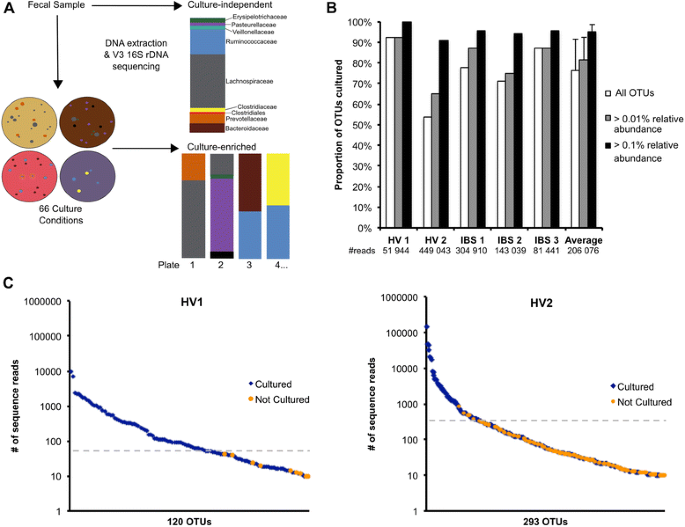
在这项研究中,人类肠道微生物群的可耕种的比例确定粪便样本的两个健康的捐赠者和三个肠易激综合征(IBS)患者。IBS样本包括自可培养细菌社区可能会影响到健康状况和肠易激综合症是一种常见的肠道疾病,肠道微生物组成的改变,涉及疾病的发病机理32]。使用人文分子分析方法(12),厌氧粪便样本使用33媒体和孵化的盘子厌氧培养和耗氧导致66年文化条件(无花果。1)。使用的媒体是由商用组件,不需要复杂的特性,例如瘤胃液体或粪便提取物用于先前的研究[8,33),根据以往的奥运会选择分子剖析研究,与其他媒体报道的具体培养肠道微生物群,包括拟杆菌胆汁七叶灵,双歧杆菌选择性的媒体,肠道微生物群中(10,添加益生元,耐碳水化合物(33)(附加文件1:表S1)。的可耕种的部分粪便微生物群是由池所有殖民地从每种类型的媒体(每粪便样本)和执行16 s rRNA基因测序在每个单独板池,称为人文分子分析。汇集了殖民地的一部分是留给未来细菌隔离。因为相同的DNA提取和测序方法被用于培养板池和粪便样本,辣子鸡的文化可以直接与文化无关的辣子鸡测序相比原来的粪便样本,以确定哪些辣子鸡培养在每种类型的媒介。
人文分子分析检测粪便样本中大部分辣子鸡
平均76%的辣子鸡观察文化无关测序的粪便样本也发现了culture-enrichment(无花果。1 b)。辣子鸡培养的最大比例为93%的健康志愿者1 (HV1)示例。没有观察到明显的区别和肠易激综合症之间的比例的辣子鸡培养健康的样品(平均79和73%的辣子鸡是培养,分别)。使用阈值的0.1%和0.01(文化无关的测序),平均81和95%的辣子鸡是培养,分别(无花果。1 b),这表明人文丰富的分子分析可以发现大多数粪便社区的成员。比较几种OTU挑选方法,包括AbundantOTU [20.]和UCLUST [34)对不同参考数据库,导致类似的比例培养对于每一个样本,表明这些结果强劲OTU集群(附加文件的方法2:图S1)。然而,辣子鸡培养的比例是影响文化无关序列深度;随着文化无关序列读取次数的增加,辣子鸡培养减少的百分比(附加文件2:图S2)。浅测序深度(< 5000年文化无关测序读),用于以前的研究(10),人为地抬高培养的微生物群的比例由于不完整的文化无关粪便样本的抽样。对于每一个样本,排名,丰富文化无关的辣子鸡测序显示几个高度丰富的辣子鸡和长尾分布较低的丰富的辣子鸡,如预期的那样在人类肠道微生物群社区(35(图。1 c;额外的文件2:图S3)。0.1%以上的相对丰度,从HV1辣子鸡都恢复了文化和只有五个辣子鸡HV2没有培养(无花果。1 c)。无教养的辣子鸡是低丰度(相对丰度< 0.8%)文化无关的测序。十二个辣子鸡相对丰度大,0.1%没有培养的捐助,包括样品蓝藻,梭状芽胞杆菌,柔膜细菌,拟杆菌门。然而,这12个辣子鸡的9其他粪便样本中检测到的文化。
一些细菌属出席丰富文化无关测序,但不到1%恢复了文化在更高的丰度在盘子里(附加文件2:图S4e和S5),从而证明文化恢复低丰度的优势细菌从胃肠道微生物群。比较每个板块社区文化无关的粪便样本显示,没有一个媒体准确地代表粪便样品的组成,表明辣子鸡检测到文化代表可行的生物,而不是DNA沉积在板块(附加文件2:图S4a-d)。为了进一步证明这一点,消毒粪便样本从IBS1捐赠者和DNA提取相同的样本被镀在两个媒体,代表培养社区大多数相似和不同粪便样本的组成(附加文件2:图S4b-d)。成长和DNA的组成控制非常不同于粪便样本或池板。事实上,负增长控制两种类型的媒体最相似,提供证据表明16 s测序的盘子代表可行的细胞坏死细胞,而不是DNA或沉积的粪便样本。古德曼et al。10]曾表明,测序汇集殖民地培养的粪便样本稀释10000倍导致不到2%的序列被来自不可行的细胞。
最小条件culture-enrichment反映了inter-individual人类肠道微生物群的异质性
每个媒体的培养社区样本之间的不同,反映出inter-individual异质性的微生物群3];因此,最小集的媒体捕捉辣子鸡的多数在所有样品很难预测,类似于之前发布的结果呼吸道微生物(12]。通过确定所需的最低文化条件恢复所有可耕种的辣子鸡出席超过0.1%在粪便样本,我们观察到的文化条件不同对所有五个粪便样本(附加文件2:图S6)。有趣的是,23日33厌氧培养条件需要在五个样本,表明大部分的媒体使用冗余,和额外的粪便样本可能需要使用其他媒体。有明显的差异从厌氧细菌社区孵化与有氧增长(附加文件2:图S5);埃希氏杆菌属是最丰富的OTU大部分有氧媒体未来的研究可以减少使用有氧条件。
培养社区受到粪便样本的储存条件
以来的直接培养厌氧粪便样本可能并不总是可行的,我们确定了影响冻结的−80°C或暴露于环境氧可耕种的粪便社区。新鲜、厌氧粪便样本被培养为以前和人文概况比较与粪便样本,冷冻或暴露于氧气(附加文件2:图S7a-e)。所有条件测试表明,培养微生物组剖面不同冻结后,与Bray-Curtis不同距离大于0.3(附加文件2:图S7a, d),而粪便样本差异影响有氧曝光,Bray-Curtis不同距离从0.15到0.65(附加文件2:图S7b, c, e)。因为粪便样本的组成不同,每个媒介选择用于不同的细菌,没有丰富的细菌群体确定为持续改变后存储。最佳复苏所需的细菌的粪便样本收集和镀后立即保持厌氧没有冻结,暴露在氧气和存储在−80°C改变可耕种的社区。
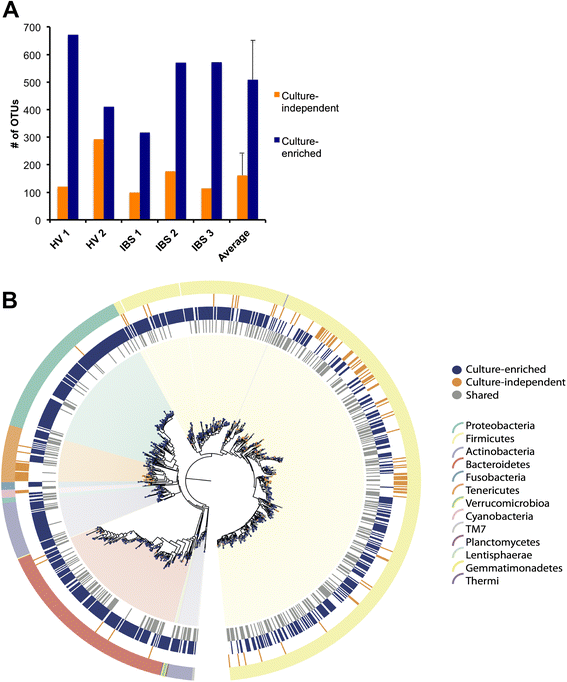
人文分子分析捕获细菌多样性大于文化无关序列
重要的是,我们观察到culture-enrichment恢复比文化无关测序辣子鸡。考虑到所有辣子鸡五个样本中发现两种方法,1051年辣子鸡(总数的67%)被culture-enrichment只发现,390年辣子鸡(25%)被发现的方法,和118年辣子鸡(8%)发现只有文化无关测序。对于每一个样本,辣子鸡是观察到人文测序比文化无关序列(图2)。这表明即使有深度测序的粪便样本(平均深度= 206 076读),检测细菌多样性文化与文化无关的方法。检测到的辣子鸡只有文化主要是分布在6个类群:放线菌,拟杆菌门,厚壁菌门,Fusobacteria,变形菌门,Tenericutes。这些低丰度的生物体可能包括结肠粘膜的细菌和其他网站在胃肠道,他们可能出现在更高的地方丰富但在过低丰度在凳子上探测到在这项研究中使用的文化无关测序深度。辣子鸡,文化只能或两种方法探测到均匀分布在细菌分类学(无花果。2 b),这表明培养辣子鸡代表粪便微生物群的成员,而不是板污染。无教养的辣子鸡包括Tenericutes分支和几厚壁菌门演化支。
有针对性的培养的Lachnospiraceae隔离
人文分子分析应用于额外的健康(HV3)和肠易激综合症(IBS4)粪便样品的有针对性的培养Lachnospiraceae隔离证明细菌群感兴趣可以从粪便样本分离,细菌成分不清楚时,利用奥运会的结果分析之前的样品(附加文件2:图S5)。有广泛的Lachnospiraceae多样性的人类肠道微生物群但这普遍和丰富的家庭差由参考基因组(36]。从分析人文分析之前的粪便样本,它是确定社区种植在厌氧BHI +犬和牛肉琼脂是主导Lachnospiraceae(附加文件2:图S5)。与我们的culture-enrichment结果一致,菊粉可以增加butyrate-producing细菌的增长,包括Lachnospiraceae体内,(37和体外38]。HV3和BHI IBS4粪便样本培养厌氧+犬和牛肉和殖民地孤立的(相对于板池)。桑格16 s rRNA基因的测序146孤立殖民地导致79年的识别Lachnospiraceae隔离,包括属的代表Blautia,Marvinbryantia,瘤胃球菌属,Dorea,真细菌,Anaerostipes,梭状芽胞杆菌,Coprococcus(附加文件3:表S2)。此外,18隔离有不到97%的相似性核糖体数据库项目(RDP)参考隔离在属级(21),系统支持的距离107年RDP隔离Lachnospiraceae参考序列(图。3)。这些18隔离可能是小说物种目前所代表的RDP类型隔离。比较的16 s rRNA基因序列Lachnospiraceae隔离的人类微生物组计划(HMP)通缉类群导致匹配到12序列有100%的身份,包括四个生物体中优先级(附加文件4:表S3)。“通缉”类群代表无教养的但丰富的和普遍的生物高分子聚合物16 s rRNA基因测序数据集(39]。隔离小说的代表肠道微生物,这些生物的全基因组测序将关键信息添加到参考基因组集合并导致更准确的作业对宏基因组,转录组和蛋白质组学的研究。
讨论
尽管其他研究已经试图文化肠道微生物群利用恒化器等复杂的方法(40)和微流体设备(41),我们表明,使用广泛的文化条件和简单的琼脂板导致细菌在肠道微生物群的全面培养。使用显著减少和复杂的文化条件不如Lagier et al。8),我们仍能捕捉到大部分的辣子鸡文化无关测序。事实上,我们相信,这是第一个研究表明> 50%的辣子鸡的粪便样本可以恢复文化。我们还表明,文化揭示细菌多样性大于被文化无关序列。这些生物、缺失或弱势文化无关的分析可能导致主机表型方法来检测和隔离这人口未来的研究至关重要。我们没有观察到不同比例的辣子鸡、肠易激综合症之间可以培养健康的粪便样本;然而,样本容量小,应该进行进一步的研究来证实这一点。从人文应用数据生成的分子分析在本研究的第一部分,我们能够进行有针对性的隔离Lachnospiraceae。这导致18物种的恢复不仅代表一个参考数据库从培养两个粪便样本两种媒体条件下,证明了文化是有价值的访问小说肠道微生物群的成员。
尽管大多数粪便辣子鸡是培养在这项研究中,一些辣子鸡、可能包含一种以上的病毒株可能已经错过了文化;宏基因组可以逐步解决该问题。一些culture-enrichment辣子鸡是只发现和文化无关测序,表明这两种方法是互补的研究肠道微生物群自组合比方法更敏感。的无教养的辣子鸡可能已经错过了由于以下原因:随机抽样在示例中,如果细菌是罕见的DNA可能起源于死细胞,增长缓慢的生物可能已经击败了挑剔的社区成员,或者在这项研究中使用的媒体可能是失踪的增长或预防增长所需营养细菌对生存至关重要的合作伙伴。这也可以解释为什么一些辣子鸡没有被培养在一个粪便样本,但从另一个捐赠者中恢复过来。
与先前的研究一致(8,11),我们观察到许多辣子鸡,只有检测到文化。某些细菌群体可能无法在引物用于16 s测序复杂的社区,如大便,因为可怜的底漆对齐会导致这些序列被淘汰在混合社区DNA聚合酶链反应(36]。然而,这些细菌可以使用选择性生长的分子分析自媒体和随后被人文可能会有更少的竞争DNA的16 s rDNA放大板池。之前报道,细菌在< 106细胞每克粪便可能错过的测序深度用于大多数微生物研究[8),但不丰富的细菌生长能力的媒体会增加恢复的DNA测序这些罕见的成员的人口。此外,粪便细菌孢子的DNA可能弱势16 s测序,根据DNA提取的方法(42),但萌发孢子在文化将有助于提高检测孢子形成的人口。
人文分子分析特定的细菌群体的针对性的复苏将是一个强大的工具研究肠道微生物群的利益由于细菌可以恢复后测序显示媒体支持其发展,为代表的额外文件2:图S5。此外,由于该方法允许板上的细菌群落的发展,它并不排除syntrophic交互增长可能需要一些细菌的43]。此外,板的后续培养池使用的抗生素和其他选择媒体组合组件可用于开发特定的细菌更精致的文化条件(11]。应用这种方法对小说的代表人类肠道微生物群的复苏将添加到引用数据库和导致更精确的分配未来的“使”研究。促进微生物领域以外的相关调查和测试假设产生文化无关的研究,关键是隔离细菌从肠道微生物群的利益、人文分子分析允许这样的隔离。
结论
我们证明大部分的人类肠道微生物群可以捕捉到人文分析,这种方法可以应用于特定细菌的复苏组。这种方法还强调了文化能力揭示多样性恢复细菌丰度低,这可能是未被充分代表的其他方法。孤立的细菌将使我们探索细菌的治疗潜力产品可能直接影响主机或微生物群落。进入细菌的人工培养的微生物群将提供详细的功能描述,并将促进生物活性的发现在细菌和inter-bacterial交互在健康和疾病。
缩写
牛肉,煮肉琼脂;嗨,脑心浸液琼脂;胃肠道、肠胃;高分子聚合物,人类微生物组计划;高压、健康志愿者;肠易激综合症、肠易激综合征;OTU操作分类单位;QIIME,定量了解微生物生态学;RDP核糖体数据库项目。
引用
Bik EM Eckburg PB,伯恩斯坦CN, Purdom E, Dethlefsen L,尹浩然,萨金特。人类肠道微生物菌群的多样性。科学。2005;308:1635-8。
Hayashi H, Sakamoto M, Benno y的系统发育分析人类肠道微生物群使用16 s rDNA克隆库和严格厌氧培养方法。Microbiol Immunol。2002; 46:535-48。
人类微生物组项目财团。结构、功能和人类健康的微生物多样性。大自然。2012;486:207-14。
卡莫迪大卫L,莫里斯CF, RN, Gootenberg DB,按钮我,沃尔夫,等。饮食迅速地改变了人类肠道微生物组。大自然。2014;505:559 - 63。
Dethlefsen L Relman哒。不完全恢复和个性化的响应人类的远端肠道微生物群反复抗生素扰动。《美国国家科学院刊S a . 2011; 108:4554 - 61。
Gevers D, Kugathasan年代,Denson L, Vazquez-Baeza Y, Van Treuren W,任B, et al。最近诊断为克罗恩病的首次治疗微生物。细胞微生物。2014;15:382 - 92。
Rajilić-StojanovićM,比亚吉E, Heilig HGHJ, Kajander K, Kekkonen R,蒂姆斯年代、et al。全球和深刻的分子分析微生物群签名在肠易激综合症患者的粪便样本。杂志。2011;141:1792 - 801。
Lagier JC, Armougom F,万米,Hugon P, Pagnier我,罗伯特·C et al。微生物文化组学:人类肠道微生物研究的范式转变。Microbiol感染。2012;18:1185 - 93。
大梁恐鸟。促进肠道微生物研究使用栽培。当今Microbiol 2015; 27:127-32。
古德曼,Kallstrom F,信仰JJ,雷耶斯,摩尔,Dantas本人交出密码G, et AL。广泛的个人人类肠道微生物群文化集合的特点和操作在无菌的老鼠。《美国国家科学院刊S a . 2011; 108:6252-7。
Rettedal EA, Gumpert H,大梁恐鸟。Cultivation-based多元人类肠道微生物群的表型出现允许目标恢复以前未耕作的细菌。Nat Commun。2014; 5:4714。
Sibley CD, Grinwis我,TR, Eshaghurshan CS,法里亚毫米,多德,et al .文化丰富囊性纤维化呼吸道微生物的分子分析。《公共科学图书馆•综合》。2011;6:e22702。
Drossman d .胃肠功能紊乱和罗马III的过程。杂志。2006;130:1377 - 90。
阿特拉斯r .微生物媒体的手册。波卡拉顿:CRC出版社;2004年。
Sibley CD, Grinwis我、字段TR -帕金斯博士Norgaard JC,练习刀功DB, et al。麦凯琼脂使常规量化“链球菌milleri”小组的囊性纤维化患者。J地中海Microbiol。2010; 59:534-40。
惠兰FJ Verschoor CP,斯登JC,罗西L, Luinstra K,尹浩然,勒布。地形的损失在上呼吸道的微生物群落。安Thorac Soc。2014; 11:513-21。
正义与发展党,巴特拉姆(merrill Lynch)医学博士斯登JC, Moreno-Hagelsieb G,这本书JD。代multimillion-sequence 16 s rRNA基因库从复杂的微生物群落组装paired-end illumina公司读取。:环境Microbiol。2011; 77:3846-52。
马丁·m·Cutadapt删除适配器从高通量测序序列读取。EMBnet j . 2011; 17:10-2。
巴特拉姆Masella美联社,正义与发展党,Truszkowski JM,布朗DG,这本书JD。PANDAseq: paired-end汇编illumina公司序列。BMC Bioinform。2012; 13:31。
你们y识别和量化的丰富物种pyrosequences对齐16 s rRNA的共识。Proc IEEE国际会议Bioinform生物医学。2011;2010:153-7。
嘉里蒂王Q,通用,Tiedje JM,科尔JR .朴素贝叶斯分类器核糖体rna序列的快速分配到新的细菌分类。:环境Microbiol。2007; 73:5261-7。
DeSantis TZ, Hugenholtz P,拉森N,罗哈斯M,布罗迪埃尔,凯勒K, et al。绿色煤电,chimera-checked 16 s rRNA基因数据库和工作台与ARB兼容。:环境Microbiol。2006; 72:5069 - 72。
Kuczynski Caporaso詹,J, Stombaugh J, bitting K,布什曼FD,科斯特洛埃克,et al . QIIME允许社区高通量测序数据的分析。Nat方法。2010;7:335-6。
Asnicar F, G Weingart,逗TL, Huttenhower C, Segata n紧凑与GraPhlAn系统数据和元数据的图形表示形式。同行j . 2015; 3: e1029。
基尔斯M, Moir R,威尔逊,Stones-Havas年代,张M, Sturrock年代,et al . Geneious基础:一个集成的和可扩展的桌面软件平台的组织和分析序列数据。Bioinform。2012; 28:1647-9。
埃德加钢筋混凝土。肌肉:多重序列比对和高准确性和高吞吐量。核酸研究》2004;32:1792-7。
小波d jModelTest:平均系统模型。另一个星球杂志。2008;25:1253-6。
Silvestro D, RAxML旧事。raxmlGUI:一个图形化的前端。Org潜水员另一个星球。2012;12:335-7。
Stamatakis RAxML version 8:系统发育分析的工具和大型的发展史后分析。Bioinform。2014; 30:1312-3。
Matsen FA Kodner RB,时常要大肠pplacer:线性时间序列的最大似然和贝叶斯系统布置在一个固定的参考树。BMC Bioinform。2010; 11:538。
兰姆伯特,德拉蒙德,无花果树A v1.3.1:树图绘图工具。2009年。http://tree.bio.ed.ac.uk/software/figtree/。
柯林斯SM。IBS的肠道微生物群作用。Nat牧师杂志。2014;11:497 - 505。
金BS,金约Cerniglia CE。在体外培养条件对维持人类胃肠道微生物群的一个复杂的人口。J生物科技生物医学》。2011;2011:838040。
埃德加钢筋混凝土。搜索和集群数量级的速度比爆炸。Bioinform。2010; 26:2460-1。
高周Y, H, Mihindukulasuriya KA, La Rosa PS,威利公里,Vishnivetskaya T, et al。生物地理学的生态系统健康的人体。基因组医学杂志。2013;14:R1。
集团引进财团人类微生物组项目工作组数据生成。评估16 s rDNA-Based社区为人类微生物组研究分析。《公共科学图书馆•综合》。2012;7:e39315。
Van den Abbeele P,杰拉德P, Rabot年代,Bruneau, El之后,尹浩然,Derrien。阿糖基木聚糖和菊粉不同调制的粘膜和腔的肠道微生物群mucin-degradation人性化的老鼠。环境Microbiol。2011; 13:2667 - 80。
马丁·斯科特•KP JC,邓肯SH,弗林特HJ。生命起源以前的刺激人类结肠butyrate-producing细菌和双歧杆菌体外。《生态。2014;87:30-40。
Fodor AA,德桑蒂斯TZ,威利公里,獾JH, Y,赫本T, et al。“通缉”从人类全基因组测序的微生物类群。《公共科学图书馆•综合》。2012;7:e41294。
是的,麦当劳Schroeter K,富恩特斯年代:Heikamp-Dejong我Khursigara厘米,德·沃斯·WM et al .评价微生物群落的重现性、稳定性和组成在人类远端肠道恒化器模型。J Microbiol方法。2012;95:167 - 74。
马L,金正日J, Hatzenpichler R, Karymov妈,休伯特N,哈难IM, et al .靶向微流控的隔离种植验证人类微生物组计划中列出的肠道细菌最希望类群。《美国国家科学院刊S a . 2014; 111:9768 - 73。
Kuske CR、Banton KL Adorada DL,鲜明的PC,希尔KK,杰克逊PJ。小规模的DNA样品制备方法领域PCR检测微生物细胞和孢子在土壤中。:环境Microbiol。1998; 64:2463 - 72。
Kaeberlein T,刘易斯K,爱泼斯坦党卫军。孤立“uncultivable”在纯培养微生物在一个模拟的自然环境。科学。2002;296:1127-9。
确认
我们要感谢M.I. Pinto-Sanchez招聘IBS患者在这项研究中关于手稿和大肠一直有用的建议。
资金
这项工作是支持由克罗恩氏及结肠炎加拿大授予MGS,和加拿大卫生研究院研究MGS, PB, SMC。出来作为一个跨学科的加拿大研究主席支持微生物的研究。
可用性的数据和材料
数据集(16 s rRNA序列)支持本文的结论已经存入顺序读取存档(SRA) PRJNA321331 BioProject ID。
作者的贡献
韩国乐团和IH进行实验。志愿者和病人样本提供的背影,SCM, PB。数据分析是由韩国乐团,FJW出来。韩国乐团、PB、设计研究和写的手稿。所有作者阅读和批准最终的手稿。
相互竞争的利益
作者宣称没有利益冲突。
伦理批准和同意参与
所有参与者入学前签署知情同意。本研究通过汉密尔顿综合研究伦理委员会(REB # 11 - 445)。这项研究符合《赫尔辛基宣言》。
作者信息
作者和联系
相应的作者
附加文件
额外的文件1:表S1。
媒体列表用于奥运会的分子分析。(XLSX 30 kb)
额外的文件2:图S1。
比较的影响OTU挑选方法辣子鸡培养的比例。图S2的测序深度的影响比较辣子鸡培养的比例。图S3培养和无教养的辣子鸡排名通过丰富IBS1-3样本。图S4比较培养社区与文化无关粪便样本的测序。图S5热图的家庭层次类群丰度在人文和文化无关测序。图S6文化条件来捕捉最丰富的辣子鸡粪便样本图S7培养新鲜粪便样本与冷冻样品和厌氧粪便样本与有氧样本。多克斯(2200 kb)
额外的文件3:表S2。
的分类Lachnospiraceae分离培养从捐赠者HV7 IBS4粪便样本。(XLSX 38 kb)
额外的文件4:表S3。
HMP通缉的匹配Lachnospiraceae隔离从HV7 IBS4样本。(XLSX 59 kb)
权利和权限
开放获取本文是分布式根据创作共用署名4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),允许无限制的使用、分配和复制在任何媒介,你提供给适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。知识共享公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于数据可用在这篇文章中,除非另有说明。
关于这篇文章
引用这篇文章
刘,j.t,Whelan, F.J., Herath, I.et al。捕捉人类肠道微生物群的多样性通过人文分子分析。基因组医学872 (2016)。https://doi.org/10.1186/s13073 - 016 - 0327 - 7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13073 - 016 - 0327 - 7
关键字
- 肠易激综合症
- 粪便样本
- 粘菌素
- 核糖体数据库项目
- 粪便微生物群