文摘
背景
炎症性肠病(IBD)的特点是慢性炎症的胃肠道与肠道微生物的变化有关。在这里,我们试图确定毒株特异性功能与IBD相关的结果。
方法
我们执行宏基因组测序的每月的粪便样本,从20 IBD患者和12控制总样本(266)。这些是分类学的异形MetaPhlAn2和功能使用HUMAnN2异形。丰富的物种被确定使用MaAsLin和不同毒株特异性pangenome使用PanPhlAn单进行了分析。
结果
我们发现明显高于丰富的患者可以忍受的兼性厌氧菌增加肠道炎症性肠病的氧化应激。我们还发现戏剧性,然而瞬态,花朵瘤胃球菌属gnavus在IBD患者中,常与疾病活动增加共病。我们确定了两个截然不同的演化支r . gnavus菌株,其中一个是丰富IBD患者。研究功能差异这两个演化支,我们增强了r . gnavuspangenome通过测序九隔离IBD患者。我们确认199 IBD-specific,毒株特异性基因参与氧化应激反应,附着力,iron-acquisition和粘液利用率,可能赋予一种自适应的优势r . gnavus肠道炎症性肠病的进化枝。
结论
这项研究增加了进一步的证据的假设增加氧化应激可能是一个主要因素塑造肠道菌群的失调在IBD和暗示r . gnavus可能是一个重要的成员改变肠道社区在炎症性肠病。
背景
炎症性肠病(IBD)是一种慢性炎性疾病,胃肠道与两个主要临床表现:克罗恩病(CD)和溃疡性结肠炎(UC)。基因易感个体IBD的发病机制可能涉及免疫反应的过度肠道微生物组(1]。肠道微环境的变化可能有助于改变肠道微生物群落组成在炎症性肠病。IBD的炎症是增加代活性氧(ROS)和活性氮物种(RNS),导致氧化应激对宿主细胞和肠道微生物组(2,3,4]。此外,肠道微生物在IBD显示失调表现为增加丰富的功能通路参与微生物氧化应激反应(5]。然而,物种和/或微生物菌株造成这一功能尚不清楚。
可用性的增加整体metagenome排序,而不是16 s rRNA基因测序,提供了机会来研究微生物的分类组成以前所未有的决议,允许strain-level和功能分析。此外,一些计算工具最近启用宏基因组测序数据的高分辨率研究中的特定毒株变异丰富物种(6,7]。越来越清楚的是,应变变化可能导致重要的生理和功能的差异如何微生物与宿主(8]。例如,同一物种的不同菌株可以引起不同的宿主免疫反应(8),和在IBD的背景下,分析大肠杆菌显示一个特定附着入侵的浓缩大肠杆菌(AIEC)菌株在炎症性肠病与健康个体9]。
炎症性肠病的特点是复发的炎症缓解期,强调跟踪病人和他们的微生物的重要性。因为暂时的炎症性肠病的症状不同,重要的是收集多个样本每个病人了解IBD的改变肠道微生物组。此外,健康人的纵向研究揭示了大型inter-subject变化在肠道微生物组(10,11,12),表明的重要性理解IBD跨多个个人针对疾病的影响。一个明显的研究在纵向IBD样品进行宏基因组测序(13]。最近开发的工具,现在可以分析大规模,纵向,宏基因组群研究IBD肠道微生物组在应变级别。
在这项研究中,我们的目标是解决炎症性肠病的两个突出问题:1)如何氧化应激在IBD形状的肠道微生物物种的组成和应变水平?2)IBD-associated菌株有特定的基因适应性提高IBD肠道健康吗?我们使用了宏基因组测序的纵向队列分析功能被微生物物种他们容忍氧化应激的能力,我们发现增加了大量的兼性厌氧生物物种IBD的直觉。我们发现戏剧性,瞬态花朵的相对丰度瘤胃球菌属gnavus在炎症性肠病,常与疾病活动增加相应。我们进一步实验aerotolerance的特点r . gnavus确定一个r . gnavus进化枝中丰富的IBD患者和有不同功能的曲目包括基因只出现在这个进化枝。这些基因通常涉及的函数可以提高殖民IBD的肠道,包括氧化应激反应、附着力、铁收购,粘液的利用率。这项研究强调了strain-level的重要性分析,揭示IBD-specific分类特性和它们的功能。
方法
样品收集
棱镜的研究样本,收集在马萨诸塞州综合医院
棱镜组的子集被选中进行纵向分析。总共15 IBD例(9 CD、5加州大学,一个不确定的结肠炎)进入纵向粪便研究(LSS)。三个参与者与胃肠道症状,检测结果为阴性炎症性肠病包括控制人口。登记在这项研究并不影响治疗。每月收集粪便样本,长达12个月。治疗开始后的第一个提到的粪便样本。全面收集临床数据为每个参与者在每个访问。在每个集合的一个子集参与者采访来确定他们的疾病活动指数,Harvey-Bradshaw指数CD参与者和简单的临床结肠炎活动指数(SCCAI)加州大学参与者(附加文件1:表S1)。
埃默里大学收集的样本
增加参与者的数量在我们的分析中,儿科的一个子集群STiNKi被选为全metagenome测序包括与加州大学和9名健康对照组5个人。所有选定UC病例分为无治疗(14]。每月收集粪便样本大约10个月。第一个样本的参与者STiNKi队列是在治疗开始之前,和随后的治疗开始后样品。粪便收集和DNA提取方法详细肖et al。14]。
DNA和RNA隔离
棱镜的研究样本,收集在马萨诸塞州综合医院
从凳子上使用试剂盒提取的DNA AllPrep RNA / DNA迷你工具酶和机械裂解的一步。溶菌酶和蛋白酶K被添加到冰冻的凳子上按照试剂盒RNAprotect细菌试剂手册在室温下10分钟孵化而涡流每2分钟。样本resuspended RLT缓冲区和0.1毫米玻璃珠添加机械裂解珠打在一个迷你珠beater-8 BioSpec产品的同质化设定为3分钟。碎片被两个连续离心步骤以最大速度为5分钟。上层清液被转移到一家QIAshredder旋转列和均质溶解产物添加到AllPrep旋转柱DNA和RNA提取使用试剂盒协议。
埃默里大学收集的样本
总核酸提取通过Chemagic男男我和Chemagic DNA血从珀金埃尔默工具包- 96。这个工具结合了化学和机械与磁bead-based裂解净化。之前提取MSM-I, TE缓冲,溶菌酶,蛋白酶K, RLT缓冲区beta-mercaptoethanol被添加到每个粪便样本。凳子溶解产物的解决方案是涡混合。样品被放置在我单位的男男同性恋者。下面的步骤自动化在男男同性恋者即M-PVA磁珠被添加到凳子和涡旋混合溶解产物的解决方案。bead-bound总核酸当时从解决方案通过一个96 -杆磁头和洗三个酒精洗为手部缓冲区。珠子被洗在最后水洗缓冲区。最后,珠子是蘸洗脱缓冲re-suspend DNA样本的解决方案。珠子被从解决方案,使纯化TNA洗出液。 The eluate was then split into two equal volumes, one meant for DNA and the other for RNA. SUPERase-IN solution was added to the DNA samples, the reaction was cleaned up using AMPure XP SPRI beads. DNase was added to the RNA samples, and the reaction was cleaned up using AMPure XP SPRI beads. DNA samples were quantified using a fluorescence-based PicoGreen assay. RNA samples were quantified using a fluorescence-based RiboGreen assay. RNA quality was assessed via smear analysis on the Caliper LabChip GX.
粪便calprotectin量化
粪便calprotectin (FCP)为每个大便化验样本与鹰生物科学calprotectin酶联Immunoabsorbent试验(ELISA)设备使用标准协议。
验证组
数据从一个刘易斯的IBD研究et al。13)从NCBI下载SRA作为IBD验证队列。八十年人类微生物组的样本项目(HMP)被用作一个健康的验证组(15]。
全metagenome测序数据的分析
整个metagenome削减和人工读取数据被过滤使用KneadData使用缺省参数。社区组成计算MetaPhlan2 [16使用默认设置)。功能和通路组成计算与HUMANn2 [17使用UniRef90数据库默认设置)。不同使用MaAsLin丰富的物种被确定。Strain-level分析使用StrainPhlAn [7使用默认参数和PanPhlAn []6与()——min_coverage 1——left_max 1.70——right_min 0.30)。虽然strain-level分析使用StrainPhlAn是72种肠道微生物组有足够的保险,r . gnavus是唯一物种发现IBD-specific应变。
的系统发育树r . gnavus压力的计算r . gnavus标记基因SNP谱,由StrainPhlAn [7]。我们使用了phangorn R包(1)之间的距离计算所有概要文件(使用朱克斯和康托尔模型);(2)构建树使用层次聚类法(UPGMA(未加权的两组与算术平均法));(3)计算使用引导引导发展史。pml函数。发现物种丰度降低在华r . gnavus,我们把样本分为两组:≥10%的相对丰度r . gnavus,和那些相对丰度< 10%r . gnavus。然后,我们使用MaAsLin找到两组之间的差异丰富的物种。
装箱aerotolerance方法
基于MetaPhlAn2的相对丰度输出(16),我们被基于aerotolerance肠道微生物的种类。我们被分成两组,厌氧菌和兼性厌氧菌。定义微分aerotolerance垃圾箱,我们开始与组装列表(18)和扩展丰富的属我们的样品(附加文件2:表S2)。兼性厌氧菌的丰度的差异计算R使用嵌套方差分析。
生存在有氧条件下
大肠杆菌酒井法子(RIMD 0509952),r . gnavus(写明ATCC 29149)真细菌线虫(写明ATCC 3376)厌氧生长在脑心浸液媒体37°C (BD)补充维生素K1-hemin逾1%解决方案(BD),微量元素的解决方案(写明ATCC) 1%, 1%微量维生素的解决方案(写明ATCC), 5%胎牛血清(Hyclone), 1 g / L cellubiose, 1 g / L麦芽糖、果糖1 g / L和0.5 g / L半胱氨酸。支流文化稀释成1毫升新鲜的媒体和厌氧孵化3 h在37°C,此时镀稀释的文化是决定集落形成单位(CFU)。文化被搬到一个有氧健身组织培养旋转他们孵化与放松盖子37°C。采集标本后厌氧确定cfu 1 -和3 - h有氧孵化。
测序的11r . gnavus菌株
DNA库是由11所示r . gnavus菌株使用先前描述的自动微流体样品制备设备(19]。这个设备是用来减少试剂成本和实际时间。设备以细胞作为输入(< 2 uL整除要求),随后执行酶细胞溶菌作用,基因组DNA的净化,tagmentation构建DNA测序库,库清理,大小的选择,和洗脱。所有混合和洗脱步骤中执行装置,使用两层微流控架构中的阀门。DNA使用固相捕获和清理实现可逆固定化(撒)珠内的设备。tagmentation反应进行后Illumina公司Nextera协议使用Tagment DNA酶(Illumina公司),所述金et al。19]。
测序库从设备和双重编码条码引物从Broad研究所(广泛的基因组平台)。整个体积的DNA库筛选了从设备(8μL)混合10μL NEBNext高保真2 x PCR MasterMix, 1μL向前引物和1μL反向引物。我们进行了PCR条码和放大以下协议:72°C 3分钟;30年代98°C;17 98°C的周期10年代,60°C 30年代,30年代的72°C;和72°C 5分钟。编码的DNA与撒净化清理。
编码库与Quant-iT量化(ThermoFisher)和集中在平等的浓度。DNA测序进行Illlumina MiSeq(2×150循环运行)。
组装和注释的r . gnavus基因组
原始MiSeq数据与Trimmomatic削减使用参数:领导:3落后:3 SLIDINGWINDOW: 15 MINLEN: 80年,黑桃使用默认参数的数据组装和注释浪子使用缺省参数。
代的一个定制的pangenome数据库r . gnavus
只有三个r . gnavus基因组是集成到默认pangenome数据库提供PanPhlAn [6]。为了创建一个更全面的pangenome数据库,我们使用一个额外的三个参考基因组可以从NCBI以及11新组装的r . gnavus孤立的基因组。
IBD-specific基因的识别
使用覆盖率输出中每一个基因从PanPhlAn pangenome6),我们发现IBD-specific基因,基因存在于多个IBD样品大于10×累计覆盖所有样本,检测不到的所有控件的LSS和验证组,包括80份粪便从HMP基因组。我们使用非常严格的标准IBD-specific基因的鉴定。而不是只使用样本的报道r . gnavus大于1×,作为PanPhlAn是默认的,我们使用所有样本。如果基因是存在的,即使在非常低的覆盖在一个健康的示例中,它是不被认为是一个IBD-specific基因。
结果
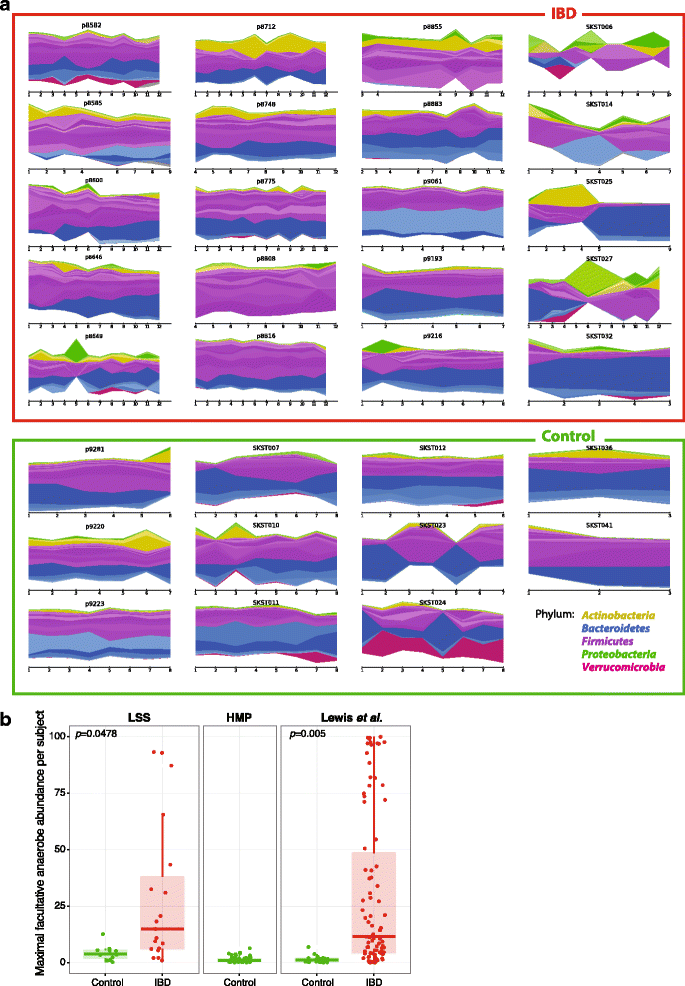
为了确定氧化应激在IBD形状的构成肠道微生物组随着时间的推移,我们生成的宏基因组测序数据纵向粪便研究(LSS)包括成年受试者的棱镜(未来的注册表在IBD研究在马萨诸塞州综合医院)队列以及埃默里大学儿科队列分析了此前使用16 s rRNA基因测序(14]。总共LSS队列包括266 20 IBD患者和12控制样本。每月收集粪便样本,长达12个月每个病人,治疗开始后的第一个提到的粪便样本。验证和支持我们的研究结果与其他炎症性肠病组,我们另外331宏基因组分析样本刘易斯et al。13),纵向微生物研究的CD患者四个时间点,0、1、4、8周。增加我们的健康对照分析,我们还研究了80元基因组样本人类微生物组计划(HMP) [15]。这些主题的概述phylum-level肠道微生物组属肠道微生物概要文件的概要文件分层显示轨迹随时间(无花果。1)。
纵向动力学微生物组成和兼性厌氧微生物概要文件在炎症性肠病。一个20个人微生物轨迹炎症性肠病(IBD)患者和11控制个人有足够的纵向数据随着时间的推移,从当前的研究(马萨诸塞州综合医院,p-identifiers)和(埃默里大学,S-identifiers)。每个主题展览一个个性化的微生物签名。每个门都有一个整体的颜色虽然属被表示为不同色调的整体颜色。b最大的相对丰度所有科目在LSS(兼性厌氧菌n= 266),刘易斯et al。(n = 368)13),和高分子聚合物(n= 80)[15军团。总的来说,兼性厌氧菌在IBD患者明显高于相对于控件(嵌套方差分析LSSp= 0.0478,刘易斯等。p= 0.005)
Aerotolerance签名IBD中发现不同丰富的物种
如何增加氧化应激的IBD肠道形状肠道微生物的组成,我们首先需要本物种的肠道微生物对氧化应激的能力(而不是通过他们的分类学分类)。基于文献综述(“方法”),我们手动封存物种分为两类:1)oxygen-utilizing生物(也称为兼性厌氧菌,它能长有或没有氧气);和(二)生物不能使用氧气作为终端电子受体(即耐氧的专性厌氧菌,在氧气都无法生长;但是,与专性厌氧菌,耐氧的细菌不是迅速受到大气氧气)(附加文件2:表S2)。
我们发现一个一致的信号增加的兼性厌氧菌大量IBD患者在LSS和刘易斯等人军团(p值= 0.0478和0.005,分别;无花果。1 b)。IBD患者LSS和刘易斯等人的军团显示的意思是兼性厌氧菌的相对丰度的19.48%和9.73,分别。相比之下,控制个人的高分子聚合物,LSS,和刘易斯等人军团的意思是兼性厌氧菌的相对丰度为1.36,1.78和1.45%,分别(无花果。1 b)。因此,炎症性肠病病人在三个不同的临床中心显示明显增加大量的兼性厌氧菌。这些兼性厌氧生物物种可能有一个健身优势相比其他厌氧健康的肠道共生的,因为他们可以容忍IBD的氧化应激增加肠道。
除了整体的兼性厌氧菌类浓缩在IBD患者中,我们发现个别类群明显富集或贫化(“方法”)。这个发现15丰富和九贫物种(q < 0.25,额外的文件3:表S3)。这些大致同意先前的研究肠道微生物的概要文件在炎症性肠病,包括损耗Clostridales在细菌性的物种(例如,Faecalibacterium prausnitzii),也观察到,例如,刘易斯et al。13]。以前的研究也发现增加大肠杆菌,肺炎克雷伯菌,韦永氏球菌属物种在IBD患者13]。当我们观察这些物种的花朵在几IBD患者趋势方向相同,这些尚不具备统计学意义在我们组和测试。包括缺乏其他差异双歧杆菌属物种在我们的研究中;这两种差异对Gevers et al。20.)队列可能是由于年龄的组合(20 - 66年在这里,3 - 17年Gevers et al。)和样本类型(大便比主要活检)之间的差异研究。
nine-identified物种显示一致的丰度差异IBD患者和控制LSS和刘易斯等人军团,八个可以解释为氧化应激的耐受性。两个兼性厌氧菌大量增加的表现IBD患者(唾液链球菌和链球菌parasanguinis),而六厌氧菌表现为炎症性肠病样本(包括丰度降低Blautia obeum和Alistipes putredinis;额外的文件3:表S3)。这些发现与以前的观测相一致的丰富物种丰富的变形菌门IBD肠道,因为这些物种是兼性厌氧菌。剩下的一个不同物种丰富,显然用氧化应激的耐受性,解释r . gnavus。尽管被分类为厌氧菌,r . gnavus在IBD患者更丰富。事实上,所有的细菌种类,r . gnavus显示最大的丰度增加IBD患者相比,控制在LSS和刘易斯等人军团(附加文件3:表S3)。
r . gnavus暂时性的主宰IBD患者的肠道微生物组
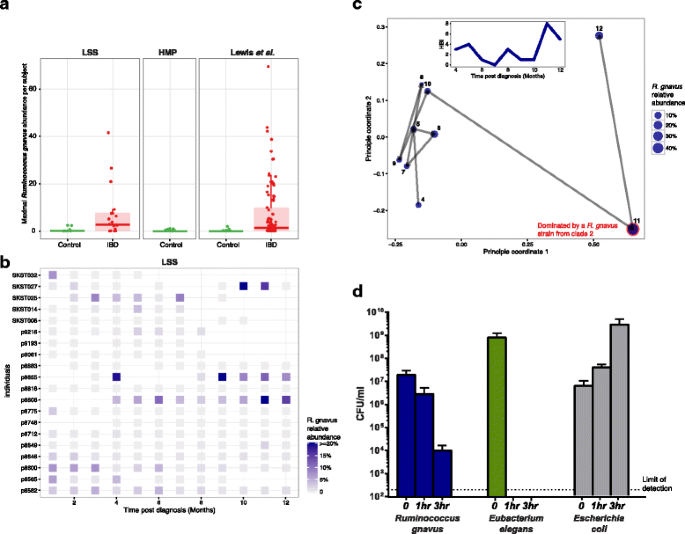
我们确定了r . gnavus花朵在8/20和26/86个人LSS和刘易斯等人的群体,分别(定义一个开花相对丰度大于5%)。r . gnavus显然是在所有不同的丰富物种支配IBD患者的肠道微生物组。最大的相对丰度r . gnavusIBD患者LSS和刘易斯等人的军团是69.5%和41.6,分别,而最大相对丰度只有2.44%在所有控件从这些军团,只有1.06%在健康HMP参与者(图。2)。
r . gnavus暂时性的主导IBD的肠道微生物组。一个最大的相对丰度r . gnavus跨样本(课程)显示所有科目在LSS (n= 266),刘易斯et al。(n= 368)和高分子聚合物(n= 80)军团。虽然大多数厌氧菌的丰度较低在IBD患者中,大量的r . gnavus在IBD患者相比,明显高于控制。b相对丰富的r . gnavus随着时间的推移,对炎症性肠病病人在LSS队列。丰富的r . gnavus不是不断高,而是在IBD瞬态增加肠道。c主坐标分析(PCoA) LSS Bray-Curtis距离了解微生物群落的IBD患者p8808超过9个月。占主导地位的r . gnavus个月4到10月12是r . gnavus进化枝1,而在11月的主要菌株r . gnavus是r . gnavus进化枝2(无花果。3)。插图显示了Harvey-Bradshaw指数(HBI)得分,临床活动性疾病和炎症的迹象,随着时间的推移这个病人。戏剧性的,瞬态的丰度增加r . gnavus11月,对应于HBI值(即增加。疾病活动)。d集落形成单位r . gnavus,真细菌线虫,大肠杆菌在0、1和3 h工作调动到大气中的氧气条件(见“方法”)。误差线代表标准偏差。虚线——检测极限。没有殖民地为专性厌氧微生物检测e .线虫在1 - 3 - h和时间点。正如所料,大肠杆菌显示生长在氧气接触,有趣的是,尽管被分类为专性厌氧微生物,r . gnavus能够容忍大气中的氧气数小时,这可以部分解释其增加丰富的氧化应激增加肠道炎症性肠病
有趣的是,丰度的增加r . gnavus在IBD患者transient-typically只生成一个或两个时间点(无花果。2 b)。这些瞬态增加病人中发现的三个不同的临床中心,因此不太可能一个地理上的异常。8808年主题LSS队列,盛开r . gnavus与峰值Harvey-Bradshaw指数(HBI),临床疾病活动,但整体相关性HBI没有达到意义(无花果。2摄氏度)。花朵的r . gnavus也与之前确认的显著下降IBD-depleted物种,包括f . prausnitzii(核反应能量= 3.04平台以及;完整列表在附加文件中4:表S4)。我们军团的纵向性质为这一发现是至关重要的,因为它是不可能确定瞬时花朵从横截面数据。
Aerotolerance分类r . gnavus
大量的增加r . gnavus令人惊讶的分类作为一个专性厌氧微生物,因为其他物种增加肠道炎症性肠病是兼性厌氧菌。因此,我们试图测试细菌的生存能力或在有氧气的存在在实验室中,我们测试了aerotolerancer . gnavus写明ATCC 29149(从健康的人类粪便分离(21])相对于一个兼性厌氧菌(大肠杆菌)和一个严格的厌氧菌,大大减少丰富在炎症性肠病(e .线虫)。我们发现,虽然无法在有氧气的存在,r . gnavus保留106可行的细胞1 h后的大气氧暴露和1043 h后可行的细胞氧的存在(见“方法”)。相比之下,e .线虫无法容忍大气中的氧气,低于我们的检测极限,即使只有1 h的接触。宽容的增加r . gnavus大气中的氧气可能导致其华在肠道炎症性肠病(无花果。二维)。
r . gnavus株代表两个系统截然不同的演化支
的存在r . gnavus在健康和控制样本,尽管在一个低得多的丰富,让我们考虑它是否有不同的功能,当出现在IBD患者的丰度高。同一种类的许多株细菌进行测序表明,不同菌株可以有截然不同的功能由于大量高度可变的附属基因。
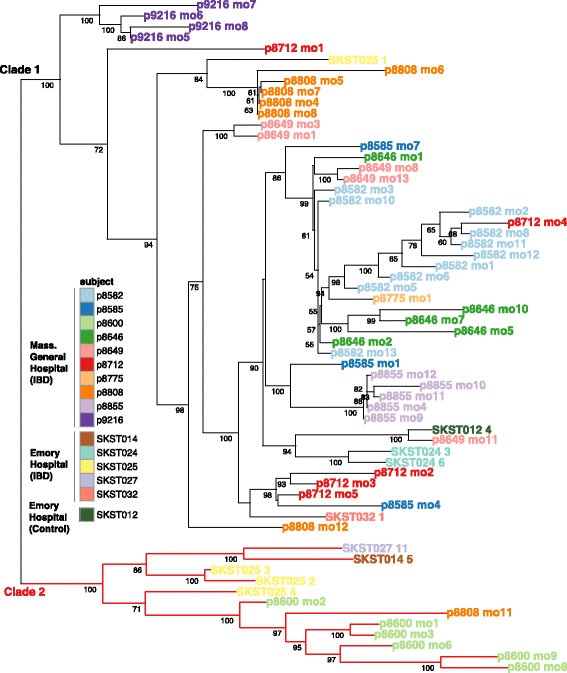
首先,我们检查是否我们可以发现不同的菌株r . gnavus在我们的样本。识别多个菌株的方法之一就是仔细看看基因注解为属于只r . gnavus基因组(表示“标记基因”)。核苷酸变化等读取映射标记基因建议这个物种的多个菌株的存在。事实上,使用单核苷酸多态性(SNP)的概要r . gnavus标记基因,我们确定了两个突出的演化支r . gnavus菌株,命名为进化枝1和分化枝2(无花果。3)。
最常见,菌株从同一个人属于同一分支,与几个例外。一个有名的例子是8808年病人的肠道微生物组个月10和11之间经历了一个转变,与增加Harvey-Bradshaw指数(HBI)的疾病。丰富的越高r . gnavus发现月11(41.6%相对丰度)不仅仅是一个扩张之前的菌株,属于进化枝1,而是绽放r . gnavus进化枝2(无花果。2摄氏度和3)。
第二,我们要检查这两个演化支之间的功能差异。我们使用了收集所有测序基因家族的参考r . gnavus基因组(表示r . gnavuspangenome)搜索基因差异存在于两个演化支,显示不同的功能潜力。执行这个分析中,我们使用PanPhlAn工具(6]。因为它的预计算pangenome数据库r . gnavus缺乏足够的代表进化枝2,我们增加pangenome覆盖进化枝2菌株的基因组测序和组装11额外的隔离r . gnavus炎症性肠病病人和婴儿(除了当前现有六个参考基因组;额外的文件5:表S5)。新r . gnavuspangenome包含11933个基因与基因组平均3117个基因。只有1178个基因被发现在所有17r . gnavus基因组,表明74%的r . gnavus基因组是变量之间的压力。变量基因组的大小范围从1708到2473个基因。
pangenome分析17所示r . gnavus参考基因组证实存在两种截然不同的演化支被他们SNP概要文件(附加文件6:图S1)。两个之前报道r . gnavus参考基因组进化枝2中存在,但无论是来自健康成人(附加文件5:表S5)。使用所有宏基因组样本LSS,刘易斯et al .,和HMP人群,我们发现r . gnavus菌株从所有控件和一些IBD患者属于进化枝1。另一方面,r . gnavus菌株IBD患者属于进化枝2。重要的是,从进化枝2株在场的病人来自三个不同的临床中心在美国,暗示这个观察的普遍性。功能特征的演化支强调三个情况下的花朵r . gnavus进化枝2(病人9216、8712和9193年)。有趣的是,r . gnavus还确定了进化枝2婴儿DIABIMMUNE队列(RJX1118 RJX1119;额外的文件6:图S1) (10丰富的]r . gnavus是大大高于健康的成年人。使用高熔点80名健康的对照,没有个人r . gnavus进化枝2被确定。综上所述,我们得出结论r . gnavus进化枝2丰富与炎症性肠病菌株与成年人。
r . gnavus进化枝2株港IBD-specific基因
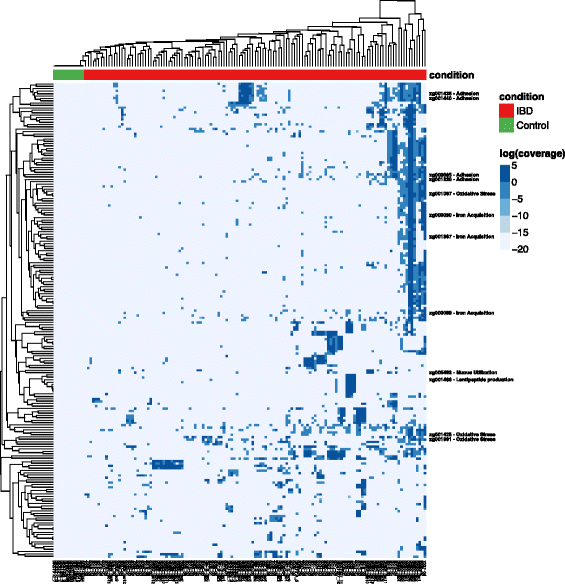
要理解每一个进化枝的功能潜力,我们寻找个人r . gnavus基因中没有检测到任何健康个体和出现在至少两个IBD样品大于10×累计覆盖。我们确定了199个这样的IBD-specific基因(图。4)。这些基因参与各种功能,可以改善殖民IBD的肠道,包括氧化应激反应,附着力,铁收购,和粘液的利用率,以及一系列其他通用函数(附加文件7:表S6)。
三个IBD-specific基因家族很可能参与氧化应激反应。两个IBD-specific基因编码的酶类和NADH氧化酶,可以作为一种强有力的过氧化物酶的蛋白质,可以提供额外的保护在肠道炎症性肠病(氧化应激22]。另一个IBD-specific基因产物,胱硫醚gamma-lyase,参与半胱氨酸的生物合成,这对谷胱甘肽的生物合成是必要的,一个重要的抗氧化剂(23]。这些结果表明,r . gnavus进化枝2株可能是专门适应容忍更高的氧化应激的IBD的直觉。
三个额外的IBD-specific基因家族参与收购铁。为几乎所有的细菌,铁是一个重要的营养,通常是一个限制营养。额外的基因参与铁收购可以自适应的殖民IBD肠道因为贫血在IBD患者中是很常见的24]。其中两个基因家族被注解为铁ABC转运蛋白和第三enterochelin酯酶,使铁螯合可用含铁细胞。
先前的研究已经报道,一些菌株r . gnavus利用粘膜聚糖。我们发现一个IBD-specific基因参与唾液酸运输,这可能提高效率的粘液利用率(25]。十三IBD-specific基因家族参与糖利用率,包括糖苷水解酶和糖转运蛋白,这可能允许利用扩大的糖作为碳源(附加文件8:表S7)。
最后,一个IBD-specific基因注释为lantipeptide合成酶参与修改肽的生物合成,其中许多有抗菌作用。的一些压力r . gnavus众所周知,生产lantipeptide ruminococcin,抑制的殖民化梭状芽胞杆菌物种(26]。Lantipeptides可以提供r . gnavus竞争优势在殖民肠道。
IBD-specific基因有可能集体适应肠道炎症性肠病的证据r . gnavus菌株。九的基因是与移动元素,如转座酶有关,噬菌体尾巴卷尺或relaxosome蛋白质表明这些基因可能是“货物基因”收购的移动遗传元素r . gnavus(附加文件8:表S7)。因此,IBD-specific基因不是“核心”的基因进化枝2,但可能已经获得和保留,因为他们提供了一个在IBD肠道微环境的适应性优势。
讨论
这里给出的结果与假设一致的主要功能失调的肠道微生物在IBD转向生物能够应付增加氧化应激(27]。重要的是,抵抗氧化应激是广泛的,但稀疏,分布在整个肠道微生物组发展史。因此不能很好地解决简单的分类装箱。微生物转向兼性厌氧菌有几个重要的炎症性肠病的病理生理学意义。我们发现,最强的,但短暂,偏差在IBD患者的微生物组成的绽放r . gnavus从平均0.1%的控制在IBD活动性疾病患者多达69%。的生理生态位r . gnavus可能是黏液溶解的从肠道黏液层(利用聚糖作为能源)。我们推测,戏剧性的变化r . gnavus丰度影响粘液的微妙的平衡层,由于显著增加社区的一部分利用黏液层。这种不均衡可以反过来影响肠道屏障的完整性,潜在的炎症性肠病病人的肠道通透性增加。
是否增加了丰富的r . gnavus在炎症性肠病是炎症的原因或结果目前未知。至少,r . gnavus是一个最好的殖民者的IBD肠道发炎。然而,也有可能r . gnavus导致或加剧了过度的免疫反应在IBD患者肠道微生物组。除了IBD的丰度增加,其他研究已经涉及r . gnavus作为一个潜在的pathobiont。3例r . gnavus菌血症的情况下感染髋关节植入物周围的骨头r . gnavus已报告(28,29日,30.]。在一项研究中关于治疗UC患者粪便微生物移植,病人从捐赠者高凳子r . gnavus更有可能复发(31日在疾病),暗示因果作用。密集采样的病人耀斑之前,在他们的治疗和缓解后将使该领域进一步研究的作用r . gnavus在炎症性肠病的疾病活动。
最初的错误分类r . gnavus,加上16 s扩增子测序了解识别的局限性,在以前的IBD研究困惑的分析,和离开的意义r . gnavus在IBD的失调被低估了。r . gnavus门的一员厚壁菌门存在于肠道的~ 90%个人(尽管在低丰度)。但其属分类最近争议。原来归类为的一员瘤胃球菌属属的摩尔et al。21),它最近被转移到Blautia基于16 s rRNA基因测序(属32,33]。具体地说,真正的瘤胃球菌属IBD患者物种丰度下降,掩盖了大量的增加r . gnavus在这些示例中,当检查只在属的水平。
结论
本研究的一个主要优点是,纵向的耦合与全metagenome测序样品让我们研究IBD前所未有的决议的肠道微生物菌株及其功能的潜力。提供的准确的物种——strain-level识别整个metagenome测序理解丰富的尤其重要r . gnavus,如16 s rRNA的基因序列Blautia物种非常相似,因此大多数16 s分析总结属Blautia。具体来说,在炎症性肠病,大量的不同Blautia物种的变化方向相反,完全屏蔽r . gnavus花朵。我们使用了宏基因组测序超越分类分析和研究各种之间的功能差异r . gnavus菌株。综上所述,我们的研究结果揭示的进化枝r . gnavus菌株可能更好的适应了IBD的肠道,通过氧化应激反应机制,附着力,铁收购,粘液的利用率。
引用
豪尔B, Gardet Xavier RJ。遗传学和炎症性肠病的发病机制。大自然。2011;474:307-17。
Balmus IM, Ciobica, Trifan Stanciu c的影响氧化应激和抗氧化治疗炎症性肠病:临床方面和动物模型。沙特杂志。2016;22:3-17。
保尔森NA,安徒生V, Møller JC, Møller HS,安F,杰森Purup年代,et al。比较分析发炎和non-inflamed结肠活检显示强大的蛋白质组学在溃疡性结肠炎患者炎症。BMC杂志。2012;12:76。
专业DN,廖J,杨G-Y,杨CS。氧化应激和溃疡性colitis-associated致癌作用:研究在人类和动物模型。致癌作用。2003;24:353 - 62。
Gevers摩根XC逗TL,索科尔H, D,提出了元KL、病房DV, et al。障碍肠道微生物组的炎症性肠病和治疗。基因组医学杂志。2012;13:R79。
朔尔茨M,病房DV, Pasolli E, Tolio T,佐弗M公司Asnicar F, et al . Strain-level微生物流行病学和人口基因组学从猎枪宏基因组。Nat方法。2016;13:435-8。
Truong DT,邰蒂,Pasolli E, Huttenhower C, Segata n .微生物strain-level人口从基因组结构和基因多样性。基因组研究》2017;27:626-38。
Geva-Zatorsky N, Sefik E,夸L, Pasman L,谭TG, Ortiz-Lopez, et al .挖掘人类肠道微生物群的免疫调节生物体。细胞。2017;168:928-43。e11。
Martinez-Medina M, Garcia-Gil LJ。大肠杆菌在慢性炎性肠道疾病:粘附侵袭性大肠杆菌致病性的更新。世界J Gastrointest Pathophysiol。2014; 5:213-27。
Yassour M, Vatanen T, Siljander H, Hamalainen a - M, Harkonen T, Ryhanen SJ,等。婴儿肠道微生物的自然历史和抗生素治疗对菌株的影响多样性和稳定性。Sci Transl医学。2016;8:343ra81。
夏博诺信仰JJ, Guruge杰,M,萨勃拉曼尼亚年代,西多夫H,古德曼,et AL。人类肠道微生物群的长期稳定。科学。2013;341:1237439。
Gevers Franzosa EA,黄K,草甸摩根富林明,D,柠檬KP, Bohannan BJM, et al .识别个人使用的微生物宏基因组编码。《美国国家科学院刊S a . 2015; 112: E2930-8。
刘易斯JD、陈EZ Baldassano RN, Otley AR,格里菲斯,李D, et al .炎症,抗生素,和饮食环境压力小儿肠道微生物组的克罗恩病。细胞微生物。2015;18:489 - 500。
肖KA,贝莎M, Hofmekler T, Chopra P, Vatanen T, Srivatsa,等。生态失调、炎症,和对治疗的反应:纵向研究的儿科学科与新诊断炎症性肠病。基因组医学。2016;8:75。
人类微生物组项目财团。结构、功能和人类健康的微生物多样性。大自然。2012;486:207-14。
Truong DT, Franzosa EA,逗TL,朔尔茨M, Weingart G, Pasolli E, et al。MetaPhlAn2增强metagenomic分类分析。Nat方法。2015;12:902-3。
Abubucker年代,Segata N, Goll J,舒伯特,伊泽德J, Cantarel提单,等。代谢重建人类微生物宏基因组数据及其应用。公共科学图书馆第一版杂志。2012;8:e1002358。
Albenberg L, Esipova电视,法官CP, bitting K,陈J,劳克林,et al .管腔内的氧梯度之间的相关性和径向分区的肠道微生物群。胃肠病学。2014;147:1055 - 63。e8。
金正日的年代,德Jonghe J, Kulesa AB,费尔德曼D, Vatanen T, Bhattacharyya RP,等。高通量自动化微流体样品准备准确的微生物基因组学。Nat Commun。2017; 8:13919。
Gevers D, Kugathasan年代,Denson洛杉矶,Vazquez-Baeza Y, Van Treuren W,任B, et al。最近诊断为克罗恩病的首次治疗微生物。细胞微生物。2014;15:382 - 92。
摩尔WEC,约翰逊JL Holdeman LV。校正类杆菌、丁酸弧菌属和描述Desulfomonas将军11月和10属Desulfomonas新物种,丁酸弧菌属、真细菌,梭状芽孢杆菌和瘤胃球菌属。Int J系统另一个星球Microbiol Microbiol Soc。1976; 26:238-52。
西山Y,梅西V,武田K,川崎,佐藤J,渡边T, et al .氢peroxide-forming属于酶类的NADH氧化酶氧化还原酶家族:在细菌生存和生理的作用。J Bacteriol。2001; 183:2431-8。
Masip L, Veeravalli K,乔治奥g .谷胱甘肽的许多面临细菌。Antioxid氧化还原信号。2006;8:753 - 62。
巴希尔Kaitha S, M,阿里·t .缺铁性贫血在炎症性肠病。世界J Gastrointest Pathophysiol。2015; 6:62 - 72。
Tailford勒,欧文CD,沃尔什J,罗斯特呃,Hardy-Goddard J, LE Gall G, et al。发现人类肠道微生物群的分子内trans-sialidases表明小说粘膜适应性机制。Nat Commun。2015; 6:7624。
Dabard J, Bridonneau C,菲利普C, Anglade P, Molle是的D, Nardi M, et al . Ruminococcin,瘤胃球菌属产生的一个新的lantibiotic gnavus应变从人类粪便分离。:环境Microbiol。2001; 67:4111-8。
Rigottier-Gois l .失调在炎症性肠病:氧气假说。ISME j . 2013; 7:1256 - 61。
汉森SGK, Skov MN, Justesen我们。两种情况的瘤胃球菌属gnavus与憩室炎相关的菌血症。中国Microbiol。2013; 51:1334-6。
Kim YJ Kang HY,汉族Y,李女士,李HJ。血液感染通过瘤胃球菌属gnavus患者胆囊穿孔。厌氧菌。2017;47:129-31。
Roux l,埃尔赛义德F, Duffiet P,鲍尔T, Heym B,盖拉德J-L, et al .瘤胃球菌属全髋关节置换术gnavus感染一个62岁的老人与溃疡性结肠炎。中国Microbiol。2015; 53:1428-30。
Fuentes年代,雷森NG, van der machteld Spek MJ,哈特曼JH, Huuskonen L, Korpela K, et al。微生物变化和签名的粪便微生物群移植后长期缓解溃疡性结肠炎。ISME j . 2017; 11 (8): 1877 - 89。
刘C, Finegold SM,歌Y,劳森。球菌样的梭状芽胞杆菌的重新分类,瘤胃球菌属hansenii,瘤胃球菌属hydrogenotrophicus,瘤胃球菌属luti,瘤胃球菌属长生蜿和瘤胃球菌属schinkii作为Blautia球菌样的将军11月,梳子。11月,Blautia hansenii梳子。11月,Blautia hydrogenotrophica梳子。11月,Blautia luti梳子。11月,Blautia有的时候梳子。11月,Blautia schinkii梳子。11月11月和描述的Blautia wexlerae sp.,分离出人类的粪便。Int J系统另一个星球Microbiol。2008; 58:1896 - 902
劳森PA, Finegold SM。重新分类的瘤胃球菌属obeum作为Blautia obeum梳子。11月Int J系统另一个星球Microbiol。2015; 65:789 - 93。
确认
我们感谢蒂芙尼Poon帮助样本数据生成和协调。我们也感谢冬青鲟鱼和伊丽莎白·安德鲁斯棱镜管理。我们感谢亨利·j·r·gnavus hai提供菌株。
资金
A.B.H.是海伦的默克公司研究员干草惠特尼的基础。'支持NIH的趋势1 k99dk113224;卡拉曼家族基金会;查尔斯·a·王相信博士后奖学金。R.J.X.和h是由国立卫生研究院授予U54DE023798,和美国的克罗恩氏和结肠炎基金会。支持h NSF资助mcb - 1453942。R.J.X.由国家卫生研究院的基金支持R01DK92405 P30DK43351。
可用性的数据和材料
在当前的研究中生成的数据集是可以从NCBI SRA BioProject下PRJNA385949。隔离下基因组可从NCBI BioProject PRJNA388337。
作者信息
作者和联系
贡献
ABH和我进行分析。ABH、我、高压、RJX和CH写的手稿。XJ,电视,和DG了生物信息学分析。AG),助教和NF进行微生物学实验。GL和PB测序r . gnavus基因组。JS RW, MC,安娜和詹MGH主题。SK和MB了埃默里科目。RJX和CH构思。ABH,我、DG、JS、高压ESL, RJX, CH方法和研究进展。英语、RJX和CH担任组长。所有作者阅读和批准最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
的设计研究是马萨诸塞州综合医院的机构审查委员会批准(协议编号2004 p001067)。获得书面知情同意参与这项研究,以及研究设计符合《赫尔辛基宣言》。
同意出版
不适用。
相互竞争的利益
DG是人类微生物研究所受雇于詹森。CH赛里斯疗法的科学顾问委员会。其余作者宣称他们没有利益冲突。
出版商的注意
施普林格自然保持中立在发表关于司法主权地图和所属机构。
附加文件
额外的文件1:表S1。
在马萨诸塞州综合医院临床样本收集的元数据。(XLSX 14 kb)
额外的文件表2 S2。
名单由aerotolerance属扔进垃圾箱。(XLSX 9 kb)
额外的文件3。表S3。
差异丰富的物种在炎症性肠病。不同丰富的物种在IBD患者相比,控制重大LSS和刘易斯等人军团(q < 0.20,使用MaAsLin)。(XLSX 14 kb)
额外的文件4:表S4。
丰富的物种在不同r . gnavus花朵。(XLSX 13 kb)
额外的文件5:表S5。
隔离所有当前可用r . gnavus基因组来源。隔离基因组测序和组装在这个手稿用星号表示。(XLSX 9 kb)
额外的文件6:图S1。
对两个不同的演化支Pangenome分析r . gnavus。Pangenome分析r . gnavus使用PanPhlAn显示的每一个基因的存在与否r . gnavuspangenome。x轴显示宏基因组以及样品r . gnavus参考基因组;y轴显示的基因簇r . gnavuspangenome。聚类结果显示两种演化支r . gnavus1,我们称之为进化枝和2进化枝。利用宏基因组样本LSS /刘易斯HMP军团,我们发现r . gnavus菌株从所有健康的成年控件和一些IBD样本功能类似r . gnavus进化枝1。另一方面,只有IBD样本r . gnavus菌株的功能类似r . gnavus进化枝2。的红色透明的矩形含有丰富的基因r . gnavus炎症性肠病组。的绿色透明矩形包含许多基因缺失的基因r . gnavus炎症性肠病组。(PDF 2396 kb)
额外的文件7:表S6。
为选定的基因注释r . gnavusIBD-specific基因中提到的主要文本。(XLSX 9 kb)
额外的文件8:S7表。
基因注释的所有199 IBD-specific基因。(XLSX 15 kb)
权利和权限
开放获取本文是分布式根据创作共用署名4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),允许无限制的使用、分配和复制在任何媒介,你提供给适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。知识共享公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于数据可用在这篇文章中,除非另有说明。
关于这篇文章
引用这篇文章
大厅,学士,Yassour, M., Sauk, J.et al。一本小说瘤胃球菌属gnavus进化枝丰富在炎症性肠病病人。基因组医学9103 (2017)。https://doi.org/10.1186/s13073 - 017 - 0490 - 5
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s13073 - 017 - 0490 - 5
关键字
- Gnavus
- 瘤胃球菌属Gnavus
- Pangenome
- MetaPhlAn
- 兼性厌氧菌