条文本
文摘
背景最近的证据表明,微生物的作用在胰腺导管腺癌(PDAC)病因学和进展。
客观的探讨粪便、唾液微生物群作为一个潜在的诊断的生物标志物。
方法我们应用猎枪宏基因组和16 s rRNA扩增子测序样品从西班牙病例对照研究(n = 136),其中57例,50控制,和29慢性胰腺炎患者在发现阶段,和德国的病例对照研究(n = 76),在验证阶段。
结果粪便宏基因组分类器比唾液进行分类和识别患者PDAC的精度0.84接受者操作特征曲线下面积(AUROC)基于一组27微生物物种,与一致的准确性在早期和晚期疾病阶段。性能进一步提高0.94 AUROC当我们结合microbiome-based预测与血清水平的碳水化合物抗原(CA)波,目前唯一的非侵入性,食品和药物管理局批准,低特异性PDAC诊断的生物标志物。此外,microbiota-based分类模型局限于PDAC-enriched物种高度特异当验证25公开宏基因组研究人口对各种健康状况(n = 5792)。microbiome-based模型有较高的预测精度验证德国人口(n = 76)。一些粪便PDAC标记种类在胰腺肿瘤检测和non-tumour组织使用16 s rRNA测序和荧光原位杂交。
结论综上所述,我们的研究结果表明,非侵入式的、健壮的和特定的粪便microbiota-based筛查PDAC的早期检测是可行的。
- 胰腺癌
- 肠道微生物
- 癌症预防
- 胰腺肿瘤
- 筛选
数据可用性声明
数据在公共、开放访问存储库。所有数据都包含在相关研究文章或作为补充信息上传。样品的原始测序数据可用在欧洲核苷酸存档(ENA)标识符PRJEB38625和PRJEB42013研究。这些样本作为补充提供的元数据表S1和S2。过滤分类和功能配置文件作为输入用于统计模拟管道在S1和S2补充数据是可用的。分析代码和结果可用https://github.com/psecekartal/PDAC.git。
这是一个开放的分布式依照创作共用署名4.0条Unported (4.0) CC许可,允许他人复制、分配、混音、转换和发展这项工作为任何目的,提供了最初的工作是正确地引用,执照的链接,并表明是否变化。看到的:https://creativecommons.org/licenses/by/4.0/。
来自Altmetric.com的统计
本研究的意义
已经知道这个问题是什么?
胰腺导管腺癌(PDAC)正在增加在世界范围内,构成高疾病负担和死亡率,但准确的,非侵入性诊断选项仍然不可用。
改变口腔,粪便和胰腺微生物组成与PDAC的风险增加有关。
有什么新发现吗?
凳子microbiota-based分类描述,预测PDAC准确性和特异性高、独立的疾病阶段,与潜在作为非侵入性诊断代理。
粪便metagenomic分类器识别PDAC精度为0.84接受者操作特征曲线下面积(AUROC)西班牙队列,根据27个物种。精度提高到0.94 AUROC结合不太特定的碳水化合物抗原(CA)波血清标志物。
分类器是一个独立的验证德国PDAC队列(0.83 AUROC),和PDAC疾病特异性被证实对25公开宏基因组研究人群与不同的健康状况(n = 5792)。
标记在粪便样本中类群丰富的存在(韦永氏球菌属,链球菌,Akkermansia),还与微分类群丰富健康和胰腺肿瘤组织(拟杆菌,乳酸菌,双歧杆菌属)是由荧光进行验证原位杂交。
本研究的意义
它会如何影响临床实践在可预见的未来吗?
粪便microbiome-based检测PDAC可能会提供一个非侵入式的、有成本效益的和健壮的PDAC早期诊断方法。
PDAC-specific微生物提供签名,包括跨组织的微生物种群之间的联系,提供小说microbiome-related假设关于疾病病因学预防和可能的治疗干预。
介绍
胰腺导管腺癌(PDAC)是最常见的胰腺癌和癌症相关死亡的主要原因,尽管发病率相对较低。1 2高致命性的PDAC是两晚的结果诊断和治疗选择有限3:症状是不具体的,经常出现只在疾病晚期阶段,此时肿瘤可以局部切除或转移性疾病。目前,PDAC使用成像诊断测试。4敏感的和负担得起的测试的早期检测PDAC可以因此改善结果。在胰腺组织中已经探讨了PDAC标记,5尿液6 7和血清。8 9然而,到目前为止,唯一的食品和药物管理局(FDA)批准PDAC生物标志物仍血清糖类抗原(CA)波。CA19-9疾病特异性有限水平可以提升其他伴随的条件(如胆道梗阻),因此主要用作PDAC监视一个标记,而不是筛查或诊断。10 - 14
PDAC复杂的病因学,建立风险因素,包括年龄、慢性胰腺炎、糖尿病、肥胖、哮喘、血型和生活方式(如吸烟和饮酒过量)。15日16这些风险因素的作用在PDAC病因学也可以补充或有时确实由微生物的变化。例如,口腔卫生不良和牙周炎与PDAC风险增加有关,17观察也延伸到牙周炎,caries-associated微生物物种。18 19这些物种的变化有时是更广泛的口腔微生物成分变化的一部分20 21或者一直探索PDAC的危险因素。22同样,肠道微生物组成第23 - 25和十二指肠,26日27日通过16 s rRNA扩增子测序量化,以前PDAC风险有关。
人类胰腺港口与口腔和肠道微生物组股票物种,25 28-32尽管它的确切成分仍然难以捉摸由于挑战与污染控制在低细菌生物样本。33在小鼠模型中,来自肠道的微生物会引起致癌胰管,25 30暗示作用的微生物PDAC最近扩展到真菌病原学和进展。34此外,胰腺肿瘤微生物也可能与疾病进展和PDAC患者的长期生存。31日
然而,这些进步的翻译为临床应用PDAC-specific微生物签名迄今仍很大程度上未被探索。这里,我们目前的强劲的识别,基于特定的微生物PDAC签名的宏基因组调查西班牙(ES)研究人口57 PDAC新诊断和首次治疗的患者,29日慢性胰腺炎(CP),患者和50匹配控制。我们采样的唾液,粪便,胰腺正常和肿瘤组织和评估微生物组合使用全基因组鸟枪宏基因组,16 s rRNA扩增子测序和荧光原位杂交(鱼)化验。最好的PDAC患者之间的歧视和non-PDAC科目通过统计模型是基于一组27粪便微生物物种可以量化诊断设置目标的方式。microbiome-based模型的预测精度确认在一个独立的德国(DE) PDAC验证人口包括44 PDAC患者和32控制和进一步加强CA19-9当结合血清水平。我们进一步验证这些模型的疾病特异性与现有的数据从25个研究9疾病的(n = 5792)。35-59几个PDAC-enriched物种也在癌组织中发现,口腔疾病和肠道的数量与可能的链接,支持他们的潜在作用在PDAC发病机理,如前所报道。25 30 31 34
方法
招聘和样本收集
应用病例对照设计。受试者预期从医院招募了2016年和2019年之间·拉蒙-卡哈尔在马德里和Vall d ' hebron医院在巴塞罗那,西班牙,使用相同的协议生物样本采集、处理和存储。学科与新诊断PDAC (n = 57) > 18岁被确定之前,任何癌症治疗。在PDAC谁被怀疑的受试者招募,取样做过任何治疗。患者慢性胰腺炎(CP, n = 29)招募了来自同一医院。控制与年龄、性别和医院选择从住院病人住院的一个主要诊断与PDAC风险因素。参与者不能参与这项研究由于物理障碍被排除在外的能力。机构审查委员会伦理批准(CEIπ26 2015 - v7)和书面知情同意获得参与中心和研究对象,分别。流行病学和生活方式训练监控在面对面的访谈收集的数据通过一个结构化的问卷。疾病的临床资料,包括阶段和后续的数据,被同一个监视器从医院检索图,同样使用结构化的问卷调查。 Recorded jaundice status was additionally confirmed and extended by direct bilirubin measurements from blood samples in CNIO, Madrid. All data were entered, edited and managed using REDCap. Missing lifestyle and medication values in the metadata (missing overall in 3.1%) were imputed using a random forest-based algorithm for missing data imputation called missForest (n=100 trees).60归责精度高据归罪误差估计(意味着out-of-bag错误= 0.12)。血清CA19-9水平分析电化学发光免疫分析法(ECLIA,罗氏诊断、德国)制造商的指示后在实验室医学研究所和Pathobiochemistry,德国马尔堡。每个样本重复化验,化验用积极控制在每个板(在线补充表S1)。
凳子和唾液(漱口水)样本保存在RNALater和储存在4°C立即12小时,然后转移到−20°C为另一个24小时,然后储存在−80°C到DNA提取。肿瘤手术期间和未受感染的组织样本收集个人的一个子集,立即在液氮病理评估被迅速冻结,不惜−80°C。所有的样品都装上干冰。
一个独立的验证人口招募的手术,埃朗根大学医院(32 PDAC和32个控制样本)和部分平移肝病,内科,歌德大学诊所,法兰克福(12 PDAC样本)使用相同的生物样本采集、协议处理和存储。选择匹配的控制从住院病人住院的一个主要诊断与PDAC风险因素。这项研究是由当地伦理委员会批准(sgi - 3 - 2019, 451 _18 B),并从参与者得到书面知情同意。临床数据,包括疾病阶段和后续数据,从医院的临床记录图表检索各自的患者(在线补充表S2)。血清CA19-9水平进行分析的常规免疫测定(罗氏诊断、德国)后,制造商的指示。粪便样本保存在OMNIgene-Gut om - 200瓶(闆Laborsysteme GmbH,德国)并立即存放在−80°C到DNA提取。
样品处理
粪便和唾液样本解冻在冰上,整除,基因组DNA提取使用试剂盒Allprep PowerFecal DNA / RNA设备根据制造商的指示(试剂盒、希尔登,德国)。从胰腺肿瘤的基因组DNA, non-tumoral组织样本提取使用试剂盒DNeasy血液和组织装备Del Castillo的协议修改等26:细胞细胞溶解机械(5毫米不锈钢珠在150年代25 Hz),紧随其后的是溶菌酶治疗(20毫克/毫升)和蛋白酶和核糖核酸酶消化2 h (56°C)。所有样本被随机分配到提取批次。占提取潜在的细菌污染,聚合酶链反应(PCR)和测序试剂盒,我们包括消极的控制与每个组织DNA提取(萃取空格)批(在线补充图1)。
16 s rRNA扩增子测序
胰腺组织DNA纯度为16 s rRNA前置放大使用引物PCR 331 f (5 ' -TCCTACGGGAGGCAGCAGT-3 ')61年和979 r (5“-GGTTCTKCGCGTTGCWTC-3”)。62年循环条件下由一个初始模板变性在98°C 2分钟,紧随其后的是30个周期98°C的变性10年代,退火20年代在65°C,在72°C扩展30年代在72°C和最后一个扩展了10分钟。其次是使用SPRIselect size-selective清理磁珠(0.8 left-sized;贝克曼库尔特、沥青、美国加州)。粪便DNA没有preamplified和唾液。
目标放大16 s rRNA V4地区(引物序列F515 5“-GTGCCAGCMGCCGCGGTAA-3”和R806 5 ' -GGACTACHVGGGTWTCTAAT-3 '),63年都使用了KAPA HiFi HotStart PCR混合(罗氏、巴塞尔、瑞士)两步编码PCR协议(NEXTflex 16 s V4 Amplicon-Seq设备;Bioo科学、美国德克萨斯州奥斯汀市)从制造商的指示进行小的修改。PCR产品汇集,纯化使用size-selective SPRIselect磁珠(0.8 left-sized)然后测序在2×250个基点的Illumina公司MiSeq (Illumina公司,圣地亚哥,加利福尼亚州,美国)的基因组学的核心设施,欧洲分子生物学实验室,海德堡。
16 s rRNA扩增子数据处理
生读质量减少,去噪和过滤使用DADA2嵌合PCR文物。64年由此产生的确切的扩增子序列变异(asv)分类学的分类和映射到一组引用的操作分类单位使用MAPseq(辣子鸡)在98%的序列相似性。65年读,不自信地映射到参考一致细菌和古细菌二级支持结构小亚基rRNA模型使用的66年和集群与98%的平均使用HPC-CLUST连杆,辣子鸡67年如前所述。68年结果,我们分类单元表获得两项决议:100%相同的asv和98% open-reference辣子鸡;除非另有指示,分析在主文本参考辣子鸡。
计数表被删除噪音过滤样品保留小于500读和类群在少于五个样品;这被读取的数据集总数的2.5%。18唾液样本,技术复制合并后确认他们强烈与社区组成。胰腺组织和肿瘤样本,asv观察到负控制样本,被读取的映射称为试剂盒污染物。33这些步骤之后,我们保留308 16 s rRNA扩增子样本143例进一步分析(130唾,118粪便,20影响胰腺组织,肿瘤组织的23与17个匹配PDAC组织样本)。
猎枪宏基因组测序
宏基因组库212粪便100份唾液样本和准备使用内超二世撒高清设备,根据反应物的浓度,与目标插入大小为350,和测序Illumina公司HiSeq 4000平台(Illumina公司,圣地亚哥,加利福尼亚州,美国)在2×150个基点paired-end设置目标深度8英镑/样本的基因组学的核心设施,欧洲分子生物学实验室,海德堡。每个样本测序数据中提供相关的git存储库(https://github.com/psecekartal/PDAC.git)。三个唾液和粪便样本,技术复制合并后确认他们在社区构成强烈相关。
Metagenome数据处理
宏基因组数据处理使用建立工作流NGLess v0.7.1。69年生读修剪质量(≥45 bp phr分数≥25)和过滤人类基因组(版本hg19,映射在贯穿≥45 bp)≥90%。结果过滤读取被映射(贯穿≥45 bp≥97%)的代表基因组5306了解基因组从proGenomes获得集群数据库v2。70年
使用莫土语分析器v2.5分类资料获得71年和过滤只保留物种观察到一个相对丰度≥10−5≥2%的样本。基因功能概要文件获得对全球microbioal基因映射目录(GMGCv1,科埃略等72年,http://gmgc.embl.de/),由蛋酒v4.5开发总结读计数73年注释同源组和KEGG模块。特性的相对丰度≥10−5≥15%的样本保留作进一步分析。
微生物数据统计分析
所有数据分析了R进行了统计计算框架v3.4或更高。
稀薄样品类群多样性(α多样性,平均超过100稀疏迭代)被计算为有效的类群希尔系数q = 0(即类群丰富),q = 1(香农熵指数)和q = 2(逆辛普森指数)和平衡措施比率。除非另有说明,结果在主文本参考类群丰富。α多样性的差异进行了测试使用方差分析(方差分析)其次是事后测试和Benjamini-Hochberg修正,作为指定的主要文本。
点之间的差异在社区组成(β多样性)被量化为Bray-Curtis不同原料或平方根改变计数、abundance-weighted Jaccard指数和abundance-weighted未加权的蒂娜指数,如前所述。74年这些指标之间的趋势通常是一致的,除非另有说明。结果报告Bray-Curtis非转换数据不同。关联的社区组成microbiome-external因素量化使用adonis2 PERMANOVA的实现和基于距离R冗余分析包素食v2.5。75年量化潜在的混杂单变量个别类群的丰度和科目的变量之间的联系(见正文),我们进行了方差分析或非参数克鲁斯卡尔-沃利斯测试,根据大量发行版(在线补充图2 - 3和在线补充表S4-S5)。从血液样本中胆红素水平测定,证实了地位和黄疸的临床记录。由于缺少黄疸地位好几个人,值用于进一步分析估算从现有的数据(图1,在线补充表S1-S3)。
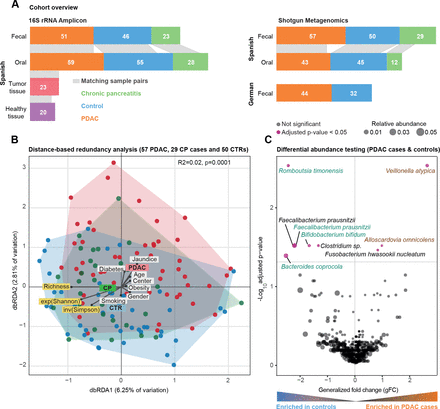
社区西班牙粪便微生物组数据的分析。(一)研究概述。灰色乐队之间的酒吧情节表明样本匹配的身体在个人网站。(B) Bray-Curtis基于距离冗余分析(dbRDA)胰腺导管腺癌(PDAC),慢性胰腺炎(CP)和控制(CTR)粪便微生物西班牙(ES)队列中的数据。PDAC样品显示为红颜色的圆圈,CP患者像蓝色绿色和控制。丰富,香农指数(exp (Shannon))和逆辛普森(发票(辛普森))多样性措施也想起箭头同样测试元数据变量。meta-variable从中心的距离代表了混杂效应值(见“方法”)。(C) Wilcoxon ES粪便微生物组的测试结果数据来测试PDAC之间丰富的类群和控制用例(见“方法”)。轴是log10(罗斯福纠正p值),x轴是普遍的褶皱变化,点的大小代表了一个特定物种的相对多度。红点代表显著差异在两组丰富的物种,罗斯福校正后虽然黑点显示非重要物种。 Green and brown-coloured species are selected in metagenomic model-1 as predictors of PDAC. FDR, false discovery rate.
多变量统计建模和模型评估
为了训练多变量统计模型对胰腺癌的预测,我们首先删除分类单元整体较低丰度和患病率(丰富分界点:0.001)。然后,功能被log10正常化转换(为了避免无限值对数,pseudo-count 1 e-05被添加到所有值)其次是标准化作为集中log-ratio (log.clr)。数据被随机分成测试和训练集的重复10倍交叉验证的10倍。为每个测试折叠,剩下的褶皱被用作训练数据训练一个L1-regularised(套索)逻辑回归模型76年使用中的实现LiblineaR v2.10 R包。77年训练模型被用来预测忽略时测试集最后,所有的预测都是用来计算接受者操作特征曲线下的面积(AUROC) (图2)。
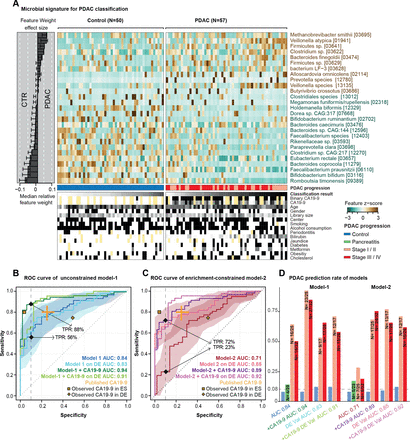
预测微生物签名的胰腺导管腺癌(PDAC)。(一)正常的粪便微生物的27个选定的物种丰富样品显示为一个热图。右面板代表每个选择的特征的贡献对整个模型1,和鲁棒性(的比例模型的功能是作为预测)的每个特性提出了比例。分类得分从每个个体的交叉验证和测试条件meta-variables显示在面板的底部,黄色代表信息缺失。(罪犯)内部交叉验证的结果无约束模式1(没有特征选择),enrichment-constrained model 2(受限的积极特性)和碳水化合物抗原(CA)波的组合(使用一个阈值37μL /毫升)和微生物特性(见“方法”)显示为接受者操作特征(ROC)曲线和95%可信区间阴影在相应的颜色。真阳性比率(tpr)给出90%的比例特异性截止。验证所有的模型在一个独立的德国人口(DE) PDAC测试(n = 76)表示。发表CA19-9精度从教育学橙色所示。黄点代表观察CA19-9精度在我们人群(数据可用于33/50控制(点击率数据)和44/57患者PDAC在西班牙(ES)和8/32点击率数据,44/44的患者PDAC在德国(DE)人口)(D) tpr的模型在不同PDAC进展阶段,此外,假阳性的慢性胰腺炎患者特异性和控制90%截止显示为酒吧的情节。阶段I和II和III和IV阶段结合由于总体样本容量低。 The number of predicted cases compared with the total is also shown on the top of each bar. DE-Val, German validation population.
在第二个方法中,特征是在交叉验证过滤(即每个训练集),首先计算单一特征AUROC然后删除功能AUROC < 0.5,从而选择功能丰富的PDAC (“enrichment-constrained”模式)。
为了把microbiome-based机器学习模型的预测与CA19-9标记,编码CA19-9标记(1为正,为0 -可用)被添加到的平均预测重复交叉验证运行,导致一个或组合。或者,和组合计算乘以CA19-9的预测标志。ROC曲线和AUROC值计算的组合使用pROC v1.15 R包。78年指定的95%可信区间是相应的颜色和阴影图传说中为每个ROC曲线。
ES宏基因组分类器训练的PDAC被应用于DE数据集应用数据正常化程序之后,选择相同的特性和使用相同的正常化参数集的特性(例如,意味着标准化使用冷冻正常化SIAMCAT功能)的正常化过程ES胰腺癌的数据集。分析,预测的分界点是将10%的假阳性率在最初的ES PDAC研究人口控制(图2)。
所有步骤的数据预处理(过滤和正常化)、模型训练、预测和模型评价进行使用SIAMCAT v.1.5.0 R包79年(https://siamcat.embl.de/)。
宏基因组分类器的外部验证
评估疾病特异性的训练模型,我们得到的预测样本其他肠道宏基因组数据集(在线补充表S6)的完整列表,包括加入数字)。我们进行了文献检索,确定公开的数据集的粪便在病例对照和队列研究基因组相关的疾病。总共25项研究,涵盖5792个样本在疾病状态,9个原始测序数据下载从欧洲核苷酸存档和分类学的异形如上所述。35-59
PDAC的宏基因组分类器训练后被应用于每个外部数据集应用数据正常化常规选择相同的特性和使用相同的正常化参数(例如,意思是标准化的功能通过使用冷冻功能正常化SIAMCAT)在胰腺癌的正常化过程的数据集。然后,预测疾病特异性评估由于其他疾病样本的样本预测高分表明生态失调的分类器依赖于一般特征与特定于胰腺癌的信号,这不会导致假阳性样本利率升高其他疾病。对于这个分析,预测的分界点是10%的假阳性率在最初的PDAC研究人口控制(图3)。的影响,年龄、性别和测序深度25人口预测分数cor.test函数进行了测试(斯皮尔曼方法)在车上v3.0-3 R包。
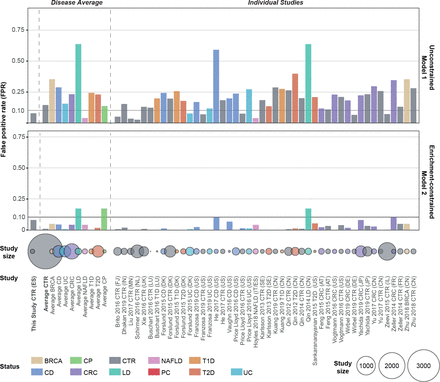
外部验证疾病特异性的胰腺导管腺癌(PDAC)粪便微生物模型。假阳性(玻璃钢)的宏基因组无约束模型1和enrichment-constrained model 2在25个外部测试集显示为一个酒吧策划(见在线补充表S4包括所有研究的列表)。验证数据集是异形和正常化以同样的方式作为初始数据集(参见“方法”)。每个研究是根据健康状况和分层模型测试来预测在给定组90%,特异性截止。低玻璃钢基因组从其他疾病患者和健康人表明模型是特定于PDAC。显示每组的受试者人数如下颜色编码的圈子。BRCA乳腺癌;CRC,结直肠癌;CD,克罗恩病;CTR, CP,慢性胰腺炎;控制;LD、肝脏疾病; NAFLD, non-alcoholic fatty liver disease; PC, pancreatic cancer; T1D, type 1 diabetes; T2D, type 2 diabetes; UC, ulcerative colitis; ES, Spanish; DE, German.
亚种和strain-level分析
宏基因组读取从proGenomes v1映射species-representative基因组数据库80年(见上图)。从独特的微生物单核苷酸变异被称为映射使用metaSNV读取,81年和物种内部等位基因样本之间的距离计算如前所述。82年等位基因之间的关联距离和PDAC疾病状态量化使用PERMANOVA分层潜在的混杂因素后(包括取样机构网站)。
Oral-intestinal菌株的传播是量化如前所述。83年简而言之,微生物之间的重叠单核苷酸变异在唾液和粪便样本对象是与主客体之间的背景来计算定量oral-faecal传输得分和p值。关联的物种和科目的分数与传播进行了测试使用方差分析和临床因素事后测试,其次是Benjamini-Hochberg修正为多个测试。
荧光原位杂交显微镜
鱼分析使用探测器专门针对16 s rRNA序列的特定的细菌分类单元(图4)。所有探测器都选择基于文献检索和显示在相应的分类单元在线补充表S7)。
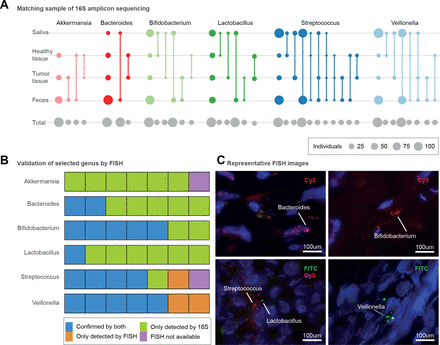
微生物的存在与不同条件下的不同部分胰腺。(A)存在不同的属在四个不同的身体网站包括粪便、唾液、胰腺肿瘤组织和健康组织样本,所推断的16 s扩增子数据。圆的大小对应于每个可用的主题比较总数(灰色,底部行)或与自身内在匹配的扩增子序列变异(彩色);匹配样本由线连接类型。第一列显示样品的总数/网站发现的属。(B)七个选择胰腺组织样本(5个肿瘤和两个non-tumour)来显示细菌存在/缺席16 s扩增子和荧光原位杂交(鱼)方法。验证的细菌存在16 s扩增子测序和鱼是蓝色所示。样品显示细菌存在根据16 s只有用绿色显示。细菌存在验证只有鱼是橙色,和样品不受鱼验证由于缺乏组织材料所示紫色。(C)代表显微镜图像拟杆菌(在细胞核内的肿瘤组织),双歧杆菌属(核外,肿瘤组织),乳酸菌(核外,non-tumour组织),链球菌(核外,non-tumour组织),韦永氏球菌属(核外,肿瘤组织)。异硫氰酸荧光素(FITC)和Cy3荧光染料被用作表示,和DAPI (4, 6, -diamidino-2-phenylindole;蓝色)用于标签细胞核。
胰腺肿瘤和正常胰腺样本获得病理部门并立即冻结在液态氮在不到30分钟的手术切除。无菌材料被用来分析不同样品。组织冻结的最小尺寸是大约0.125厘米3(0.5×0.5×0.5厘米)。样本从临时液态氮转移运输集装箱和保存在一个锁着的冰箱在-80°C。之前分析他们在干冰运输,搬到一个最佳的切削温度模具在液态氮,并立即削减cryotome获得10个小组,每组3 - 5µm。所有材料消毒后用乙醇每个样品处理。
组织部分5µm厚度是安装在带正电的幻灯片(SuperFrost,热科学)。简单地说,组织在刚做好的4%多聚甲醛后缀。增强细菌壁permeabilisation的溶菌酶治疗后(10 g / L盐酸三6.5 m),样本杂交1小时45°C的具体调查hybridiser机(DAKO)。杂交是在20µL杂交缓冲(20 nM三,pH值8.0。0.9 M氯化钠,0.02%十二烷基硫酸钠、30%甲酰胺)添加到100 ng的调查。最后,组织在洗涤溶液洗(70%的甲酰胺,10毫米三羟甲基氨基甲烷pH7.2液和01%牛血清白蛋白),乙醇脱水的一系列样品,风干和0.5µg /毫升DAPI染色(4,6,-diamidino-2-phenylindole) /不变色的解决方案(Palex医学)。鱼捕获图像使用徕卡DM5500B显微镜和CCD相机(光度SenSys)连接到PC运行CytoVision软件7.2图像分析系统(应用成像)。图像分析了盲人和得分基于探测信号的强度。
结果
PDAC与温和的微生物组成的变化时控制混杂因素在猎枪宏基因组数据
我们研究了57个新诊断,首次治疗患者PDAC, 29日慢性胰腺炎(CP),患者和50控制与年龄、性别和医院。前瞻性地招募参与者来自两家医院在巴塞罗那和马德里,西班牙,2016年至2018年,使用相同的标准(见主题特点图1一个和在线补充表S1-S3每个主题)的临床资料。我们获得粪便猎枪基因组所有科目和唾液基因组45 PDAC患者,12与CP, 43控制(参见“方法”)。分析工作流详细在线补充图1。
一些PDAC危险因素,如吸烟、饮酒、肥胖或糖尿病,本身与微生物组成有关84年,我们首先试图建立微生物签名的潜在混杂因素在我们的研究人群中,为了调整相应的分析。总共26个人口和临床变量,我们量化的边际影响微生物社区的多样性(在线补充表S4)。粪便和唾液微生物丰富α多样性(作为代理)没有与任何测试变量相关的单变量,或者PDAC状态,当占最常见的PDAC风险因素和应用的错误发现率阈值0.05 (在线补充图2,在线补充表S4)。
相比之下,微生物群落组成随年龄诊断(PERMANOVA上点之间Bray-Curtis相异,R2 = 0.01, Benjamini-Hochberg-corrected p = 0.03),糖尿病(R2 = 0.01, p = 0.04)和黄疸状态(R2 = 0.02, p = 0.009)的粪便和阿司匹林/对乙酰氨基酚使用(R2 = 0.02, p = 0.04),唾液,尽管以非常低的效应大小(在线补充表S5)。尽管病例和对照组为年龄和性别匹配的,我们将这些因素作为地层为后续分析。在这样的调整,疾病状态,态度温和,但统计上显著相关的社区组成的粪便(R2 = 0.02, p = 0.001),但不是在唾液(R2 = 0.01, p = 0.5) (图1 b,在线补充图3 - 4,在线补充表S5)。事实上,患者的粪便微生物组成PDAC既不同于控制(R2 = 0.02, p≤0.0001)和CP患者(R2 = 0.02, p = 0.003),虽然同样在非常小的尺度效应。
高精度宏基因组分类器捕获特定PDAC患者粪便微生物签名
建立了肠道微生物的存在信号粗级别的PDAC整体社区的成分,我们明年确定九物种特异单变量关联(Wilcoxon测试的相对丰度在PDAC情况下对控制,Benjamini-Hochberg-corrected p < 0.05;看到图1 c)。最显著,韦永氏球菌属atypica,梭菌属nucleatum / hwasookii和Alloscardovia omnicolens丰富的PDAC患者的粪便,而Romboutsia timonensis, Faecalibacterium prausnitzii,拟杆菌coprocola和双歧杆菌bifidum物种群被耗尽。相比之下,我们没有发现任何物种显著差异时唾液微生物丰度修正为多个测试,包括之前报道协会等Porphyromonas gingivalis, Aggregatibacter actinomycetemcomitans,22奈瑟氏菌属elongata或链球菌18(在线补充图5)。
单变量相关的粪便中物种,几个被自己适度预测PDAC状态(在线补充图5)。合并这些单个信号到一个包罗万象的模型,我们接下来建立multispecies宏基因组装配套索逻辑回归模型分类器的10倍交叉验证(见“方法”)。当应用没有进一步约束,获得模型之间的歧视患者PDAC和高精度控制在我们的研究人群(“模式1”;AUROC = 0.84;图2)。在模型中最突出的积极标志物种Methanobrevibacter smithii,Alloscardovia omnicolens,韦永氏球菌属atypica和拟杆菌finegoldii。我们注意到,通过设计,套索回归选择代表inter-correlated集之间的特性;因此,这些物种可能是更大的代表物种集丰度与高度相关。所有的26个人口统计学和流行病学变量描述我们的研究人群被选为模型的预测功能,和微生物签名(见更多的信息比其他任何功能在线补充图6和7)。进一步,这些变量分别与微生物相关的物种代表模型中,裁决作为潜在的混杂因素。这表明该分类器捕获一个诊断肠道微生物组签名PDAC可能是独立于其他疾病的危险因素和潜在的混杂因素。
一个类似的模型构建CP患者与控制没有预测能力(AUROC = 0.5;在线补充图8),与观测一致,这些组构图基本无异。同样,没有健壮的PDAC签名检测唾液微生物(AUROC = 0.48;在线补充图9)。然而,粪便模型区分从那些PDAC CP患者表现更好的AUC 0.75,但是模型鲁棒性是有限的样本量低与CP组(在线补充图8)。我们进一步探讨高分辨率的预测关联功能微生物组的概要文件。模型基于KEGG模块的丰度(在线补充图10)实现了精度AUROC = 0.74,但特征选择同样没有健壮的验证折叠,因此拟合高数量的变量(模块)与一组有限的样本。因此我们追求的物种分类器,因为它们提供了稳定的模型。
最初的肠道microbiome-based分类器包括几个物种枯竭PDAC相对于控件,如Faecalibacterium prausnitzii,拟杆菌coprocola,双歧杆菌bifidum或Romboutsia timonensis(图2 b)。对于这些物种,它曾建议损耗与肠道炎症,一般来说,而不是特定的疾病。85年因此我们重新训练分类器的约束(丰富)呈正相关微生物的特征是专门选择在每个交叉验证。结果enrichment-constrained模型(model 2)看见患者PDAC AUROC精度= 0.71。区别与无约束模型,模型1,主要是由于对灵敏度是一个点球,自信减少检测患者的PDAC,当训练对稀疏数据符合预期。
宏基因组分类器组合抗原CA19-9水平增加准确性
抗原的血清水平CA19-9通常用于监控PDAC进步,86 87但同时也被认为是一个潜在的PDAC早期诊断标记,尽管有温和的灵敏度(0.80,95%可信区间0.72到0.86)和特异性(0.75,95%可信区间0.68到0.80)。12CA19-9血清水平可供77人的一个子集(33/50控制和44/57 PDAC)患者在我们的西班牙人口(在线补充图S11)。鉴于CA19-9直接分泌肿瘤,我们假设,提供的读数CA19-9血清水平和我们的微生物分类器是互补的,和他们的组合可以提高PDAC预测的准确性。事实上,占CA19-9增加我们的无约束模型1的准确性AUROC = 0.84 - 0.94,主要由敏感性的增加(图2 b)。更引人注目的是,当我们修改enrichment-constrained model 2 CA19-9信息,我们观察到大量增加的准确性AUC = 0.71 - 0.89,同样由灵敏度显著提高,从而基本上废除了性能损失相对于模式1 (图2 c,在线补充图S11)。没有明显偏向于更高的CA19-9水平在以后的疾病阶段ES或数量(在线补充图S11)。
我们的西班牙研究人群包括25 PDAC患者在疾病早期阶段(T1, T2)和32个科目在后期(T3、T4)。疾病阶段并不影响性能的microbiome-based模型(图2 d);特别是,回忆不是偏向后期。
metagenome-based分类器对独立验证一组的性能
测试是否观察到微生物签名概括超出了我们的焦点西班牙研究人群,我们下一个挑战在两个验证场景模型。首先,我们测试了预测精度在一个独立研究人口44 PDAC患者和32匹配控制,招募了来自两家医院在埃朗根和法兰克福,德国(见图1、方法和在线补充表S3),正在处理的样品相同的西班牙人。在这个人口DE验证,无约束模型1 (图2 b)和enrichment-constrained model 2 (图2 c)执行类似的甚至优越的精度训练人口,都有和没有CA19-9互补的水平,和类似的趋势在疾病阶段(图2 d)。
接下来,我们宏基因组分类器捕获PDAC-specific签名确认,而不是不具体的,更一般的疾病有关的变化,我们进一步验证他们对独立,外部宏基因组数据集不同的健康状况。总,我们分类5792公开肠道基因组从25在18个国家的研究,包括主题与CP(本研究),1型或2型糖尿病、结肠癌、乳腺癌、肝脏疾病、非酒精脂肪肝疾病,包括克罗恩病和溃疡性结肠炎,以及健康对照组(图3和在线补充表S6)。
当调到90%的特异性(允许10%的假阳性预测)在我们的焦点ES研究人口,56%的患者的无约束模型1显示召回的PDAC ES人口和48% DE验证人口假阳性率(6%),和64%的补充信息时CA19-9水平(用于控制8/32和43/44的患者病例队列)。模式1的疾病特异性,然而,是有限的,预测的PDAC国家平均15%的对照组所有外部数据集。大多数这些假阳性的调用被观察到在两个中国人口克罗恩病的患者48或肝硬化。44节段性回肠炎与损耗有关签名所观察到的类似的在我们的模型中(尤其是f . prausnitzii88年),而肝脏疾病和胰腺功能受损分享一些生理特征。然而,所有其他肝脏疾病和克罗恩病集显示错误检出率较低,表明该效应可能是由于,在某种程度上,技术和人口之间的影响研究。的确,我们注意到,在这两个中国人口研究的受试者明显比我们年轻人群(50 Qin_2014±11年;He_2017 28.5±8年;70±12 y耳朵ES人口)。这个年龄的作用是系统:所有验证集,PDAC预测分数与主题相关的年龄(方差分析p = 0.007;ρ斯皮尔曼= 0.16),以及性的主题(p < 106;)和测序深度(p = 0.0008;ρ斯皮尔曼= 0.1)(在线补充图S12,在线补充表S6)。
的enrichment-constrained model 2显示低检出率PDAC患者在这两个群体,虽然召回是恢复CA19-9组合模型。model 2非常具体的PDAC,平均0 - 5% PDAC预测在几乎所有外部人口,17%的最大预言中提到的44人口与肝脏疾病。特别是,发现微生物签名也健壮的错误分类的2型糖尿病患者(< 2%的假阳性率);这是潜在的筛选应用相关的,因为这些病人是一个主要的PDAC风险集团(图3)。
细菌PDAC港口特点,符合口腔和肠道微生物群落
改变胰腺分泌,由于肿瘤生长在胰管,会影响消化功能,因此合理的构成特点肠道微生物组签名,如上面所描述的。这将意味着PDAC进展可以间接导致微生物变化(即反向因果关系)。此外,胰管直接与十二指肠,为细菌提供一个解剖的链接25 30 89和真菌34移植胰腺和致癌作用。31日
我们因此提出几种肠道微生物类群与PDAC应该检测胰腺肿瘤。我们描绘分类学的粪便和唾液样本,以及活检肿瘤(n = 23)和邻近健康胰腺组织(n = 20)患者的PDAC从我们的研究人群使用16 s rRNA扩增子测序,应用严格过滤排除假定的试剂污染物通常出现在样品细菌生物量较低33 90(见“方法”)。我们观察到一个令人惊讶的丰富多样的胰腺微生物,至少有13个细菌属在≥25%的样本,主要包括与特征PDAC签名粪便微生物类群91年(图4一个,在线补充图13)。在这其中,乳酸菌spp,Akkermansia muciniphila和拟杆菌spp富集在肿瘤相对于non-tumour胰腺组织(Wilcoxon测试,错误发现rate-corrected p < 0.006)。
的一个子集五肿瘤和两个non-tumoral胰腺组织样本,我们可以进一步验证的患病率Akkermansiaspp,乳酸菌spp,双歧杆菌属仕达屋优先计划、韦永氏球菌属仕达屋优先计划,拟杆菌spp和链球菌spp用鱼化验genus-specific引物(在线补充图4,在线补充表S7)。一般来说,扩增子和鱼数据整合,尽管amplicon-based检测可能出现更敏感由于组织的数量分析。有趣的是,然而,Akkermansiaspp,虽然观察到扩增子测序在26/30,没有检测到使用鱼在任何被测试的样品(图4 c,在线补充图14)。
口腔、肠道和胰腺微生物之间的联系
我们下一个追踪的扩增子序列变异(asv)唾液,粪便,肿瘤组织和健康组织样本在对象(图4一),最高分类使用16 s rRNA数据分辨率可以达到。韦永氏球菌属spp,典型丰富PDAC患者粪便,非常流行唾液(100%的受试者)和粪便样本(87.5%)在整个研究人口,而口语和粪便类型也与肿瘤和asv non-tumour组织。有趣的是,我们没有发现个体内的比赛韦永氏球菌属asv肿瘤与邻近组织样本,表明tumor-dwelling韦永氏球菌属spp可能不同于那些在健康的组织。此外,我们的数据证实先前的报道乳酸菌仕达屋优先计划26和双歧杆菌属仕达屋优先计划25存在PDAC肿瘤和non-tumour组织。两个属,我们发现肿瘤类型与口服或粪便asv,但不是两个,而没有从健康组织asv与粪便样本,表明不同的胰腺亚种群可能与口腔和肠道。
使用成对的唾液和粪便猎枪基因组,我们进一步证实,粪便PDAC-associated微生物菌株可能来自口腔(在线补充结果)。
讨论
早期发现PDAC仍然是一个艰巨的挑战,持续努力的核心来减轻这种癌症的负担。目前,fda唯一批准的生物标志物PDAC血清CA19-9,主要用于疾病监测而不是筛选,由于固有的敏感性和特异性的局限性:CA19-9水平可以提升几个条件与胰腺癌无关,而学科缺乏刘易斯抗原不产生CA19-9。10 - 12提出了小规模研究PDAC标记根据胰腺组织,5尿液6 7和血清8 9有限的适用性。然而目前没有筛选工具的PDAC诊所,为疾病的早期阶段。
前瞻性招募新诊断的研究中,首次治疗病人和匹配的对照组口服的,粪便和组织微生物进行分析(图1一个),我们开发了宏基因组分类器,强劲而准确地预测PDAC仅仅基于特征粪便微生物物种(图2)。PDAC签名被我们multispecies模型正交的PDAC危险因素(图1 b和2)。这表明,在实践中,粪便微生物可用于屏幕PDAC,补充其他测试的标记,添加了诊断准确性测试相结合,作为结直肠癌提出了。39事实上,我们与CA19-9微生物分类器的组合数据,可供我们的人口的一个子集,显著增强PDAC检测的准确性(图2罪犯)。
先前的研究已经探讨PDAC和口语之间的联系在18到22岁的26日92 93或粪便23日24微生物的分类16 s rRNA序列的分辨率有限,但提供了相互矛盾的报道关于个别类群的关联模式,可能由于异构的实验和分析方法。原始序列和患者的立场临床数据的某几个PDAC数据集做了对比研究的挑战,从而达成共识PDAC-associated微生物签名迄今未能出现。几个之前报道口服类群包括单变量PDAC关联p . gingivalis actinomycetemcomitans,美国酸奶和梭菌属spp没有确认在我们的研究人群(在线补充图4);我们一般没有观察到任何唾液PDAC签名对个别物种或multispecies模型。
我们仔细检查分析人口、生活方式、和临床混杂因素,因为这些可以显示比疾病更强的微生物协会。84年我们此外验证宏基因组对独立样本分类器,然而不断加工,人口(图2罪犯)和外部各种健康状态的数量从25个不同的研究(n = 5792)35-59(图3)。“控制和外部验证是必不可少的在评估预测模型的疾病特异性,特别是PDAC等疾病的发病率较低。这是我们分析证实:在我们两个宏基因组分类,模式1显示高精度AUROC = 0.84的ES研究人口,由高召回PDAC患者。然而,模式1显示有限疾病特异性外部验证,获取非特异性物种枯竭信号之间的区别的情况下,控制人口,而且共享的对象与其他疾病。其中包括通用的炎症签名例子,损耗f . prausnitzii大肠rectale或b . bifidum。发表的宏基因组分类器对各种疾病,特别是之前报道PDAC签名,分享相似的局限性:焦人口高度调整精度,但非特异性功能共享与其他疾病。这种缺乏特异性限制了他们翻译成临床实践。相比之下,我们的model 2,限制PDAC-enriched特性,实现只有温和的准确性在我们人群(AUC ES = 0.71, AUC = 0.85 DE)由于惩罚敏感性,但非常低的高度PDAC-specific虚假预测利率在外部种群,包括已知的PDAC与2型糖尿病组等风险。特别是PDAC-enriched特性模型1和模型2显示小重叠特征粪便微生物特性对于其他癌症,如结肠癌,表明我们的微生物的组合模型与CA19-9水平(高度敏感,但不是特定于PDAC)是光明的。我们注意到外部人口剩余假阳性率可能部分是由于技术异质性,在所有外部人口抽样和加工使用独立的协议,这一元PDAC协会个别物种可能是有用的,但不是特定疾病(补充讨论)。PDAC-enriched物种在model 2的面板显示潜力microbiome-based PDAC筛查,鉴于对血清结合互补信息CA19-9显著增加准确性(AUC = 0.89和0.92)。
我们的模型显示类似的性能在PDAC疾病阶段,没有偏向后期(图2罪犯)。这表明微生物特征签名出现在疾病的进展和早期的粪便微生物可以PDAC的早期检测。
我们的数据是严格观察和横断面。然而,有很强的迹象表明,所确定的粪便微生物变化不仅仅是胰腺功能受损或系统性影响的结果,虽然不能排除间接影响。可以跟踪一些类群在肠道和胰腺之间,与单变量浓缩在肿瘤相对于邻近的健康组织,表明直接关联的PDAC肠道微生物组。我们证实了以前的观测25 30 31 89 91人类胰腺港口一个微生物,通过扩增子测序,鱼迄今最全面的分类单元(图4)。胰腺组织和肿瘤只包含细菌生物量低,因此容易污染16 s rRNA扩增子数据33,而鱼测试需要特定的假设,所以健康的全面编目和病变胰腺微生物成分仍然是新兴。在我们的研究中,我们仔细过滤数据集对已知工具包污染物和证实存在的各种关键属使用鱼化验。我们此外观察到精确的扩增子序列变异的个体内的重叠之间的口交,粪便和组织样本,确认共享存在跨多个网站几个物种在最高可以达到分类为扩增子数据分辨率。
粪便的数量特征PDAC-associated类群因此可以追溯到胰腺肿瘤。同样,我们观察到显著增加oral-intestinal应变水平传输PDAC患者,特别是PDAC签名的类群,表明这些可能是个体内的采购,从口腔(在线补充结果)。这些发现表明,口腔、肠道和胰腺微生物可能是复杂的联系,和多体网站研究设计,比如这里提出有必要解决各自的角色和交互PDAC病因学。
总之,描述粪便微生物签名启用健壮的宏基因组的PDAC检测分类器疾病特异性高,补充现有的标记,和与潜在的具有成本效益的PDAC筛查和监测。此外,鉴于先前的报道在小鼠模型和人类microbe-mediated胰腺癌生成,25 30 94我们相信的介绍面板PDAC-associated细菌物种可能相关的超出了他们用于诊断,提供承诺未来的入口点疾病预防和治疗干预。
数据可用性声明
数据在公共、开放访问存储库。所有数据都包含在相关研究文章或作为补充信息上传。样品的原始测序数据可用在欧洲核苷酸存档(ENA)标识符PRJEB38625和PRJEB42013研究。这些样本作为补充提供的元数据表S1和S2。过滤分类和功能配置文件作为输入用于统计模拟管道在S1和S2补充数据是可用的。分析代码和结果可用https://github.com/psecekartal/PDAC.git。
伦理语句
病人同意出版
伦理批准
参与者预期从医院招募·拉蒙-卡哈尔在马德里和医院Vall dHebron在巴塞罗那,西班牙。机构审查委员会伦理批准(CEIπ26 2015 - v7)和书面知情同意是来自参与中心和研究参与者,分别。一个独立的验证人口招募的手术,埃朗根大学医院(32 PDAC和32个控制样本)和部分平移肝病,内科,法兰克福歌德大学诊所(12 PDAC样本)。这项研究是经当地伦理委员会批准(sgi - 3 - 2019, 451 _18 B),临床数据,包括疾病阶段和后续的数据,从医院的临床记录图表检索各自的病人。
确认
我们感谢博克的成员,Malats和西团体激励讨论和所有的贡献。此外,我们感谢EMBL的基因组测序的核心设施的支持。
引用
脚注
纳米和PB联合高级作者。
推特@ps_ecekartal、@TSBSchm @o__maistrenko、@ZellerGroup @JonelTrebicka, @nmalats @BorkLab
艾克,tsb和EM-M同样起到了推波助澜的作用。
合作者PanGenEU研究调查。西班牙国家癌症研究中心(CNIO),马德里,西班牙:Nuria Malats弗朗西斯科X真实,Evangelina洛佩兹de Maturana Paulina Gomez-Rubio以斯帖Molina-Montes,洛拉阿隆索,Mirari马尔克斯是罗杰·米尔恩安娜居多,塔尼亚Lobato,莉迪亚Estudillo。意大利维罗纳大学:丽塔Lawlor阿尔多斯卡帕,普Beghelli。国家癌症登记处爱尔兰、软木、爱尔兰:琳达,达米安奥德利。医院Madrid-Norte-Sanchinarro,马德里,西班牙:耶稣Rodriguez帕斯卡•曼努埃尔•伊达尔戈。医院·拉蒙-卡哈尔,马德里,西班牙:阿尔弗雷多Carrato, Alejandra Caminoa,卡门Guillen-Ponce,梅赛德斯Rodriguez-Garrote,费德里科•Longo-Munoz,雷耶斯Ferreiro,凡妮莎Pachon, M洛杉矶Vaz。医院德尔,巴塞罗那,西班牙:马尔·伊格莱西亚斯,卢卡斯Ilzarbe克里斯蒂娜Alvarez-Urturi,泽维尔Bessa,费利佩•Bory卢西亚马尔克斯是Ignasi pof,费尔南多•Burdio路易斯•格兰德希梅诺哈维尔。西班牙巴塞罗那医院Vall dHebron: Xavier Molero,路易莎Guarner, Joaquin Balcells Mayte Salcedo。德国慕尼黑工业大学:Christoph Michalski艾琳•埃斯波西托Jorg Kleeff Bo。瑞典,斯德哥尔摩卡罗林斯卡研究所:马提亚Lohr,黄Jiaqui卡罗琳韦贝克(音译),余论。 Hospital 12 de Octubre, Madrid, Spain: José Perea, Pablo Peláez. Hospital de la Santa Creu i Sant Pau, Barcelona, Spain: Antoni Farré, Josefina Mora, Marta Martín, Vicenç Artigas, Carlos Guarner, Francesc J Sancho, Mar Concepción, Teresa Ramón y Cajal. The Royal Liverpool University Hospital, UK: William Greenhalf, Eithne Costello. Queen’s University Belfast, UK: Michael O’Rorke, Liam Murray, Marie Cantwell. Laboratorio de Genética Molecular, Hospital General Universitario de Elche, Spain: Víctor M Barberá, Javier Gallego. Instituto Universitario de Oncología del Principado de Asturias, Oviedo, Spain: Adonina Tardón, Luis Barneo. Hospital Clínico Universitario de Santiago de Compostela, Spain: Enrique Domínguez Muñoz, Antonio Lozano, Maria Luaces. Hospital Clínico Universitario de Salamanca, Spain: Luís Muñoz-Bellvís, J.M. Sayagués Manzano, M.L. Gutíerrrez Troncoso, A. Orfao de Matos. University of Marburg, Department of Gastroenterology, Phillips University of Marburg, Germany: Thomas Gress, Malte Buchholz, Albrecht Neesse. Queen Mary University of London, UK: Tatjana Crnogorac-Jurcevic, Hemant M Kocher, Satyajit Bhattacharya, Ajit T Abraham, Darren Ennis, Thomas Dowe, Tomasz Radon. Scientific advisors of the PanGenEU Study: Debra T Silverman (NCI, USA) and Douglas Easton (U. of Cambridge, UK).
魔法(MicrobiotA-focused德国跨学科合作)的研究人员。部分平移肝病,内科,法兰克福癌症研究所,法兰克福歌德大学:Jonel Trebicka,汉斯伊拉斯谟,费边Finkelmeier,罗伯特•Schierwagen Wenyi顾,奥拉夫yyc,弗兰克·Uschner, Stefan Zeuzem。格赖夫斯瓦尔德大学外科学系:斯蒂芬Kersting,梅勒妮Langheinrich。埃尔兰根大学外科学系:Georg f·韦伯,罗伯特•Grutzmann基督教Pilarsky。埃尔兰根大学内科:沃茨斯蒂芬。
贡献者埃克设计的研究,开展实验工作,获取和分析数据,写了第一个手稿起草和修订后的手稿。tsb设计研究,获取和分析数据,写了第一个手稿起草和修订后的手稿。新兴市场-M designed the study, contributed to patient recruitment and the collection of biomaterials and clinical data, acquired and analysed data, and wrote the first manuscript draft.SR-P contributed to patient recruitment and the collection of biomaterials and clinical data and conducted experimental work.JW, OMM, WAA, BAA, AC, HP-E, FF, PG-R, SKe, ML, MM, XM, RT-R, JT contributed to patient recruitment and the collection of biomaterials and clinical data. RJA, AF, AMG, KZ contributed to data analysis. LE contributed to patient recruitment and the collection of biomaterials and clinical data and conducted experimental work. RH, FJ, SKa, AT conducted experimental work and acquired data. AO, TvR contributed to data analysis. MSI, PSI contributed to patient recruitment. VB acquired data. GZ designed the study and contributed to data analysis. FXR designed the study and contributed to data analysis and wrote the first manuscript draft. NM conceived the study, designed the study, contributed to patient recruitment and the collection of biomaterials and clinical data and wrote the first manuscript draft. PB conceived of the study, designed the study, contributed to data analysis and wrote the first manuscript draft. All authors reviewed, edited and approved the final version of the manuscript.
资金我们承认资助EMBL CNIO,世界癌症研究(# 15 - 0391),欧洲研究委员会(erc - adg - 669830 MicrobioS),人类生物信息学的海德堡BMBF-funded CenterCentre (HD-HuB)在德国生物信息学网络基础设施(de.NBI # 031 a537b),洋底de Investigaciones疗养地(FIS), Instituto de Salud卡洛斯III-FEDER西班牙(格兰特数字PI15/01573、PI18/01347 FIS PI17/02303);红色Tematica de Investigacion Cooperativa在癌症、西班牙(格兰特数字RD12/0036/0034, RD12/0036/0050 RD12/0036/0073);三世贝科卡门Delgado / Miguel de AESPANC-ACANPAN Perez-Mateo;EU-6FP集成项目(# 018771 - moldiag天竺鼠);EU-FP7-HEALTH (# 259737 - canceralia)。投资者没有参与研究设计,病人登记,分析,手稿撰写或审核。
相互竞争的利益EM-M tsb EK, JW,石,广州,LE, SR-P, FXR、纳米和PB未决的专利申请(申请号:EP21382876.7)基于微生物生物标记胰腺癌的早期检测。其他作者声明没有利益冲突。
出处和同行评议不是委托;外部同行评议。
补充材料此内容已由作者(年代)。尚未审查由BMJ出版集团有限公司(BMJ)和可能没有被同行评议。任何意见或建议讨论仅代表作者(年代)和不了BMJ的支持。和责任起源于BMJ概不负责任何依赖的内容。内容包括任何翻译材料,BMJ并不保证翻译的准确性和可靠性(包括但不限于当地法规、临床指南,术语,药物名称和药物剂量),和不负责任何错误或遗漏引起的翻译和改编或否则。